Этот пример показывает, как создать пользовательскую функцию и включить ее в симуляцию модели.
Этот пример принимает, что у вас есть практическое знание:
Рабочий стол MATLAB®
Создание и сохранение программ MATLAB
Этот пример использует модель, описанную в Модели Дрожжей Гетеротримерный Цикл Белка G.
Эта таблица показывает, что реакции раньше моделировали цикл белка G и соответствующие параметры уровня (константы скорости) для каждой реакции. Для обратимых реакций параметр форвардного курса перечислен сначала.
| Нет. | Имя | Reaction1 | Параметры уровня |
|---|---|---|---|
| 1 | Взаимодействие лиганда приемника | L + R <-> RL | kRL, kRLm |
| 2 | Гетеротримерное формирование белка G | Gd + Gbg -> G | kG1 |
| 3 | G активация белка | RL + G -> Ga + Gbg + RL | kGa |
| 4 | Синтез приемника и ухудшение | R <-> null | kRdo, kRs |
| 5 | Ухудшение лиганда приемника | RL -> null | kRD1 |
| 6 | G деактивация белка | Ga -> Gd | kGd |
| 1 Легенда разновидностей: L = лиганд (альфа-фактор), R = факторный альфой приемник, Gd = неактивный G-alpha-GDP, Gbg = свободные уровни комплекса G-beta:G-gamma, G = неактивный комплекс Gbg:Gd, Ga = активный G-alpha-GTP | |||
Этот пример принимает что:
Ингибитор (разновидности Inhib) замедляет деактивацию активного белка G (реакция 6 выше, Ga –> Gd).
Изменение в сумме ингибитора (разновидности Inhib) задано в пользовательской функции, inhibvalex.
Ингибитор (разновидности Inhib) влияет на реакцию путем изменения суммы параметра уровня kGd.
Этот пример показывает, как создать и вызвать пользовательскую функцию в выражении SimBiology®. А именно, это показывает, как использовать пользовательскую функцию в выражении правила.
Можно использовать пользовательские функции в:
Выражения скорости реакции (свойство ReactionRate)
Управляйте выражениями (свойство Rule)
Выражения события (свойство EventFcns или свойство Trigger)
Требования для использования пользовательских функций в выражениях SimBiology:
Создайте пользовательскую функцию. Для получения дополнительной информации смотрите function.
Измените текущую папку на папку, содержащую ваш пользовательский файл MATLAB. Сделайте это при помощи команды cd или при помощи поля Current Folder на панели инструментов рабочего стола MATLAB. Также добавьте папку, содержащую ваш файл к пути поиска файлов. Сделайте это при помощи команды addpath или смотрите Папки Изменения на Пути поиска файлов (MATLAB).
Вызовите пользовательскую функцию в реакции SimBiology, правиле или выражении события.
Если ваше выражение правила или скорости реакции не непрерывно и дифференцируемо, смотрите Используя События, чтобы Обратиться к Разрывам в Выражениях Правила и Скорости реакции прежде, чем моделировать вашу модель.
Следующая процедура создает пользовательскую функцию, inhibvalex, который позволяет вам задать, как сумма ингибитора изменяется в зависимости от времени. Входные параметры время, начальная сумма ингибитора и параметр, который управляет суммой ингибитора. Вывод функции является суммой ингибитора.
В рабочем столе MATLAB выберите File > New > Script, чтобы открыть редактора MATLAB.
Скопируйте и вставьте следующее объявление функции:
% inhibvalex.m function Cp = inhibvalex(t, Cpo, kel) % This function takes the input arguments t, Cpo, and kel % and returns the value of the inhibitor Cp. % You can later specify the input arguments in a % SimBiology rule expression. % For example in the rule expression, specify: % t as time (a keyword recognized as simulation time), % Cpo as a parameter that represents the initial amount of inhibitor, % and kel as a parameter that governs the amount of inhibitor. if t < 400 Cp = Cpo*exp(-kel*(t)); else Cp = Cpo*exp(-kel*(t-400)); end
Сохраните файл (назовите файл inhibvalex.m) в директории, которая находится на пути поиска файлов MATLAB, или к директории, к которой можно получить доступ.
Если местоположение файла не находится на пути поиска файлов MATLAB, измените рабочую директорию на расположение файла.
Загрузите проект gprotein в качестве примера, который включает переменную m1, объект модели:
sbioloadproject gproteinОбъект модели m1 появляется в рабочем пространстве MATLAB.
Следующая процедура создает выражение правила, которое вызывает пользовательскую функцию, inhibvalex, и задает эти три входных значения к этой функции.
Добавьте повторное правило присвоения в модель, которая задает эти три входных значения к пользовательской функции, inhibvalex:
rule1 = addrule(m1, 'Inhib = inhibvalex(time, Cpo, Kel)',... 'repeatedAssignment');
Вход time является ключевым словом SimBiology, распознанным временем симуляции
Создайте эти два параметра, используемые правилом rule1, и присвойте значения им:
p1 = addparameter(m1, 'Cpo', 250); p2 = addparameter(m1, 'Kel', 0.01);
Создайте разновидности, используемые правилом rule1:
s1 = addspecies(m1.Compartments, 'Inhib');Значение параметра уровня kGd затронуто суммой ингибитора, существующего в системе. Добавьте правило в модель, чтобы описать это действие, но сначала изменить свойство ConstantValue параметра kGd так, чтобы это могло отличаться правилом.
Измените свойство ConstantValue параметра kGd к false.
p3 = sbioselect(m1, 'Type', 'parameter', 'Name', 'kGd'); p3.ConstantValue = false;
Добавьте повторное правило присвоения в модель, чтобы задать, как параметр kGd затронут разновидностями Inhib.
rule2 = addrule(m1, 'kGd = 1/Inhib', 'repeatedAssignment');
Пользовательская функция, inhibvalex, вводит разрыв в модели когда время = 400. Чтобы гарантировать точные результаты симуляции, добавьте событие в модель, чтобы сбросить решатель во время разрыва. Установите событие инициировать во время разрыва (время = 400). Событие не должно изменить модель, поэтому создать функцию события, которая умножает значение разновидностей на 1.
addevent(m1, 'time>=400', 'G=1*G');
Сконфигурируйте настройки симуляции (configset object) для объекта модели m1, чтобы регистрировать все состояния во время симуляции.
cs = getconfigset(m1);
cs.RuntimeOptions.StatesToLog = 'all';
Моделируйте модель.
simDataObj = sbiosimulate(m1);
Постройте график результатов.
sbioplot(simDataObj);
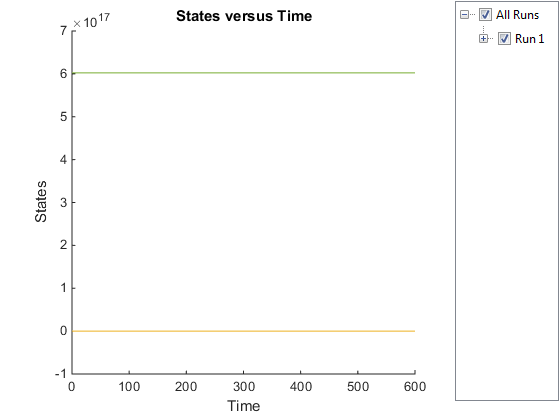
График не показывает разновидности причитающихся процентов к широкому спектру в разновидностях, amounts/concentrations.
Постройте только разновидности интереса. Ga.
GaSimDataObj = selectbyname(simDataObj,'Ga');
sbioplot(GaSimDataObj);
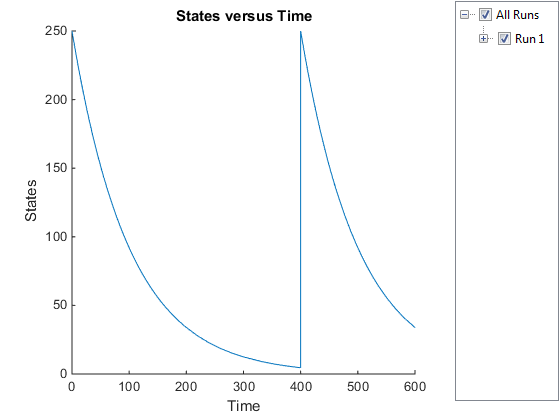
Заметьте изменение в профиле разновидностей Ga во время = секунды 400 (время симуляции). Это - время, когда сумма ингибитора изменяется, чтобы отразить пересложение ингибитора к модели.
Постройте только ингибитор (разновидности Inhib).
InhibSimDataObj = selectbyname(simDataObj,'Inhib');
sbioplot(InhibSimDataObj)