Можно исследовать структуру stats, которая возвращена и nlmefit и nlmefitsa, чтобы определить качество модели. Структура stats содержит поля со взвешенными невязками условного выражения (поле cwres), и человек взвесил невязки (поле iwres). Поскольку модель принимает, что невязки нормально распределены, можно исследовать невязки, чтобы видеть, как хорошо это предположение содержит.
Этот пример генерирует синтетические данные с помощью нормальных распределений. Это показывает, как подходящие статистические данные смотрят:
Хороший при тестировании против того же типа модели, как генерирует данные
Плохой, когда протестировано против неправильных моделей данных
Инициализируйте 2D модель с 100 людьми:
nGroups = 100; % 100 Individuals nlmefun = @(PHI,t)(PHI(:,1)*5 + PHI(:,2)^2.*t); % Regression fcn REParamSelect = [1 2]; % Both Parameters have random effect errorParam = .03; beta0 = [ 1.5 5]; % Parameter means psi = [ 0.35 0; ... % Covariance Matrix 0 0.51 ]; time =[0.25;0.5;0.75;1;1.25;2;3;4;5;6]; nParameters = 2; rng(0,'twister') % for reproducibility
Сгенерируйте данные для подбора кривой пропорциональной ошибочной моделью:
b_i = mvnrnd(zeros(1, numel(REParamSelect)), psi, nGroups); individualParameters = zeros(nGroups,nParameters); individualParameters(:, REParamSelect) = ... bsxfun(@plus,beta0(REParamSelect), b_i); groups = repmat(1:nGroups,numel(time),1); groups = vertcat(groups(:)); y = zeros(numel(time)*nGroups,1); x = zeros(numel(time)*nGroups,1); for i = 1:nGroups idx = groups == i; f = nlmefun(individualParameters(i,:), time); % Make a proportional error model for y: y(idx) = f + errorParam*f.*randn(numel(f),1); x(idx) = time; end P = [ 1 0 ; 0 1 ];
Соответствуйте данным с помощью той же модели функции и ошибки регрессии в качестве образцового генератора:
[~,~,stats] = nlmefit(x,y,groups, ... [],nlmefun,[1 1],'REParamsSelect',REParamSelect,... 'ErrorModel','Proportional','CovPattern',P);
Создайте стандартную программу графического вывода путем копирования следующего функционального определения и создания файла plotResiduals.m на пути MATLAB®:
function plotResiduals(stats) pwres = stats.pwres; iwres = stats.iwres; cwres = stats.cwres; figure subplot(2,3,1); normplot(pwres); title('PWRES') subplot(2,3,4); createhistplot(pwres); subplot(2,3,2); normplot(cwres); title('CWRES') subplot(2,3,5); createhistplot(cwres); subplot(2,3,3); normplot(iwres); title('IWRES') subplot(2,3,6); createhistplot(iwres); title('IWRES') function createhistplot(pwres) h = histogram(pwres); % x is the probability/height for each bin x = h.Values/sum(h.Values*h.BinWidth) % n is the center of each bin n = h.BinEdges + (0.5*h.BinWidth) n(end) = []; bar(n,x); ylim([0 max(x)*1.05]); hold on; x2 = -4:0.1:4; f2 = normpdf(x2,0,1); plot(x2,f2,'r'); end end
Постройте невязки с помощью функции plotResiduals:
plotResiduals(stats);
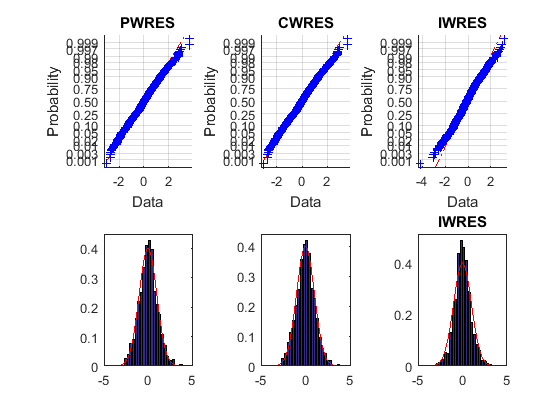
Верхние графики вероятности выглядят прямыми, означая, что невязки нормально распределены. Нижние графики гистограммы совпадают с наложенным нормальным графиком плотности. Таким образом, можно прийти к заключению, что ошибочная модель совпадает с данными.
Для сравнения, соответствуют данные с помощью постоянной ошибочной модели, вместо пропорциональной модели, которая создала данные:
[~,~,stats] = nlmefit(x,y,groups, ... [],nlmefun,[0 0],'REParamsSelect',REParamSelect,... 'ErrorModel','Constant','CovPattern',P); plotResiduals(stats);
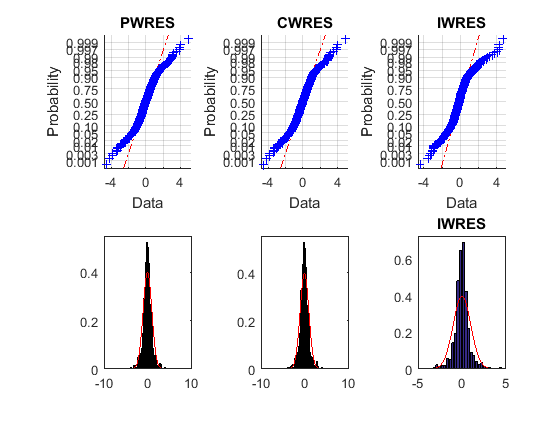
Верхние графики вероятности не являются прямыми, указывая, что невязки не нормально распределены. Нижние графики гистограммы справедливо близко к наложенным нормальным графикам плотности.
Для другого сравнения соответствуйте данным к различной структурной модели, чем созданный данные:
nlmefun2 = @(PHI,t)(PHI(:,1)*5 + PHI(:,2).*t.^4); [~,~,stats] = nlmefit(x,y,groups, ... [],nlmefun2,[0 0],'REParamsSelect',REParamSelect,... 'ErrorModel','constant', 'CovPattern',P); plotResiduals(stats);
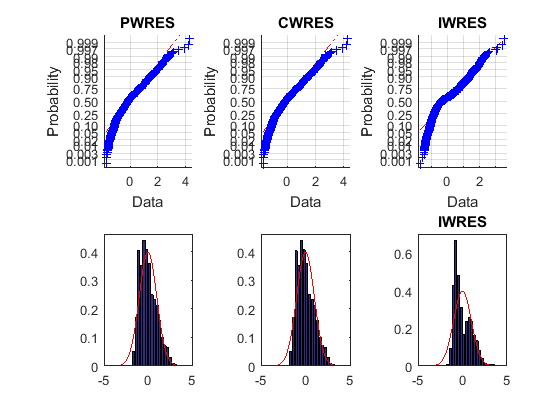
Мало того, что верхние графики вероятности не являются прямыми, но и график гистограммы, вполне скашивается по сравнению с наложенной нормальной плотностью. Эти невязки не нормально распределены, и не совпадают с моделью.