Запишите филогенетический древовидный объект в Newick-отформатированный файл
phytreewrite(File, Tree)
phytreewrite(Tree)
phytreewrite(..., 'Distances', DistancesValue,
...)
phytreewrite(..., 'BranchNames', BranchNamesValue,
...)
File | Вектор символов или строка, задающая Newick-отформатированный файл (текстовый ASCII-файл) имя, путь и имя файла или URL, указывающий на файл. |
Tree | Филогенетический древовидный объект, любой созданный с phytree (функция конструктора Object) или импортированное использование phytreeread функция. |
phytreewrite( копирует содержимое File, Tree)phytree объект от рабочей области MATLAB® до файла. Данные в файле используют формат Newick в описании деревьев.
phytreewrite( открывает диалоговое окно Save Phylogenetic Tree As для вас, чтобы ввести или выбрать имя файла.Tree)
phytreewrite (..., ' вызовы PropertyName', PropertyValue, ...)phytreewrite с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Заключите каждый PropertyName в одинарных кавычках. Каждый PropertyName является нечувствительным к регистру. Это имя свойства / пары значения свойства следующие:
phytreewrite(..., 'Distances', задает, исключить ли расстояния от выхода. DistancesValue,
...)DistancesValue может быть true (значение по умолчанию) или false.
phytreewrite(..., 'BranchNames', задает, исключить ли имена ветви из выхода. BranchNamesValue,
...)BranchNamesValue может быть true (значение по умолчанию) или false.
Считайте древовидные данные из Newick-отформатированного файла.
tr = phytreeread('pf00002.tree')
Phylogenetic tree object with 33 leaves (32 branches)Удалите все белки мыши и просмотрите сокращенное дерево.
ind = getbyname(tr,'mouse'); tr = prune(tr,ind); view(tr)
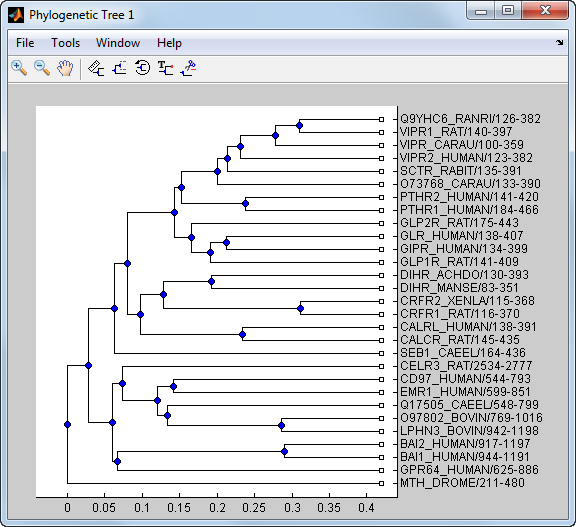
Запишите сокращенные древовидные данные в файл.
phytreewrite('newtree.tree',tr)multialignwrite | phytree | phytree object | phytreeread | phytreeviewer | seqlinkage