Чертите вторичную структуру последовательности RNA
rnaplot(RNA2ndStruct)
ha = rnaplot(RNA2ndStruct)
[ha, H]
= rnaplot(RNA2ndStruct)
rnaplot(RNA2ndStruct,
...'Sequence', SequenceValue, ...)
rnaplot(RNA2ndStruct, ...'Format', FormatValue,
...)
rnaplot(RNA2ndStruct, ...'Selection', SelectionValue,
...)
rnaplot(RNA2ndStruct, ...'ColorBy', ColorByValue,
...)
RNA2ndStruct | Вторичная структура последовательности RNA, представленной также:
Совет Используйте |
SequenceValue | Последовательность RNA вторичная структура, построенная, заданная любым из следующего:
RNA2ndStruct. Эта информация запрашивается, если вы задаете 'Diagram' формат или если вы задаете, чтобы подсветить какой-либо из следующих парных выборов: 'AU', 'UA', 'GC', 'CG', 'GU' или 'UG'. |
FormatValue | Вектор символов или строка, задающая формат графика. Выбор:
Примечание Если вы задаете |
SelectionValue | Любое из следующего:
Примечание Если вы задаете |
ColorByValue | Вектор символов или строка, задающая цветовую схему для графика. Выбор:
Примечание Если вы задаете Примечание Поскольку внутренние узлы дерева соответствуют парным остаткам, вы не можете задать |
ha | Обработайте к оси вращения. |
H | Структура указателей, содержащих подмножество следующих полей, на основе того, что вы задаете для 'Selection' и 'ColorBy' свойства:
|
rnaplot( чертит RNA вторичная структура, заданная RNA2ndStruct)RNA2ndStruct, вторичная структура последовательности RNA представлена вектором символов или строкой, задающей обозначение скобки или матрицу смежности.
ha = rnaplot(RNA2ndStruct)ha, указатель на ось вращения.
[ также возвращает ha, H]
= rnaplot(RNA2ndStruct)H, структура указателей, которые можно использовать для элементов графика в Окне рисунка MATLAB®.
Совет
Используйте указатели, возвращенные в H изменить свойства элементов графика, таких как цвет, размер маркера и тип маркера.
H содержит подмножество следующих полей, на основе того, что вы задаете для 'Selection' и 'ColorBy' свойства.
| Поле | Описание |
|---|---|
Paired | Указатели на все парные остатки |
Unpaired | Указатели на все непарные остатки |
A | Указатели на все остатки |
C | Указатели на все остатки C |
G | Указатели на все остатки G |
U | Указатели на все остатки U |
AU | Указатели на весь AU или пары UA |
GC | Указатели на весь GC или пары CG |
GU | Указатели на весь GU или пары UG |
Selected | Указатели на все выбранные остатки |
rnaplot ( вызовы RNA2ndStructPropertyName ', PropertyValue, ...)rnaplot с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
rnaplot( чертит RNA вторичная структура, заданная RNA2ndStruct,
...'Sequence', SequenceValue, ...)RNA2ndStruct, и аннотирует его положениями последовательности, предоставленными SequenceValue, последовательность RNA задана вектором символов, строкой или структурой, содержащей Sequence поле .
rnaplot( чертит RNA вторичная структура, заданная RNA2ndStruct, ...'Format', FormatValue,
...)RNA2ndStruct, использование формата задано FormatValue.
FormatValue вектор символов или строка, задающая формат графика. Выбор следующие.
| Формат | Описание |
|---|---|
'Circle' (значение по умолчанию) | Каждая основа представлена точкой на окружности круга произвольного размера. Линии соединяют базисы что пара друг с другом.
|
'Diagram' | Двумерное представление RNA вторичная структура. Каждая основа представлена и идентифицирована буквой. Магистраль и водородные связи между парами оснований представлены линиями.
Примечание Если вы задаете |
'Dotdiagram' | Двумерное представление RNA вторичная структура. Каждая основа представлена и идентифицирована точкой. Магистраль и водородные связи между парами оснований представлены линиями.
|
'Graph' | Базисы отображены в их положении последовательности вдоль абсциссы (ось X) графика. Полуэллиптические линии соединяют базисы что пара друг с другом. Высота линий пропорциональна расстоянию между парными базисами.
|
'Mountain' | Каждая основа представлена точкой в двумерном графике, где основное положение находится в абсциссе (ось X), и количество пар оснований, заключающих данную основу, находится в ординате (ось Y).
|
'Tree' | Каждая основа представлена узлом в древовидном графе. Вершины указывают на непарные базисы, в то время как каждый внутренний узел указывает на пару оснований. Древовидный корень является фиктивным узлом, не сопоставленным с любой основой во вторичной структуре.
|
rnaplot( чертит RNA вторичная структура, заданная RNA2ndStruct, ...'Selection', SelectionValue,
...)RNA2ndStruct, выделение подмножества остатков задано SelectionValue. SelectionValue может быть также:
Числовой массив, задающий индексы остатков, чтобы подсветить в графике.
Вектор символов или строка, задающая подмножество остатков, чтобы подсветить в графике. Выбор:
'Paired'
'Unpaired'
'AU' или 'UA'
'GC' или 'CG'
'GU' или 'UG'
Примечание
Если вы задаете 'AU', 'UA', 'GC', 'CG', 'GU', или 'UG', необходимо также использовать 'Sequence' свойство обеспечить последовательность RNA.
rnaplot( чертит RNA вторичная структура, заданная RNA2ndStruct, ...'ColorBy', ColorByValue,
...)RNA2ndStruct, использование цветовой схемы задано ColorByValue, вектор символов или строка, указывающая на цветовую схему. Выбор:
'State' (значение по умолчанию) — Цвет по парному состоянию: парные базисы и непарные базисы.
'Residue' — Цвет типом остатка (A, C, G, и U).
'Pair' — Цвет парным типом (AU/UA, GC/CG и GU/UG).
Примечание
Если вы задаете 'Residue' или 'Pair', необходимо также использовать 'Sequence' свойство обеспечить последовательность RNA.
Примечание
Поскольку внутренние узлы дерева соответствуют парным остаткам, вы не можете задать 'Residue' если вы задаете 'Tree' для 'Format' свойство.
Определите минимальную свободную энергию вторичная структура последовательности RNA и постройте его в круговом формате:
seq = 'GCGCCCGUAGCUCAAUUGGAUAGAGCGUUUGACUACGGAUCAAAAGGUUAGGGGUUCGACUCCUCUCGGGCGCG';
ss = rnafold(seq);
rnaplot(ss)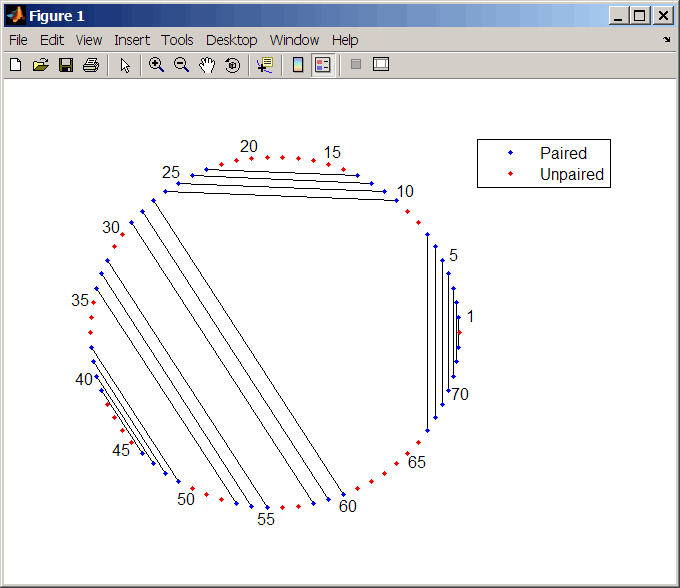
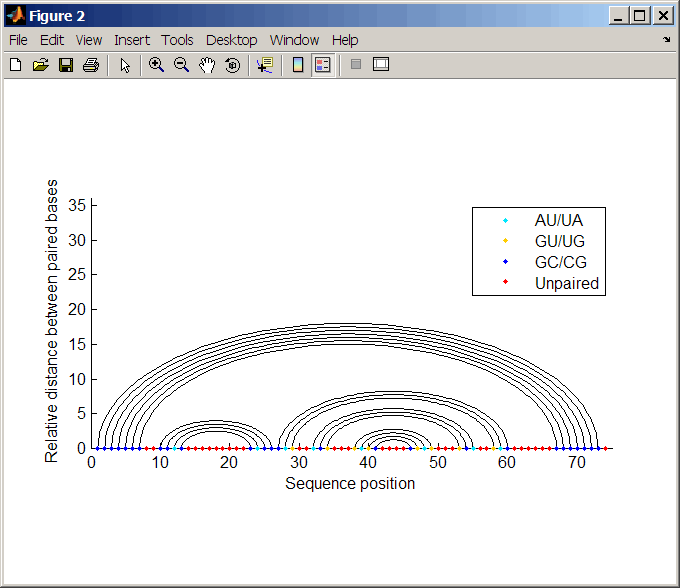
Постройте последовательность RNA вторичная структура в формате графика и окрасьте его парным типом.
rnaplot(ss, 'sequence', seq, 'format', 'graph', 'colorby', 'pair')
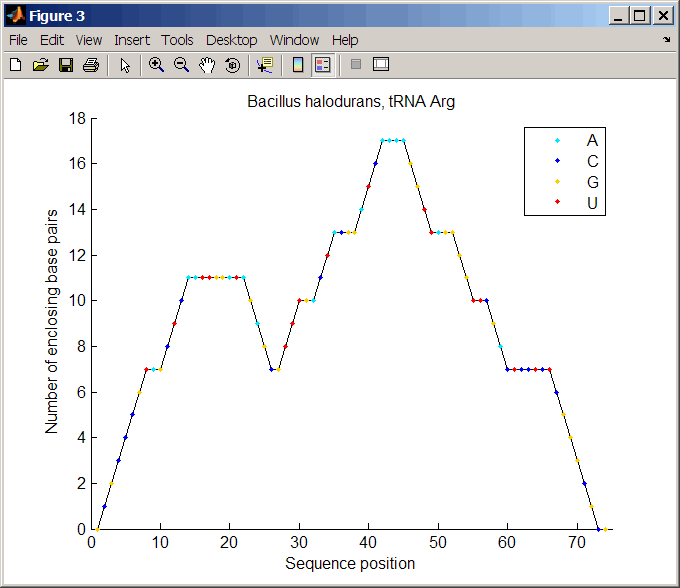
Постройте последовательность RNA вторичная структура в горном формате и окрасьте его типом остатка. Используйте указатель, чтобы добавить заголовок на график.
ha = rnaplot(ss, 'sequence', seq, 'format', 'mountain',... 'colorby', 'residue') title(ha, 'Bacillus halodurans, tRNA Arg')
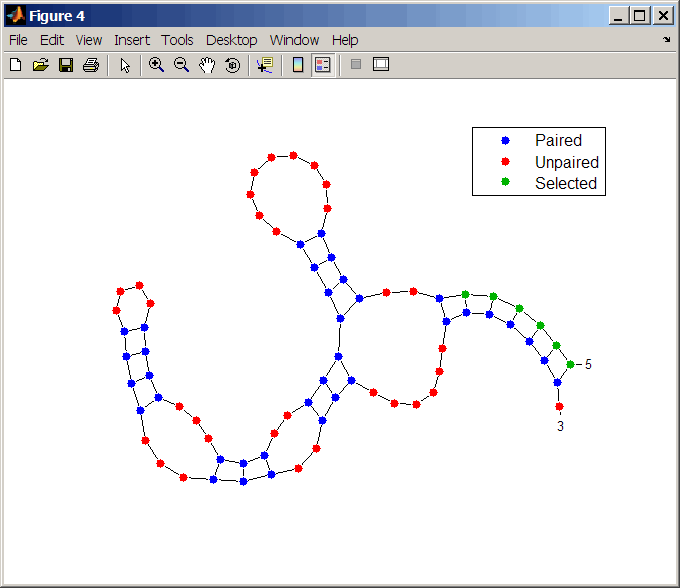
Видоизмените первые шесть положений в последовательности и наблюдайте влияние, которое изменение оказывает на вторичную структуру путем выделения первых шести остатков.
seqMut = seq; seqMut(1:6) = 'AAAAAA'; ssMut = rnafold(seqMut); rnaplot(ss, 'sequence', seq, 'format', 'dotdiagram', 'selection', 1:6); rnaplot(ssMut, 'sequence', seqMut, 'format', 'dotdiagram', 'selection', 1:6);
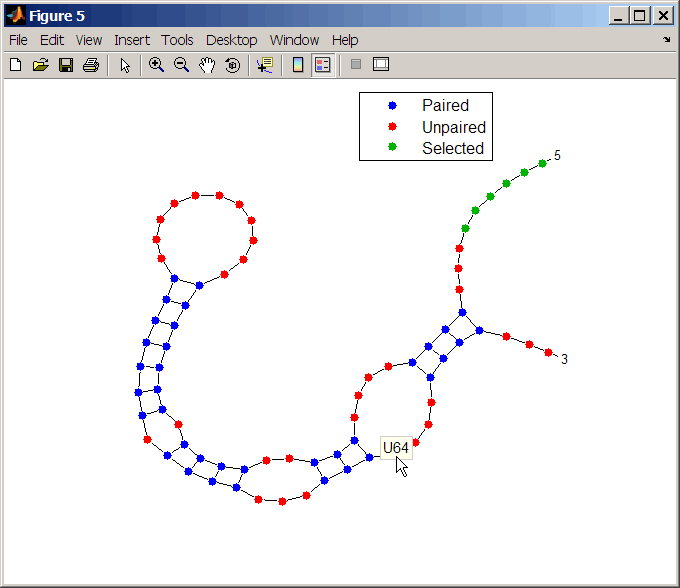
Совет
При необходимости перетащите нажатие кнопки легенду, чтобы препятствовать тому, чтобы он покрыл график. Кликните по основе в графике отобразить всплывающую подсказку с информацией о той основе.