Средство просмотра последовательностей интегрирует многие функции последовательности в панель инструментов Toolbox™ биоинформатики. Вместо ввода команд в окне команд MATLAB ® можно выбрать и ввести параметры с помощью приложения.
Первая стадия анализа нуклеотидной или аминокислотной последовательности заключается в импорте информации о последовательности в среду MATLAB. Средство просмотра последовательностей может подключаться к веб-базам данных, таким как NCBI и EMBL, и считывать информацию в среду MATLAB.
Следующая процедура иллюстрирует получение информации о последовательности из базы данных NCBI в Интернете. В этом примере используется регистрационный номер NM_000520 GenBank ®, который представляет собой человеческий ген HEXA, связанный с болезнью Тэя-Сакса.
Примечание
Данные в публичных хранилищах часто обрабатываются и обновляются; поэтому результаты этого примера могут несколько отличаться при использовании актуальных последовательностей.
В окне команды MATLAB введите
seqviewer
Либо щелкните Просмотр последовательностей (Sequence Viewer) на вкладке Приложения (Apps).
Средство просмотра последовательностей открывается без загруженной последовательности. Обратите внимание, что панели справа и снизу пусты.
Для получения последовательности из базы данных NCBI выберите File > Download Sequence from > NCBI.
Откроется диалоговое окно Загрузить последовательность из NCBI (Download Sequence from NCBI).
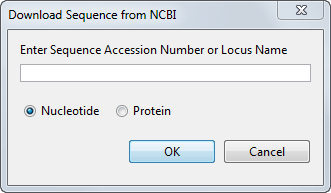
В поле Ввод последовательности введите регистрационный номер для записи базы данных NCBI, например, NM_000520. Нажмите кнопку «Параметр нуклеотида» и нажмите кнопку «ОК».
Программное обеспечение MATLAB получает доступ к базе данных NCBI в Интернете, загружает информацию о нуклеотидной последовательности для введенного номера присоединения и вычисляет некоторые основные статистические данные.
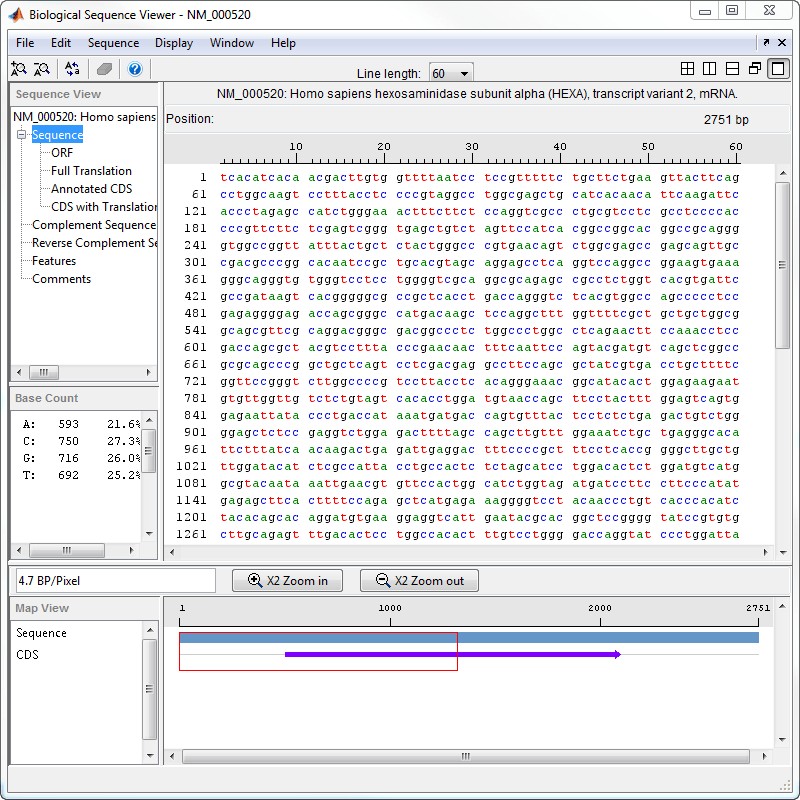
После импорта последовательности в приложение Sequence Viewer можно считывать информацию, хранящуюся вместе с последовательностью, или просматривать графические представления для ORF и CSS.
В дереве левой панели щелкните Комментарии. На правой панели отображается общая информация о последовательности.
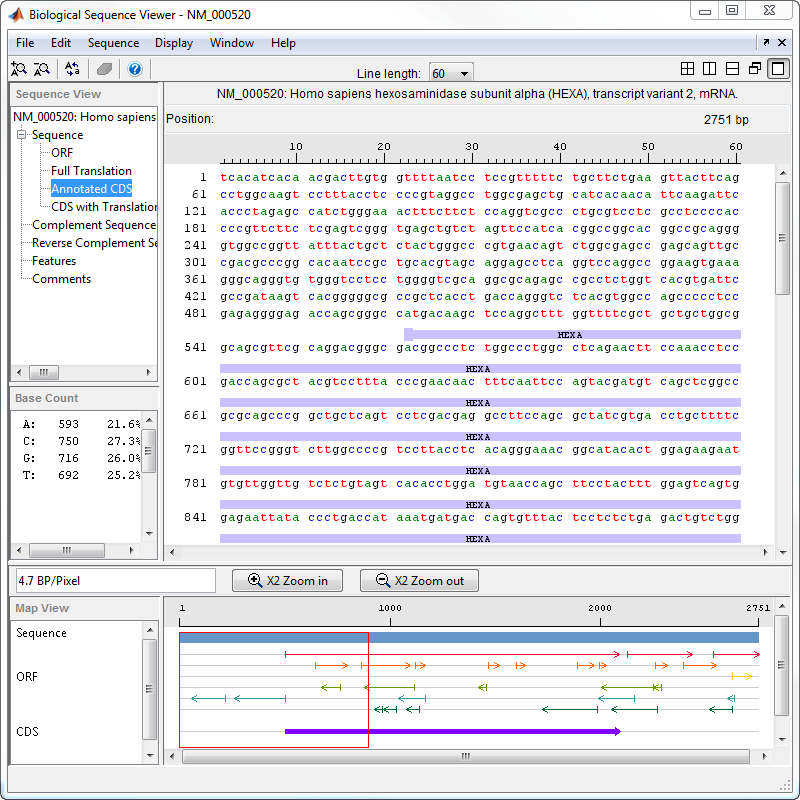
Теперь щелкните Элементы (Features). На правой панели отображается информация о функциях NCBI, включая номера индексов для гена и все последовательности CDS.
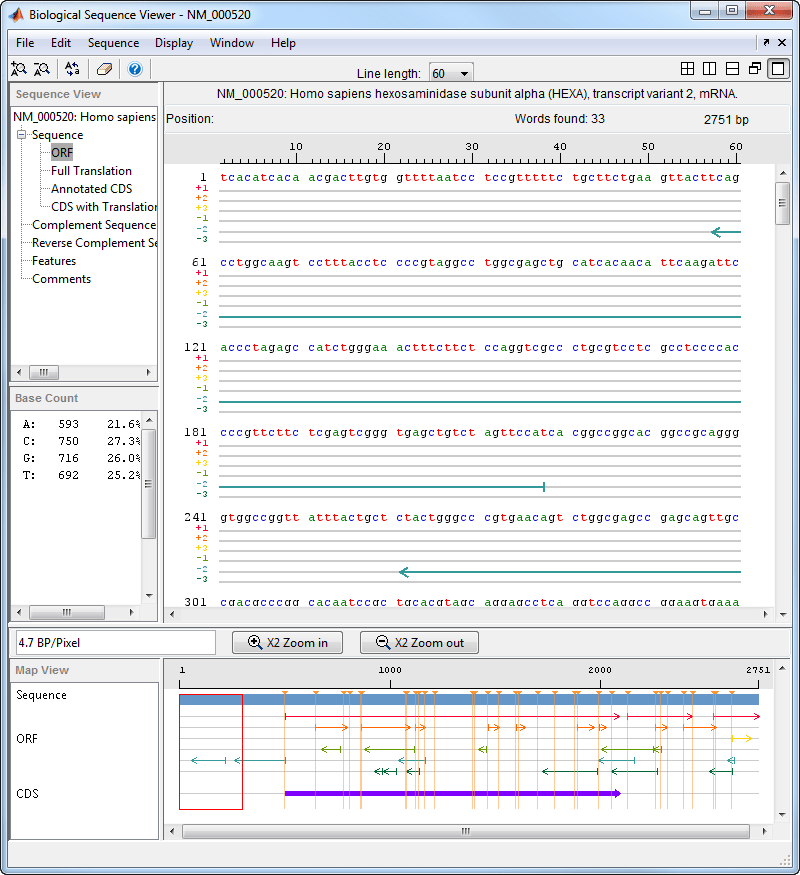
Щелкните ORF, чтобы показать результаты поиска ORF в шести кадрах считывания.
Щелкните Аннотированная CDS, чтобы показать белок, кодирующий часть нуклеотидной последовательности.
Также можно выполнять поиск характерных слов или шаблонов последовательностей с помощью регулярных выражений. Можно ввести нуклеотидные и аминокислотные символы IUB/IUPAC, которые автоматически преобразуются в соответствующие нуклеотиды и аминокислоты соответственно. Для получения подробной информации о том, как интерпретируются символы, см. таблицы нуклеотидной конверсии и аминокислотной конверсии seq2regexp. Например, при поиске слова 'TAR' если установлен флажок Регулярное выражение, приложение выделяет все вхождения 'TAA' и 'TAG' в последовательности с R = [AG].
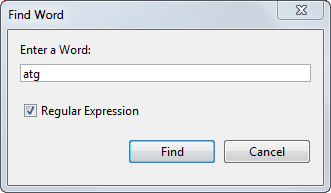
Выберите «Последовательность» > «Найти слово».
В диалоговом окне Найти слово (Find Word) введите слово или шаблон последовательности, например, atg, и нажмите кнопку Найти (Find).
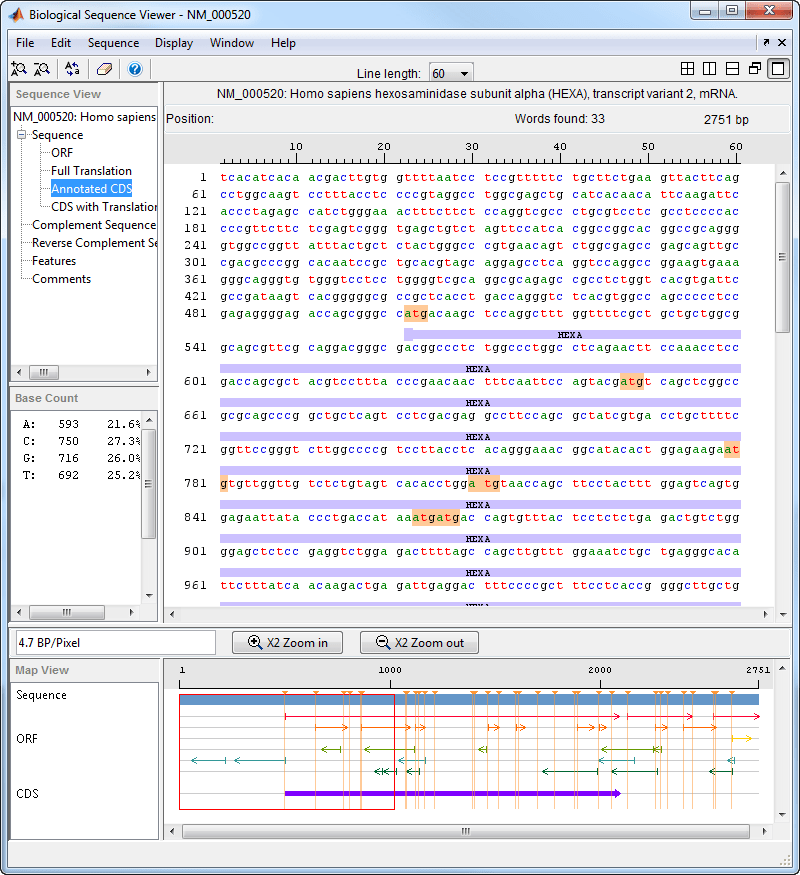
Средство просмотра последовательностей выполняет поиск и отображает местоположение выбранного слова.
Снимите флажок «Очистить выбор слова![]() » на панели инструментов.
» на панели инструментов.
Следующая процедура иллюстрирует, как идентифицировать белок, кодирующий часть нуклеотидной последовательности, и скопировать его в новое представление. Идентификация кодирующих участков нуклеотидной последовательности является общей задачей биоинформатики. После определения местоположения кодирующей части последовательности можно скопировать ее в новое представление, перевести в аминокислотную последовательность и продолжить анализ.
На левой панели нажмите кнопку ORF.
Средство просмотра последовательностей отображает ORF для шести кадров чтения на правой нижней панели. Наведите курсор на рамку для отображения информации о ней.
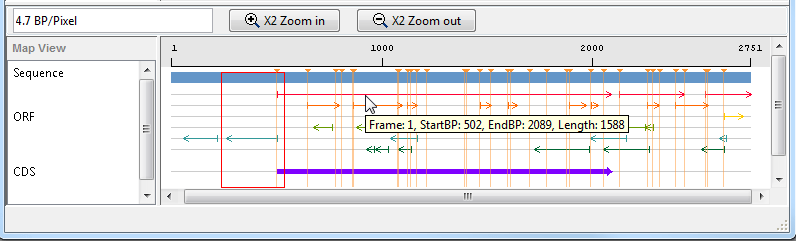
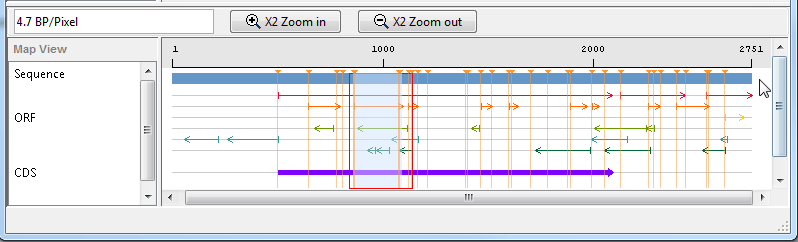
Щелкните самый длинный ORF на рамке считывания 2.
ОРФ подсвечивается для указания выбранной части последовательности.
Щелкните правой кнопкой мыши выбранный ORF и выберите Экспортировать в рабочую область (Export to Workspace). В диалоговом окне Экспорт в рабочую область MATLAB (Export to MATLAB Workspace) введите имя переменной, например, NM_000520_ORF_2, а затем щелкните Экспорт (Export).
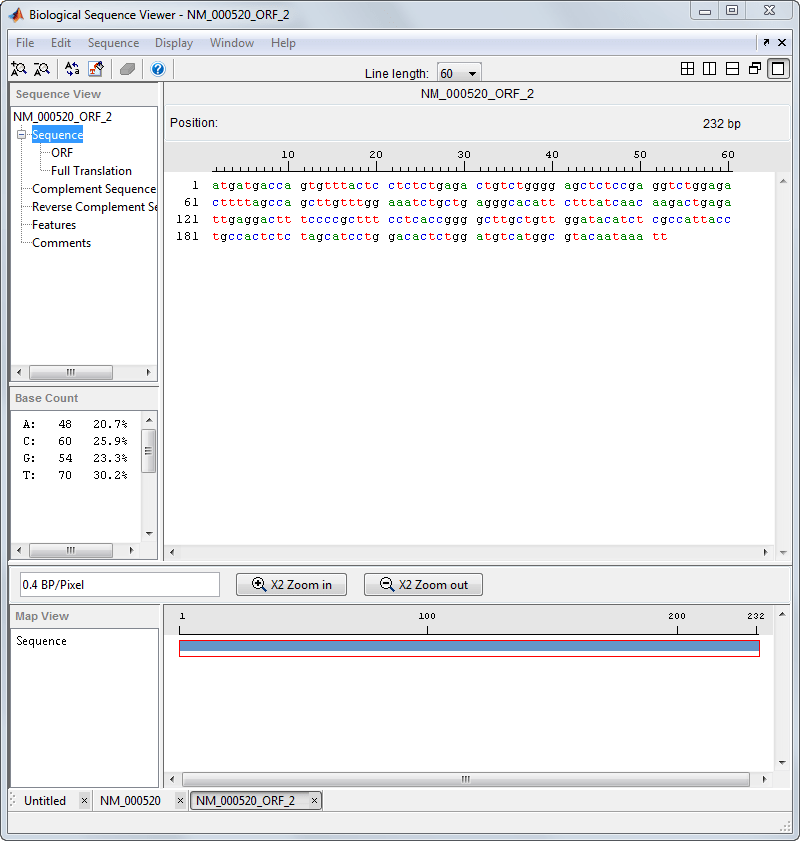
Переменная NM_000520_ORF_2 добавляется в рабочую область MATLAB.
Выберите меню «Файл» > «Импорт из рабочей области». Введите имя переменной с экспортированным ORF, например NM_000520_ORF_2, и нажмите кнопку Импорт.
Средство просмотра последовательностей добавляет вкладку в нижней части новой последовательности, оставляя исходную последовательность открытой.
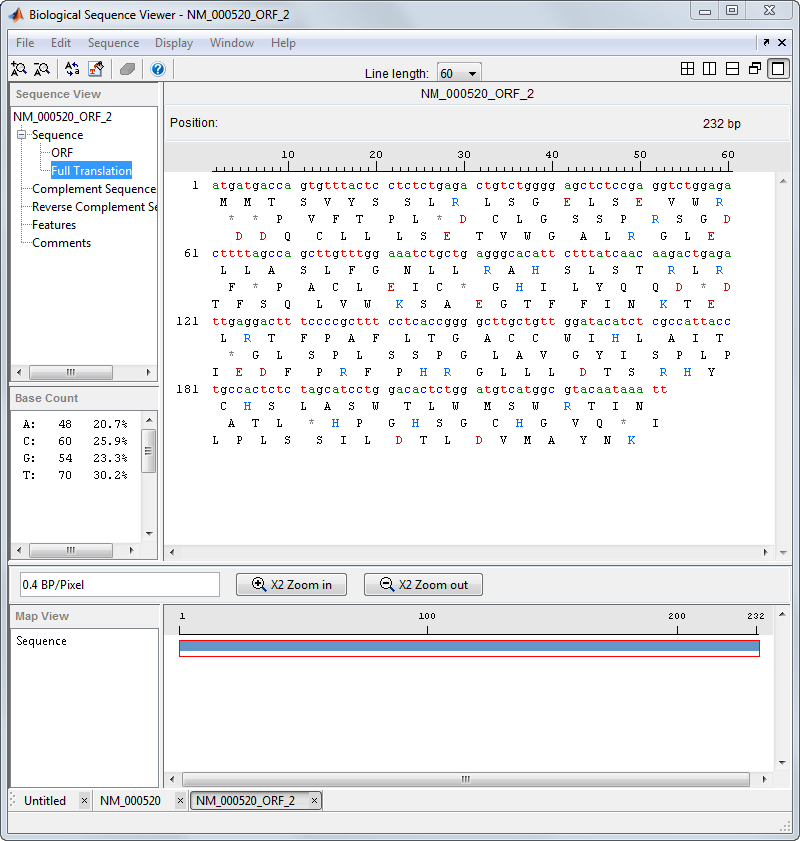
На левой панели щелкните Полный перевод. Выберите Отображение > Отображение аминокислотного остатка > Однобуквенный код.
Средство просмотра последовательностей отображает аминокислотную последовательность ниже нуклеотидной последовательности.
Закройте средство просмотра последовательностей из командной строки MATLAB, используя следующий синтаксис:
seqviewer('close')