Этот пример показывает, как импортировать данные от NONMEM-файла-формата. Данные могут быть в одних из следующих поддерживаемых форматов файлов:.XLSX.XLS.CSV, и.TXT.
Прежде, чем импортировать данные NONMEM-формата из файла, удалите любые комментарии, которые присутствуют в начале файла.
Откройте рабочий стол SimBiology® путем ввода simbiology в Командном окне MATLAB® или нажатия на SimBiology на вкладке Apps.
На вкладке Home выберите Add Data> Load Data from File.
Перейдите к папке matlabroot\help\toolbox\simbio\examplesmatlabroot является папкой, где MATLAB установлен. Откройте демонстрационный NONMEM-файл-формата под названием nonmem_bolus_dosing.txt. Этот файл содержит искусственно сгенерированные данные для 20 человек, получающих 5 доз шарика с 1 дозой шарика каждые 8 часов. Концентрации плазмы препарата были зарегистрированы в получасовых интервалах в течение 60 часов.
Если вы используете систему Macintosh, нажмите Command+Shift+G в диалоговом окне File Browser и введите полный путь в папку.
От диалогового окна Text File Import выберите Use NONMEM interpretation of headers.
Заголовок столбца CID соответствует каждому предмету (или группа) в наборе данных. Выберите group, чтобы идентифицировать столбец как столбец группы и нажать Update Preview.

Диалоговое окно импорта сопоставляет заголовки столбцов NONMEM с соответствующими категориями классификации данных для SimBiology, чтобы интерпретировать данные. Если существует неоднозначность, предупреждающее сообщение появляется в нижней части окна.
Нажмите OK, чтобы загрузить данные. SimBiology генерирует график рассеивания времени по сравнению с ответом для всех людей.
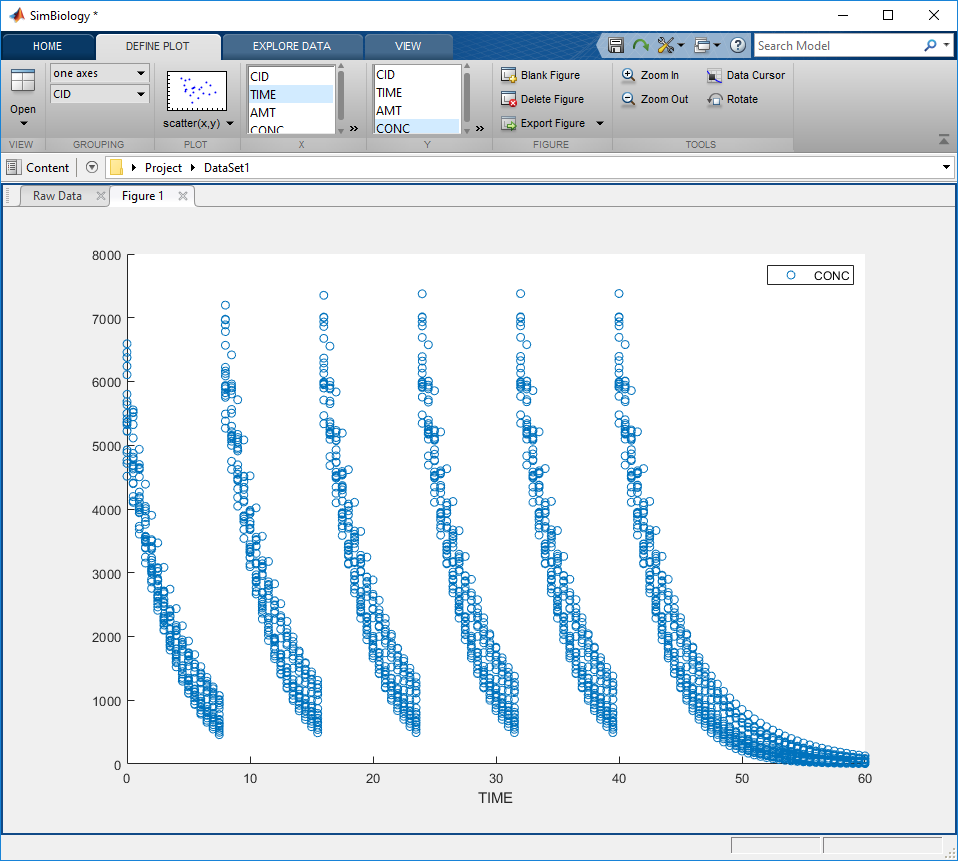
Кликните по вкладке Raw Data в нижней части графика просмотреть данные в табличной форме. Можно присвоить соответствующие модули с помощью выпадающего списка в соответствии с каждым столбцом.
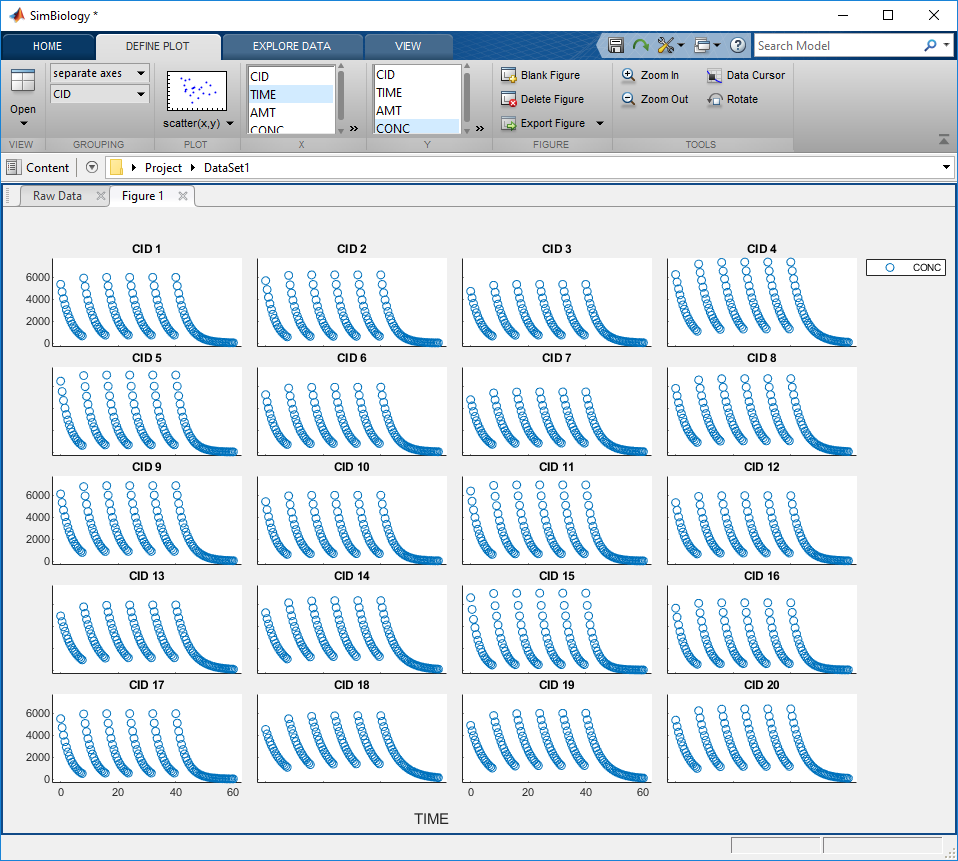
Отобразите данные с отдельными осями для каждого человека. Вернитесь к вкладке Figure 1. На вкладке Define Plot, в разделе Grouping, выбирают separate axes instead of one axes. SimBiology генерирует отдельные оси для всех людей.
SimBiology позволяет вам использовать разовые концентрацией данные, чтобы вычислить неразделенный на отсеки анализ (NCA) параметры включая AUC, разрешение и терминальную половину жизни. Для получения дополнительной информации см. NCA.
К параметрам представления NCA для каждой группы (человек) выберите Open> NCA. В Group Data Column выберите CID. Опционально, если ваши данные имеют два уровня иерархии группы, можно использовать ID Data Column, чтобы задать уровень подгруппы. Для получения дополнительной информации смотрите Данные.

После того, как вы измените установки параметров, SimBiology обновляет предполагаемые параметры NCA для каждого человека в Table of NCA Parameters. Можно также выбрать который столбцы (параметры), чтобы отобразиться путем щелчка правой кнопкой по любому заголовку столбца и выбора имен столбцов из выпадающего списка.

Можно экспортировать параметры как table или отдельные переменные к рабочему пространству MATLAB. Щелкните правой кнопкой где угодно в таблице и выберите опцию Export Statistics.