Мультисигнализирует о 1D кластеризации
S = mdwtcluster(X)
S = mdwtcluster(X,'PropName1',PropVal1,'PropName2',PropVal2,...)
S = mdwtcluster(X) кластеры построений от иерархического кластерного дерева. Входная матрица X анализируется в направлении строки с помощью функции DWT с haar вейвлет и максимальный допустимый уровень.
S = mdwtcluster(X,'PropName1',PropVal1,'PropName2',PropVal2,...) позволяет вам изменять некоторые свойства. Допустимый выбор для PropName :
mdwtcluster требует Statistics and Machine Learning Toolbox™
'dirDec' |
|
'level' | Уровень разложения DWT. Значение по умолчанию: |
'wname' | Имя вейвлета используется в DWT. Значением по умолчанию является |
'dwtEXTM' | Режим расширения DWT (см. |
'pdist' | См. Statistics and Machine Learning Toolbox |
'linkage' | См. Statistics and Machine Learning Toolbox |
'maxclust' | Количество кластеров. Значение по умолчанию равняется 6. Входная переменная может быть вектором. |
'lst2clu' | Массив ячеек, который содержит список данных, чтобы классифицировать. Если
Значением по умолчанию является |
Структура output S такова это для каждого раздела j:
S.Idx(:,j) | Содержит кластерные числа, полученные из иерархического кластерного дерева (см. |
S.Incons(:,j) | Содержит противоречивые значения каждого узла, не являющегося листом в иерархическом кластерном дереве (см., что программное обеспечение Statistics and Machine Learning Toolbox функционирует |
S.Corr(j) | Содержит cophenetic коэффициенты корреляции раздела (см., что программное обеспечение Statistics and Machine Learning Toolbox функционирует |
Если maxclustVal вектор, затем IdxCLU многомерный массив, таким образом что IdxCLU(:,j,k) содержит кластерные числа, полученные из иерархического кластерного дерева для k кластеры.
load elecsig10
lst2clu = {'s','ca1','ca3','ca6'};
% Compute the structure resulting from multisignal clustering
S = mdwtcluster(signals,'maxclust',4,'lst2clu',lst2clu)
S =
IdxCLU: [70x4 double]
Incons: [69x4 double]
Corr: [0.7920 0.7926 0.7947 0.7631]
% Retrieve indices of clusters
IdxCLU = S.IdxCLU;
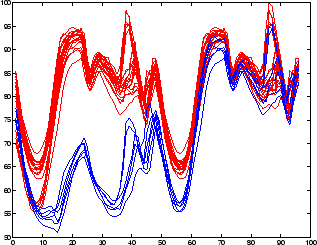
% Plot the first cluster
plot(signals(IdxCLU(:,1)==1,:)','r');
hold on;
% Plot the third clustering
plot(signals(IdxCLU(:,1)==3,:)','b')
% Check the equality of partitions
equalPART = isequal(IdxCLU(:,1),IdxCLU(:,3))
equalPART =
1
% So we can see that we obtain the same partitions using
% coefficents of approximation at level 3 instead of original
% signals. Much less information is then used.