Sequence Viewer интегрирует многие функции последовательности в тулбоксе Bioinformatics Toolbox™. Вместо того, чтобы ввести команды в Командном окне MATLAB®, можно выбрать и ввести опции с помощью приложения.
Следующая процедура иллюстрирует, как просмотреть последовательность аминокислот для ORF, расположенного в последовательности нуклеотида. Можно импортировать собственную последовательность аминокислот, или можно получить последовательность белка от базы данных GenBank®. Этот пример использует инвентарный номер GenBank NP_000511.1, который является альфа-подблоком для человеческого фермента, сопоставленного с болезнью Тея-Сакса.
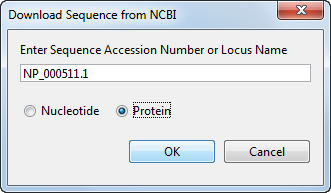
Выберите File > Download Sequence from > NCBI.
Диалоговое окно Download Sequence from NCBI открывается.
В поле Enter Sequence введите инвентарный номер для записи базы данных NCBI, например, NP_000511.1. Кликните по переключателю Protein, и затем нажмите OK.
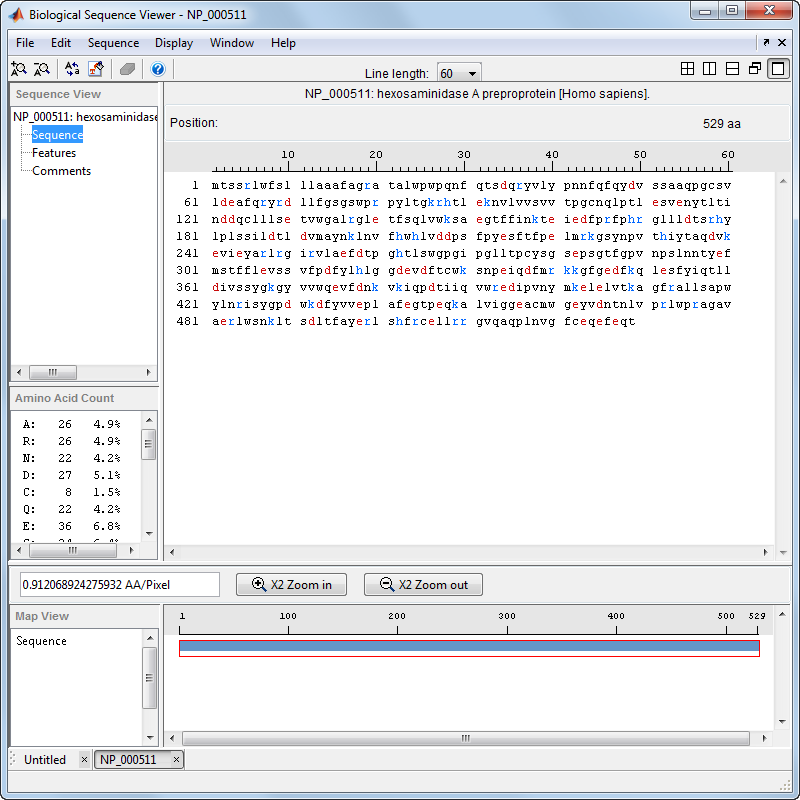
Sequence Viewer получает доступ к базе данных NCBI в сети и загружает информацию о последовательности аминокислот для инвентарного номера, который вы ввели.
Выберите Display > Amino Acid Color Scheme, и затем выберите Charge, Function, Hydrophobicity, Structure или Taylor. Например, выберите Function.
Цвета отображения превращаются в информацию о заряде подсветки об остатках аминокислоты. Следующая таблица показывает цветные легенды для цветовых схем аминокислоты.
| Цветовая схема аминокислоты | Цветная легенда |
|---|---|
| Заряд |
|
| Функция |
|
| Гидрофобность |
|
| Структура |
|
| Тейлор | Каждая аминокислота присвоена ее собственный цвет, на основе цветов, предложенных В.Р. Тейлором. |
Закройте Sequence Viewer из командной строки MATLAB с помощью следующего синтаксиса:
seqviewer('close')[1] Тейлор, W.R. (1997). Остаточные цвета: предложение по aminochromography. Разработка белка 10, 7, 743–746.