NGS Browser позволяет вам визуально проверить и заняться расследованиями, выравнивание последовательности читает в ссылочную последовательность, в поддержку исследований, которые измеряют наследственную изменчивость и экспрессию гена. NGS Browser позволяет вам:
Визуализируйте чтения последовательности, выровненные к последовательности ссылки нуклеотида.
Сравните несколько наборов данных, выровненных с последовательностью общей ссылки.
Просмотрите покрытие различных базисов и области ссылочной последовательности.
Исследуйте качество и другие детали выровненных чтений.
Идентифицируйте несоответствия из-за вызывающих основу ошибок или полиморфизмов.
Визуализируйте вставки и удаления.
Получите аннотации функции относительно определенной области ссылочной последовательности.
Исследуйте необходимые области в выравнивании, определенном различными исследованиями.
Можно визуализировать и исследовать выровненные данные прежде, во время, или после любой предварительной обработки (фильтрация, качественная перекалибровка) или аналитические шаги, которые вы выполняете на выровненных данных.
Чтобы открыть NGS Browser, введите следующее в командном окне MATLAB:
ngsbrowser
В качестве альтернативы нажмите NGS Browser на вкладке Apps.
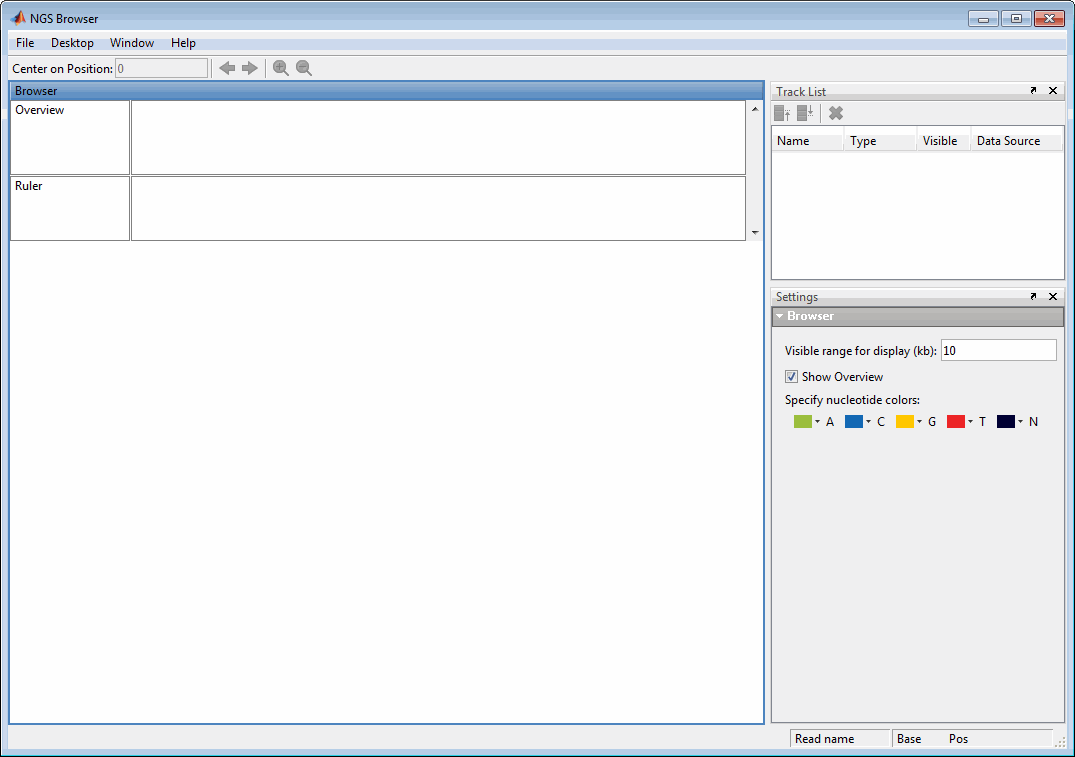
Браузер, отображающий ссылочную дорожку, одну дорожку выравнивания и одну дорожку аннотации
Можно импортировать одну ссылочную последовательность в NGS Browser. Ссылочная последовательность должна быть в файле FASTA.
Выберите File > Add Data from File.
В диалоговом окне Open выберите файл FASTA, и затем нажмите Open.
Совет
Можно использовать getgenbank функция с ToFile и SequenceOnly аргументы пары "имя-значение", чтобы получить ссылочную последовательность из базы данных GenBank и сохранить его в FASTA-отформатированный файл.
Можно импортировать несколько наборов данных считанных данных о выравнивании последовательности. Данные о выравнивании должны быть в любом из следующего:
BioMap объект
Совет
Создайте объект BioMap из SAM-или отформатированного BAM файла, чтобы заняться расследованиями, подмножество, и отфильтровать данные прежде, чем импортировать его в NGS Browser.
SAM-или отформатированный BAM файл
Примечание
Ваш SAM-или отформатированный BAM файл должны:
Имейте чтения, упорядоченные положением запуска в ссылочной последовательности.
Имейте индексный файл IDX (для SAM-отформатированного файла) или BAI и индексные файлы LINEARINDEX (для отформатированного BAM файла) сохраненный в том же месте как ваш исходный файл. В противном случае исходный файл должен храниться в месте, к которому у вас есть доступ для записи, потому что MATLAB® должен создать и сохранить индексные файлы в этом месте.
Совет
Попытайтесь использовать SAMtools, чтобы проверять, упорядочены ли чтения в вашем SAM-или отформатированном BAM файле положением в ссылочной последовательности, и также переупорядочить их, в случае необходимости.
Совет
Если у вас нет индексных файлов (IDX или BAI и LINEARINDEX) сохраненный в том же месте как ваш исходный файл, и ваш исходный файл хранится в месте, к которому у вас нет доступа для записи, вы не можете импортировать данные от исходного файла непосредственно в браузер. Вместо этого создайте BioMap объект от исходного файла с помощью IndexDir аргумент пары "имя-значение", и затем импортирует объект BioMap в браузер.
Импортировать данные о выравнивании чтения последовательности:
Выберите File > Add Data from File или File > Import Alignment Data from MATLAB Workspace.
Выберите файл SAM-formatted, отформатированный BAM файл или объект BioMap.
Если вы выбираете файл, содержащий несколько ссылочных последовательностей в диалоговом окне Select Reference, выберите ссылку или отсканируйте файл для доступных ссылок и их сопоставленных количеств чтений. Нажмите OK.
Повторите предыдущие шаги, чтобы импортировать дополнительные наборы данных.
Можно импортировать несколько наборов аннотаций функции от GFF-или отформатированных GTF файлов, которые содержат данные для одной ссылочной последовательности.
Выберите File > Add Data from File.
В диалоговом окне Open выберите GFF-или файл GTF-formatted, и затем нажмите Open.
Повторите предыдущие шаги, чтобы импортировать дополнительные аннотации.
Увеличивать и уменьшать масштаб:
| Используйте |
| или перетащите нажатие кнопки ребро rubberband в области Overview. |
|
|
К панорамированию через выравнивание:
| Используйте |
| или перетащите нажатие кнопки rubberband в области Overview. |
|
|
Совет
Используйте левые и правые клавиши со стрелками для панорамирования в одной паре оснований (BP) шаг.
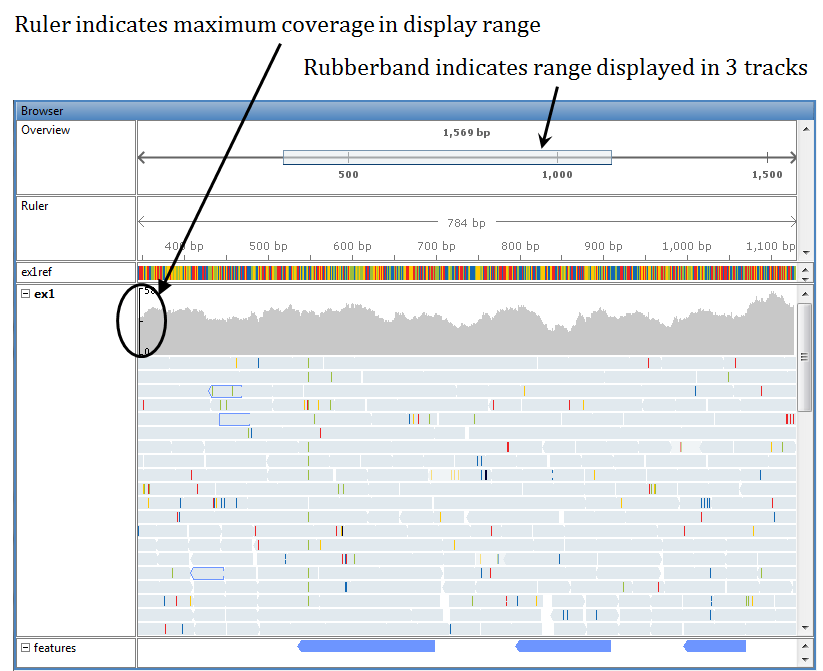
Наверху каждой дорожки выравнивания представление покрытия отображает покрытие каждой основы в ссылочной последовательности. Вертикальная линейка на левом крае представления покрытия указывает на максимальное покрытие в области значений отображения. Наведите указатель мыши на положение в представлении покрытия, чтобы отобразить местоположение и количества.
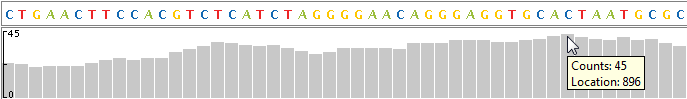
Примечание
Браузер вычисляет покрытие в разрешении пары оснований, вместо раскладывания, даже когда уменьшено масштаб.
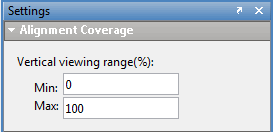
Чтобы изменить отображенное покрытие процента, щелкните где угодно в дорожке выравнивания, и затем измените настройки Alignment Coverage.
Совет
Установите Max на значение, больше, чем 100, в случае необходимости, при сравнении покрытия нескольких дорожек чтений.
Каждая дорожка выравнивания включает представление нагона коротких чтений, выровненных к ссылочной последовательности.
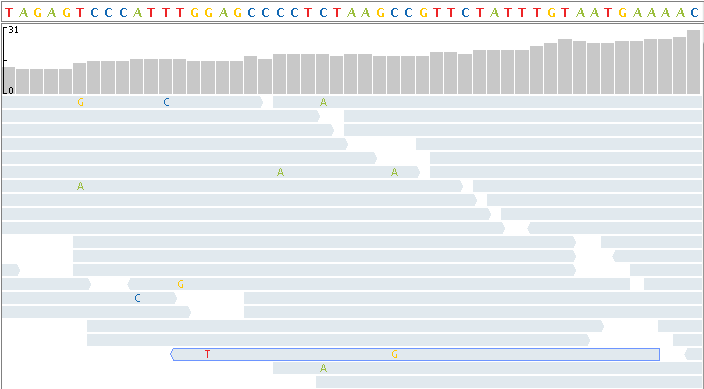
Ограничьте глубину чтений, отображенных в представлении нагона путем установки Maximum display read depth в настройках Alignment Pileup.
Совет
Ограничение глубины коротких чтений в представлении нагона не изменяет количества, отображенные в представлении покрытия.
Сравните несколько наборов данных, с каждым набором данных в его собственной дорожке, против последовательности общей ссылки. Используйте Track List, чтобы показать/скрыть, заказать, и удалить дорожки данных.
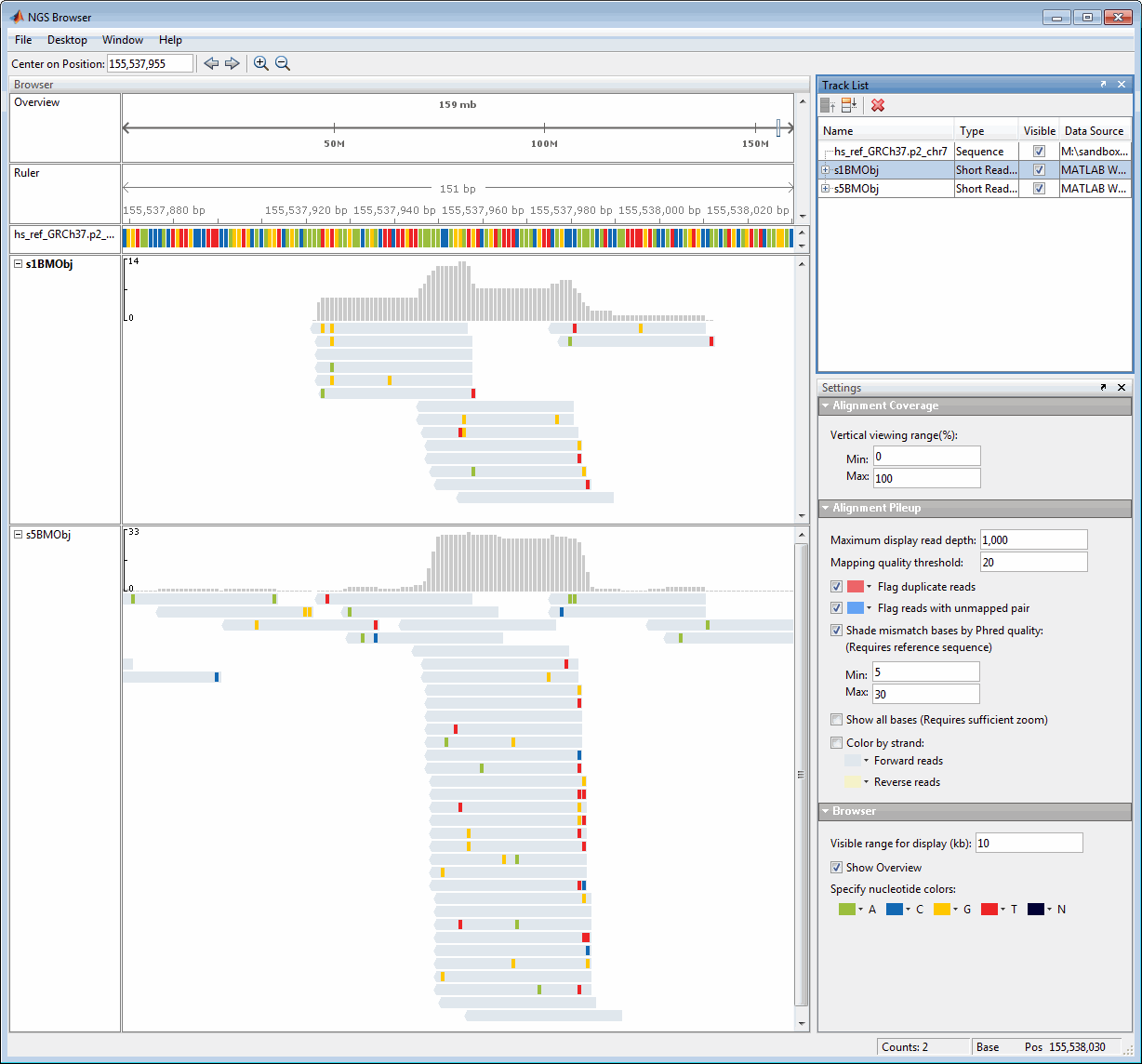
Наведите указатель мыши на положение в чтении, чтобы отобразить направление скрутки, местоположение, качество и информацию об отображении для основы, чтения и его парного помощника.

Щелкните где угодно в дорожке выравнивания, чтобы отобразить настройки Alignment Pileup.
Установите Mapping quality threshold в разделе Alignment Pileup отмечать низкокачественные чтения. Чтения с качеством отображения ниже этого уровня появляются в более легком оттенке серого.
Выберите Flag duplicate reads и выберите цвет контура.
Выберите Flag reads with unmapped pair и выберите цвет контура.
Несоответствия отображаются как окрашенные блоками или буквами, в зависимости от уровня изменения масштаба.
Уменьшивший масштаб представление чтения — Несоответствия отображаются как панели
![]()
Масштабируемый ввиду чтения — Несоответствия отображаются как буквы
![]()
В дополнение к основной информации о качестве Phred, которая отображается в подсказке, можно визуализировать качественные различия при помощи настроек Shade mismatch bases by Phred quality.
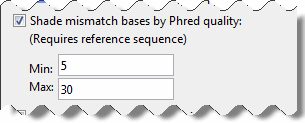
Блоки несоответствия или буквы отображаются в:
Легкий оттенок — Несоответствие базируется с баллами Phred ниже минимума
Церемония вручения дипломов средних оттенков — Несоответствие базируется с баллами Phred в минимуме к максимальной области значений
Темный оттенок — Несоответствие базируется с баллами Phred выше максимума
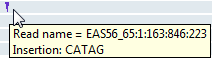
NGS Browser определяет вставки с![]() символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию об этом.
символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию об этом.
NGS Browser определяет удаления с тире.

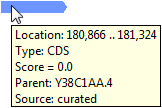
После импорта файла аннотации функции можно масштабировать и панорамирование, чтобы просмотреть аннотации функции, сопоставленные с необходимой областью в выравнивании. Наведите указатель мыши на аннотацию функции.
Распечатайте или экспортируйте изображение браузера путем выбора File > Print Image или File > Export Image.