В этом примере показано, как исследовать и тестировать конструкции с фиксированной точкой путем параллельного распределения тестов между многими компьютерами. В примере используется parfor контур для проверки точности алгоритма детектора QRS. Для выполнения параллельного моделирования требуется лицензия Parallel Computing Toolbox™.
Для выполнения этого примера скопируйте файлы примеров в локальный рабочий каталог.
copyfile(fullfile(matlabroot,'help','toolbox','fixpoint', 'examples',... 'parallel_examples','heart_rate_detector'))
В локальной рабочей папке откройте и запустите тестовый файл, в котором выполняется пример.
edit test_heart_rate_detector_in_parallel.m
test_heart_rate_detector_in_parallelКак стандарт for-loop, a parfor (Parallel Computing Toolbox) - цикл выполняет ряд инструкций в диапазоне значений. Использование parfor команда, можно настроить параллельную for-закольцовывание в коде для изучения конструкций с фиксированной точкой путем распределения тестов между многими компьютерами. В parfor цикл, итерации цикла выполняются параллельно, что может обеспечить более высокую производительность, чем стандартные for-контуры.
Этот скрипт устанавливает тестируемую систему и инициализирует массивы, которые будут содержать результаты вне parfor-луп. Затем используется цикл parfor для параллельного тестирования каждой записи. parforФункция -loop загружает данные, запускает систему, затем классифицирует и сохраняет результаты параллельно. Когда parfor-loop завершает, сценарий отображает результаты. Дополнительные сведения о тестируемой системе см. в разделе Описание тестируемой системы.
%% Run test of records in database in parallel record_names = {'ecg_01','ecg_02','ecg_03','ecg_04','ecg_05','ecg_06',... 'ecg_07','ecg_08','ecg_09','ecg_10','ecg_11','ecg_12','ecg_13'}; %% Set up the system under test data_type = 'fixedwrap'; T = heart_rate_detector_types(data_type); [mex_function_name,Fs_target] = setup_heart_rate_detector(record_names,data_type,T); %% Initialize array to contain results results_file_names = cell(size(record_names)); %% Test each record in the database in parallel parfor record_number = 1:length(record_names); % Load data record_name = record_names{record_number}; [ecg,tm,ann,Fs] = load_ecg_data(record_name,Fs_target); % Run system under test detector_outputs = run_heart_rate_detector(mex_function_name,ecg,T); % Classify results [qrs_struct,qrs_stats] = classify_qrs(ann, Fs, detector_outputs); % Save results results_file_names{record_number} = save_heart_rate_data(... mex_function_name,record_name,... data_type,ecg,tm,ann,Fs,... detector_outputs,... qrs_struct,qrs_stats); end %% Display results display_ecg_results(record_names, results_file_names);
Поскольку итерации цикла должны быть полностью независимы друг от друга, вызов save и load команды непосредственно внутри parfor-луп. Однако можно вызывать функции, которые вызывают эти команды. В этом примере функции load_ecg_data и save_heart_rate_data загрузите и сохраните необходимые данные. Дополнительные сведения об ограничениях см. в разделе Параллельные контуры (parfor) (Панель инструментов параллельных вычислений).
Тестируемая система в этом примере тестирует простой детектор QRS, который измеряет разницу во времени между обнаружениями QRS для вычисления частоты сердечных сокращений. test_heart_rate_detector_in_parallel сценарий передает записи ЭКГ алгоритму обнаружения.
Следующий график является примером, когда алгоритм детектора правильно идентифицирует обнаружения QRS для вычисления контрольного сигнала.
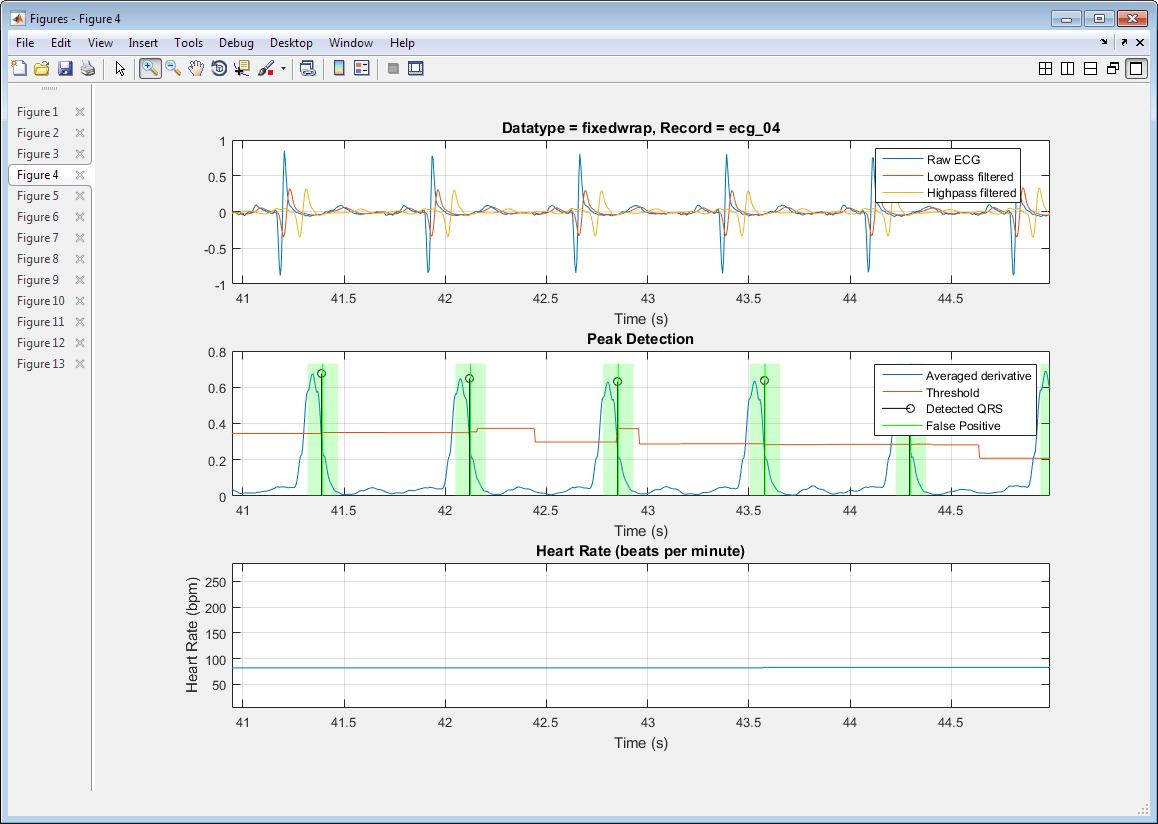
Алгоритм обнаружения упрощается для этого примера. Анализ графиков и результатов, отображаемых при выполнении примера, показывает, что алгоритм не всегда является очень точным.
Record #QRS TP FP FN DER Se +P
ecg_01 253 195 1 58 23.32 77.08 99.49
ecg_02 133 133 18 0 13.53 100.00 88.08
ecg_03 94 94 1 0 1.06 100.00 98.95
ecg_04 92 91 0 1 1.09 98.91 100.00
ecg_05 93 91 1 2 3.23 97.85 98.91
ecg_06 131 131 22 0 16.79 100.00 85.62
ecg_07 174 173 2 0 1.15 100.00 98.86
ecg_08 117 116 10 1 9.40 99.15 92.06
ecg_09 137 137 1 0 0.73 100.00 99.28
ecg_10 96 96 3 0 3.13 100.00 96.97
ecg_11 73 73 1 0 1.37 100.00 98.65
ecg_12 146 145 71 0 48.63 100.00 67.13
ecg_13 144 144 5 0 3.47 100.00 96.64
Totals 1683 1619 136 62 11.76 96.31 92.25
Legend:
#QRS: Total number of QRS Complexes
TP: Number of true positive
FP: Number of false positive
FN: Number of false negative
DER: Detection error rate in percent
Se: Sensitivity in percent
+P: Positive prediction in percent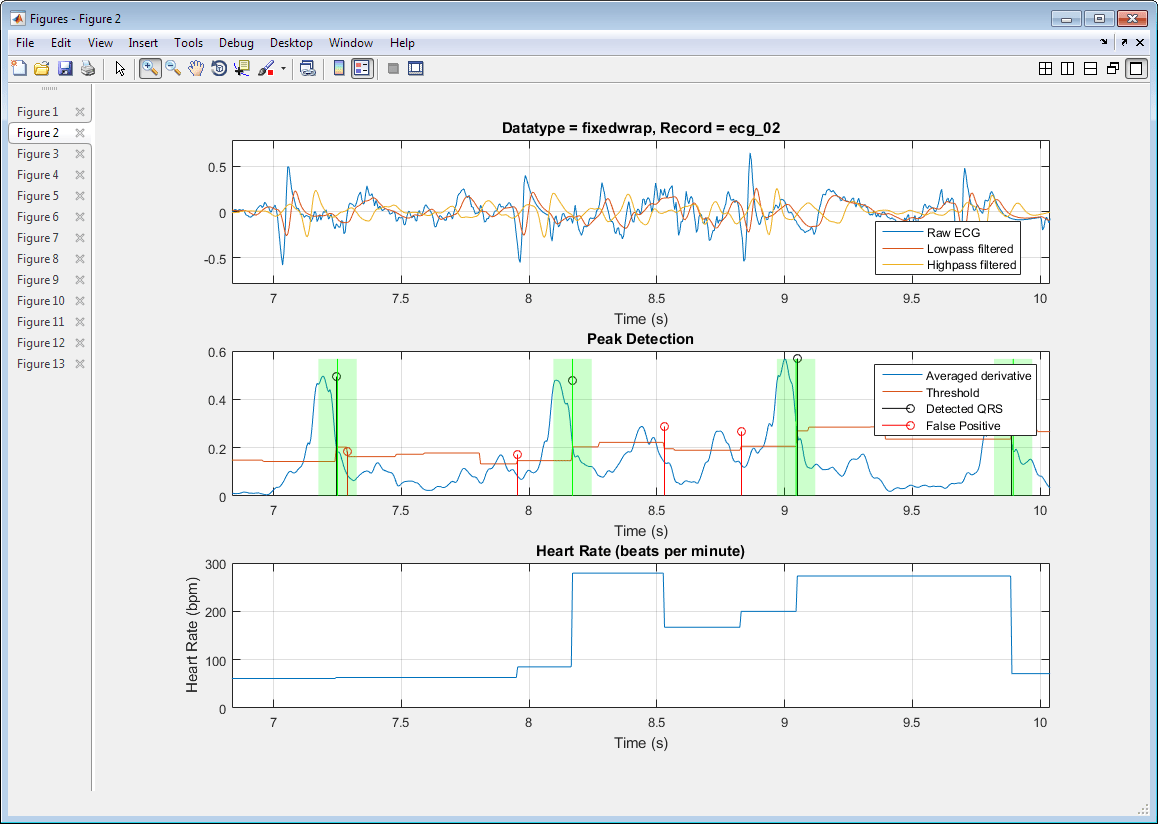
Примечание
Все записи ЭКГ, использованные в этом примере, измеряли на оборудовании для хоббистов.
Вы можете использовать базу данных PhysioNet записанных физиологических сигналов для выполнения аналогичного анализа самостоятельно.
Аннотации на этих записях не были проверены врачами.
[1] Патрик С. Гамильтон, программное обеспечение для анализа ЭКГ с открытым исходным кодом (OSEA), E.P. Limited, Somerville, MA, http://www.eplimited.com, 2002.
[2] Гари Д. Клиффорд, Франсиско Азуадже и Патрик Э. МакШарри. Передовые методы и инструменты для анализа данных ЭКГ, Artech House, 2006.
[3] Американский национальный стандарт ANSI/AAMI EC38:2007 Медицинское электрооборудование - Часть 2-47: Особые требования к безопасности, включая основные характеристики, амбулаторных электрокардиографических систем, Ассоциация по развитию медицинского приборостроения, 2008.
[4] Джордж Б. Муди, «Оценка анализаторов ЭКГ», Руководство по применению базы данных WaveForm, Отдел медицинских наук и технологий Гарвардского Массачусетского технологического института, Кембридж, Массачусетс, WFDB10.5.23, 13 марта 2014 года.
[5] Ида Лайла бинти Ахмад, Маснани бинти Мохамед, Норул Айн бинти Ав Гани, «Разработка демонстратора концепции для обнаружения комплекса QRS с использованием комбинированных алгоритмов», Международная конференция IEEE EMBS 2012 года по биомедицинской инженерии и наукам, Лангкан
[6] Р. Харикумар, С. Н. Шиваприя, «Анализ алгоритма обнаружения QRS для сердечных аномалий - Обзор», Международный журнал мягких вычислений и инженерии (IJSCE), ISSN: 2231-2307, Volume-1, Issue-5, ноябрь 2011.