Симулируйте модель SimBiology, добавляя изменений путем дискретизации модели ошибки
[ynew,parameterEstimates]
= random(resultsObj)
[ynew,parameterEstimates]
= random(resultsObj,data,dosing)
[ возвращает результаты симуляции ynew,parameterEstimates]
= random(resultsObj)ynew с добавленным шумом с помощью информации модели ошибки, заданной resultsObj.ErrorModelInfo свойство и предполагаемые значения параметров parameterEstimates.
[ использует заданную ynew,parameterEstimates]
= random(resultsObj,data,dosing)data и dosing информацию.
Примечание
Шум добавляется только к состояниям, которые являются ответами, которые являются состояниями, включенными в responseMap входной параметр при вызове sbiofit. Если для каждого отклика существует отдельная модель ошибки, шум добавляется к каждому отклику отдельно с помощью соответствующей модели ошибки.
resultsObj - Результаты оценкиOptimResults | объекта NLINResults объектРезультаты оценки, заданные как OptimResults
object или NLINResults object, который содержит результаты оценки, возвращенные sbiofit. Это должен быть скалярный объект.
data - Сгруппированные данные или время выходаgroupedData вектор | объект | массив ячеек из векторовСгруппированные данные или время выхода, заданное как groupedData object, вектор или массив ячеек из векторов выхода раз.
Если это вектор временных точек, random моделирует модель с новыми временными точками, используя оценки параметров из объекта результатов resultsObj.
Если это массив ячеек из векторов временных точек, random n раз симулирует модель, используя время выхода из каждого временного вектора, где n - длина data.
Если это groupedData объект должен иметь независимую переменную, такую как Time. Она также должна иметь групповую переменную, если обучающие данные, используемые для подбора кривой, имеют такую переменную. Можно использовать groupedData объект, чтобы запросить различные комбинации категорий, если resultsObj содержит оценки параметров для каждой категории. random моделирует модель для каждой группы с заданными категориями. Например, предположим, что у вас есть набор оценок параметров для половой категории (мужчины и женщины) и возрастной категории (молодые и старые) в ваших обучающих данных. Можно использовать random моделировать ответы старого мужчины (или любой другой комбинации), хотя такой пациент может не существовать в обучающих данных.
Если на resultsObj из оценки параметров конкретной категории, data должен быть groupedData объект.
Примечание
Если UnitConversion включен для базовой модели SimBiology, которая использовалась для подбора кривой и data является groupedData объект, data необходимо задать допустимые модули через data.Properties.VariableUnits свойство. Если это численный вектор или массив ячеек из векторов временных точек, random использует TimeUnits модели.
dosing - Информация о дозах[] | 2-D матрицу объектов дозы SimBiologyИнформация о дозах, заданная как пустой массив [] или 2-D матрица объектов дозы SimBiology (ScheduleDose object или RepeatDose object). Если dosing является матрицей объектов дозы, матрица должна содержать дозы по умолчанию или соответствовать исходным данным дозирования, используемым с sbiofit. То есть дозируйте объекты в dosing должны иметь те же значения свойств дозы (такие как TargetName) или должны быть параметризованы так же, как и исходные данные дозирования. Например, предположим, что исходная дозирующая матрица имеет два столбца доз, где дозы в первой колонке-мишени x и дозы во второй колонке-мишени y. Затем dosing должны иметь дозы в первом столбце, нацеленные на виды x и во втором столбце, нацеленные на виды y.
Если он пуст, никакие дозы не применяются во время симуляции, даже если модель имеет активные дозы.
Если не пустой, матрица должна иметь одну строку или по одной строке на группу во входных данных. Если она имеет одну строку, те же дозы применяются ко всем группам во время симуляции. Если она имеет несколько строк, каждая строка применяется к отдельной группе в том же порядке, в котором группы появляются во входных данных. Если одни группы (или временные векторы) требуют больше доз, чем другие, то заполните матрицу дозами по умолчанию (манекен).
Допускается использование нескольких столбцов, чтобы можно было применить несколько объектов дозы к каждой группе или временному вектору. Все дозы в столбце должны быть дозами по умолчанию или должны ссылаться на те же компоненты в модели (для образца дозы должны иметь ту же цель дозы TargetName) и должны иметь согласованные параметризованные свойства, как в исходных данных дозирования, используемых с sbiofit. Для примера, если Amount свойство дозы, используемой в оригинале sbiofit вызов параметризован в параметр с параметрами model-scoped 'A', все дозы для соответствующей группы (столбца) в dosing должны иметь Amount свойство параметризовано в 'A'.
Доза по умолчанию имеет значения по умолчанию для всех свойств, кроме Name свойство. Создайте дозу по умолчанию следующим образом.
d1 = sbiodose('d1');В дополнение к ручному построению объектов дозы с помощью sbiodose, если входные данные являются groupedData объект и имеет информацию о дозах, вы можете использовать createDoses способ конструирования доз из него.
Количество строк в dosing матрица и количество групп или выходных временных векторов в data определить общее количество результатов моделирования в выход ynew. Для получения дополнительной информации см. таблицу в ynew описание аргумента.
Примечание
Если UnitConversion включен для базового SimBiology® модель, которая использовалась для подбора кривой, dosing необходимо указать допустимую величину и временные модули.
ynew - Результаты симуляции с шумомSimData объектыРезультаты симуляции, возвращенные как вектор SimData объекты. Информация о состояниях представлена в ynew являются состояниями, которые были включены в responseMap входной параметр sbiofit а также любые другие состояния, перечисленные в StatesToLog свойство опций среды выполнения (RuntimeOptions) модели SimBiology.
Общее количество результатов симуляции в ynew зависит от количества групп или выходных временных векторов в data и количество строк в dosing матрица.
Количество групп или выходных временных векторов в data | Количество строк в dosing матрица | Результаты симуляции |
|---|---|---|
|
| Общее количество Дозы во время симуляции не применяются. |
|
| Общее количество Данная строка доз применяется во время симуляции. |
| N | Общее количество Каждая строка |
N |
| Общее количество Дозы во время симуляции не применяются. |
N |
| Общее количество К каждой симуляции применяется одна и та же строка доз. |
| N | N | Общее количество Каждая строка |
| M | N | Функция выдает ошибку при M ≠ N. |
parameterEstimates - Предполагаемые значения параметровПредполагаемые значения параметров, возвращенные как таблица. Это идентично resultsObj.ParameterEstimates свойство.
Этот пример использует модель гетеротримерного G-белка дрожжей и экспериментальные данные, представленные [1]. Для получения дополнительной информации о модели смотрите раздел «Фон» в Параметр Сканирование, Оценка Параметра и Анализ чувствительности в дрожжевом гетеротримерном цикле G-белка.
Загрузите модель G-белка.
sbioloadproject gproteinВведите экспериментальные данные, содержащие временной курс для фракции активного G-белка, как указано в справочной статье [1].
time = [0 10 30 60 110 210 300 450 600]'; GaFracExpt = [0 0.35 0.4 0.36 0.39 0.33 0.24 0.17 0.2]';
Создайте groupedData объект на основе экспериментальных данных.
tbl = table(time,GaFracExpt); grpData = groupedData(tbl);
Сопоставьте соответствующий компонент модели с экспериментальными данными. Другими словами, укажите, какой вид в модели соответствует какой переменной отклика в данных. В этом примере сопоставьте параметр модели GaFrac в переменную экспериментальных данных GaFracExpt от grpData.
responseMap = 'GaFrac = GaFracExpt'; Использование estimatedInfo объект для определения параметра модели kGd как параметр, который будет оценен.
estimatedParam = estimatedInfo('kGd');Выполните оценку параметра. Используйте аргумент пары "имя-значение" 'ErrorModel' чтобы задать модель ошибки, которая добавляет ошибку к данным моделирования.
fitResult = sbiofit(m1,grpData,responseMap,estimatedParam,'ErrorModel','proportional');
Просмотрите предполагаемое значение параметров kGd.
fitResult.ParameterEstimates
ans =
Name Estimate StandardError
_____ ________ _____________
'kGd' 0.11 0.00064116 Используйте random метод для извлечения данных моделирования с добавленным шумом с помощью пропорциональной модели ошибки, которая была задана как sbiofit. Обратите внимание, что шум добавляется только к состоянию отклика, то есть GaFrac параметр.
[ynew,paramEstim] = random(fitResult);
Выберите данные моделирования для GaFrac параметр.
GaFracNew = select(ynew,{'Name','GaFrac'});Постройте график результатов симуляции.
plot(GaFracNew.Time,GaFracNew.Data)
hold on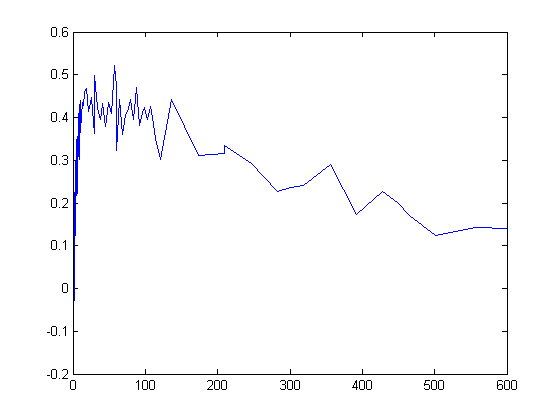
Постройте график экспериментальных данных, чтобы сравнить их с моделируемыми данными.
plot(time,GaFracExpt,'Color','k','Marker','o') legend('GaFracNew','GaFracExpt')
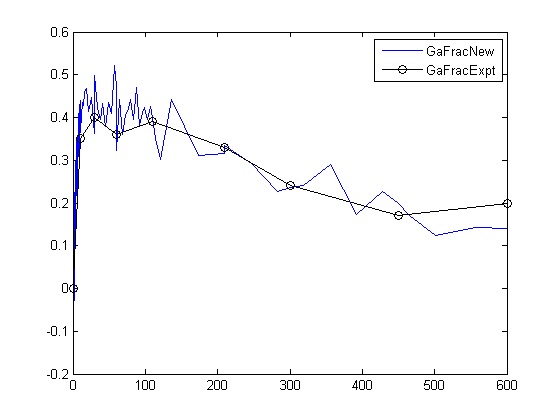
[1] Yi, T-M., Kitano, H. and Simon, M. (2003). Количественная характеристика дрожжевого гетеротримерного G-белкового цикла. PNAS. 100, 10764–10769.
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.