Создайте интенсивность по сравнению с графиком рассеивания отношения микроданных массива
mairplot(DataX, DataY)
[Intensity, Ratio]
= mairplot(DataX, DataY)
[Intensity, Ratio, H]
= mairplot(DataX, DataY)
... = mairplot(...,
'Type', TypeValue, ...)
... = mairplot(...,
'LogTrans', LogTransValue, ...)
... = mairplot(...,
'FactorLines', FactorLinesValue, ...)
... = mairplot(...,
'Title', TitleValue, ...)
... = mairplot(...,
'Labels', LabelsValue, ...)
... = mairplot(...,
'Normalize', NormalizeValue, ...)
... = mairplot(...,
'LowessOptions', LowessOptionsValue,
...)
... = mairplot(...,
'Showplot', ShowplotValue, ...)
... = mairplot(...,
'PlotOnly', PlotOnlyValue, ...)
DataX, DataY | Объект DataMatrix или вектор из значений экспрессии гена, где каждая строка соответствует гену. Например, в двухцветном эксперименте микромассивов, DataX могли быть cy3 значения интенсивности и DataY могли быть cy5 значения интенсивности. |
TypeValue | Вектор символов или строка, которая задает тип графика. Выбором является 'IR' (графики log10 продукта DataX и DataY интенсивность по сравнению с log2 отношений интенсивности) или 'MA' (графики (1/2) log2 продукта DataX и DataY интенсивность по сравнению с log2 отношений интенсивности). Значением по умолчанию является 'IR'. |
LogTransValue | Управляет преобразованием данных в X и Y от натурального звукоряда до шкалы log2. Установите LogTransValue к false, когда данные уже являются log2 шкалой. Значением по умолчанию является true, который принимает, что данные являются натуральным звукорядом. |
FactorLinesValue | Добавляет линии на график, показывающий фактор N изменение. Значение по умолчанию равняется 2, который соответствует уровню 1 и-1 по шкале log2.Совет Можно также изменить факторные линии в интерактивном режиме после создания графика. |
TitleValue | Вектор символов или строка, которая задает заголовок для графика. |
LabelsValue | Массив ячеек из символьных векторов или вектор строки, содержащий метки для данных. Если метки заданы, то нажатие на точку на графике показывает метку, соответствующую той точке. |
NormalizeValue | Управляет отображением нормированных значений отношения lowess. Введите true отображаться к lowess нормировало значения отношения. Значением по умолчанию является false. Совет Можно также нормировать данные из окна MAIR Plot после создания графика. |
LowessOptionsValue | Массив ячеек один, два, или три имени свойства / пары значения в любом порядке, которые влияют на lowess нормализацию. Выбор для имени свойства / пары значения:
Для получения дополнительной информации о предыдущем имени свойства / пары значения, смотрите |
ShowplotValue | Управляет отображением графика рассеивания. Выбором является true (значение по умолчанию) или false. |
PlotOnlyValue | Управляет отображением графика рассеивания без компонентов пользовательского интерфейса. Выбором является Примечание Если вы устанавливаете |
Intensity | Объект DataMatrix или вектор, содержащий значения интенсивности для данных об экспрессии гена микромассивов, вычисленных как:
Примечание Если |
Ratio | Объект DataMatrix или вектор, содержащий отношения данных об экспрессии гена микромассивов, вычисленных как log2 (.Примечание Если |
H | Указатель графика. |
mairplot( создает график рассеивания, который строит log10 продукта DataX, DataY)DataX и DataY интенсивность по сравнению с log2 отношений интенсивности.
[ возвращает значения отношения и интенсивность. Если вы устанавливаете Intensity, Ratio]
= mairplot(DataX, DataY)'Normalize' к true, возвращенные значения отношения нормированы.
[ возвращает указатель графика.Intensity, Ratio, H]
= mairplot(DataX, DataY)
... = mairplot (..., 'PropertyName', PropertyValue, ...)mairplot с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
задает тип графика. Выбором является ... = mairplot(...,
'Type', TypeValue, ...)'IR' (графики log10 продукта DataX и DataY интенсивность по сравнению с log2 отношений интенсивности) или 'MA' (графики (1/2) log2 продукта DataX и DataY интенсивность по сравнению с log2 отношений интенсивности). Значением по умолчанию является 'IR'.
... = mairplot(...,
'LogTrans', LogTransValue, ...)X и Y от естественного, чтобы регистрировать 2 шкалы. Установите LogTransValue к false, когда данные уже являются журналом 2 шкалы. Значением по умолчанию является true, который принимает, что данные являются натуральным звукорядом.
... = mairplot(...,
'FactorLines', FactorLinesValue, ...)N
Совет
Можно также изменить факторные линии в интерактивном режиме после создания графика.
... = mairplot(...,
'Title', TitleValue, ...)
... = mairplot(...,
'Labels', LabelsValue, ...)
... = mairplot(...,
'Normalize', NormalizeValue, ...)true отображаться к lowess нормировало значения отношения. Значением по умолчанию является false.
Совет
Можно также нормировать данные из окна MAIR Plot после создания графика.
... = mairplot(...,
'LowessOptions', LowessOptionsValue,
...)
'Порядок', OrderValue
'Устойчивый', RobustValue
'Промежуток', SpanValue
Для получения дополнительной информации о предыдущих трех именах свойства / пары значения, смотрите malowess функция.
... = mairplot(...,
'Showplot', ShowplotValue, ...)true (значение по умолчанию) или false.
... = mairplot(...,
'PlotOnly', PlotOnlyValue, ...)true или false (значение по умолчанию).
Примечание
Если вы устанавливаете 'PlotOnly' свойство к true, можно все еще отобразить метки для точек данных путем нажатия на точку данных, и можно все еще настроить горизонтальные линии изменения сгиба перетаскиванием нажатия кнопки линии.
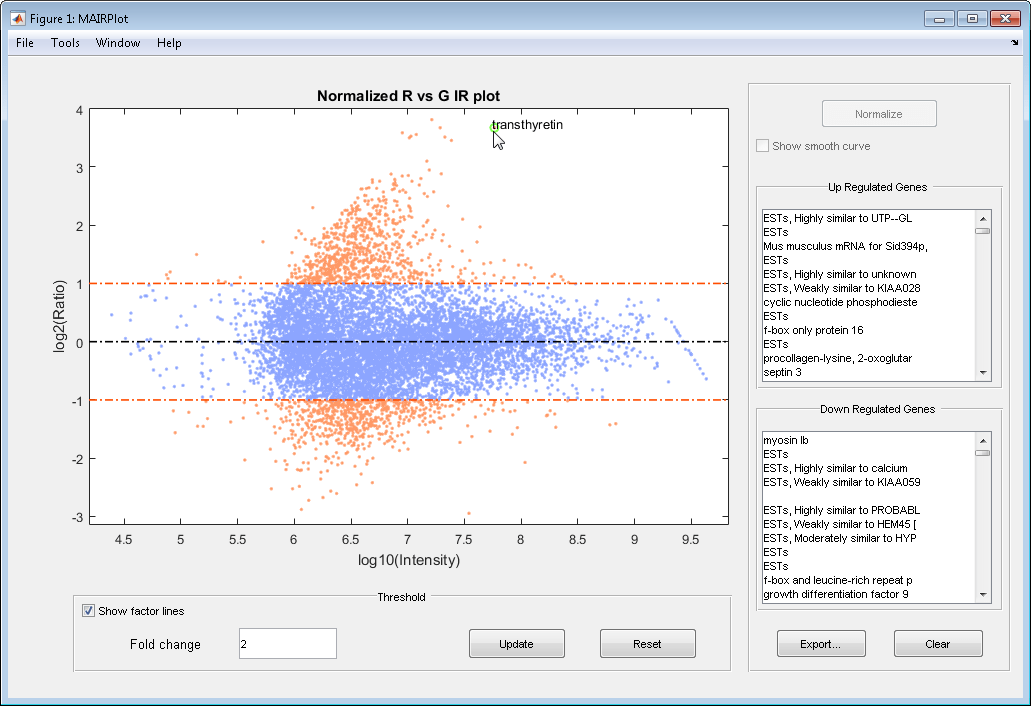
Следующее является графиком IR нормированных данных.
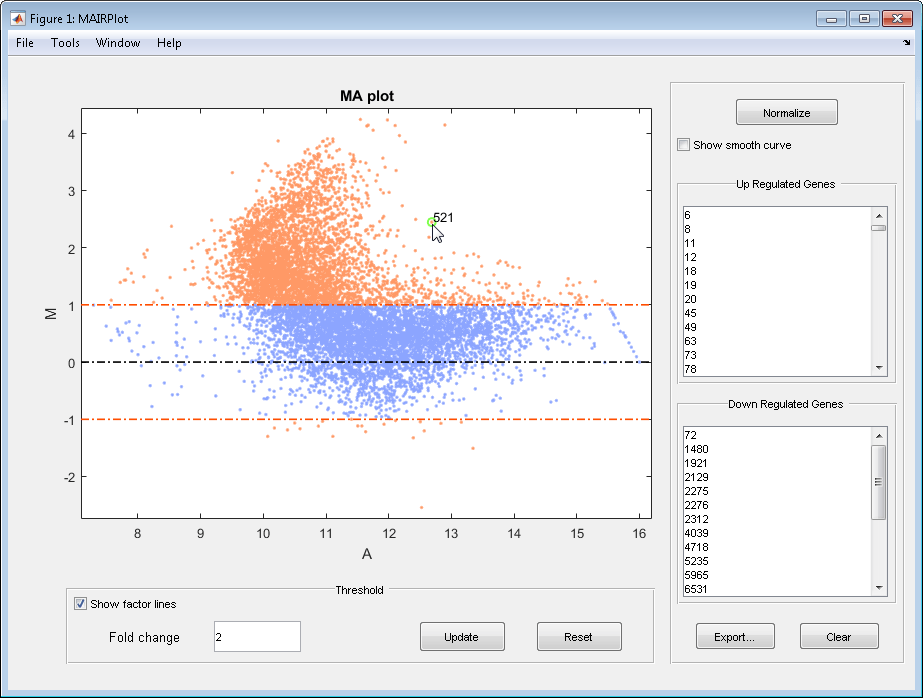
Следующее является графиком MA ненормированных данных.
Интенсивность по сравнению с графиком рассеивания отношения отображает следующее:
log10 (Интенсивность) по сравнению с log2 (Отношение) график рассеивания генов.
Две горизонтали сворачивают линии изменения на уровне изменения сгиба 2, который соответствует отношению 1 и –1 на журнале 2 (Отношение) шкала. (Линии будут на различных уровнях изменения сгиба, если вы использовали 'FactorLines' свойство.
Точки данных для генов, которые рассматриваются дифференцированно описанными (за пределами линий изменения сгиба) появляются в оранжевом.
После того, как вы отобразите интенсивность по сравнению с графиком рассеивания отношения, можно в интерактивном режиме сделать следующее:
Настройте горизонтальные линии изменения сгиба перетаскиванием нажатия кнопки одна линия или ввод значения в текстовом поле Fold Change, затем нажатие по Update.
Отобразите метки для точек данных путем нажатия на точку данных.
Выберите ген из Up Regulated или списка Down Regulated, чтобы подсветить соответствующую точку данных в графике. Нажмите и содержите Ctrl или Shift, чтобы выбрать несколько генов.
Масштабируйте график путем выбора Tools > Zoom In или Tools > Zoom Out.
Просмотрите списки значительно отрегулированных и вниз отрегулированных генов, и опционально, экспортируйте генные метки и индексы к структуре в MATLAB® Рабочая область путем нажатия на Export.
Нормируйте данные путем нажатия кнопки Normalize, затем выбора, показать ли нормированный график в отдельном окне. Если вы показываете нормированный график в отдельном окне, флажок Show smooth curve становится доступным в исходном (ненормированном) графике.
Совет
Чтобы выбрать различные lowess опции нормализации перед нормализацией, выберите Tools > Set LOWESS Normalization Options, затем введите опции в диалоговое окно Options for LOWESS.
Используйте gprread функция, чтобы создать структуру, содержащую микроданные массива.
maStruct = gprread('mouse_a1wt.gpr');Используйте magetfield функционируйте, чтобы извлечь зеленый (cy3) и красный (cy5) сигналы от структуры.
cy5data = magetfield(maStruct,'F635 Median'); cy3data = magetfield(maStruct,'F532 Median');
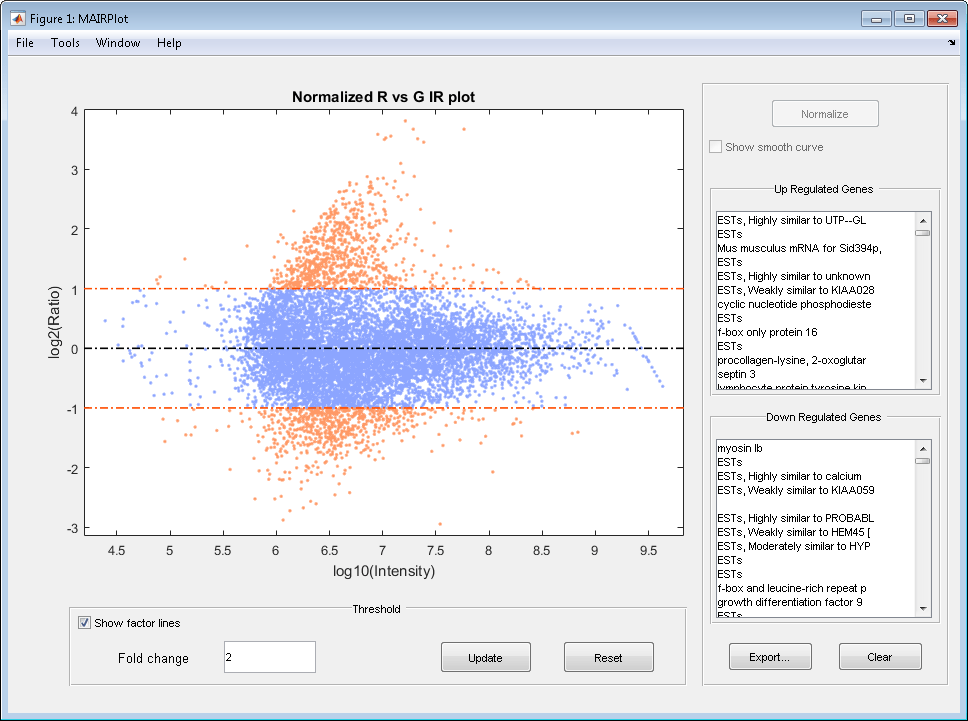
Создайте интенсивность по сравнению с графиком рассеивания отношения cy3 и cy5 данных. Нормируйте данные и добавьте заголовок и метки:
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Title','Normalized R vs G IR plot', ... 'Labels', maStruct.Names)
Возвратите значения интенсивности и отношения, не отображая график.
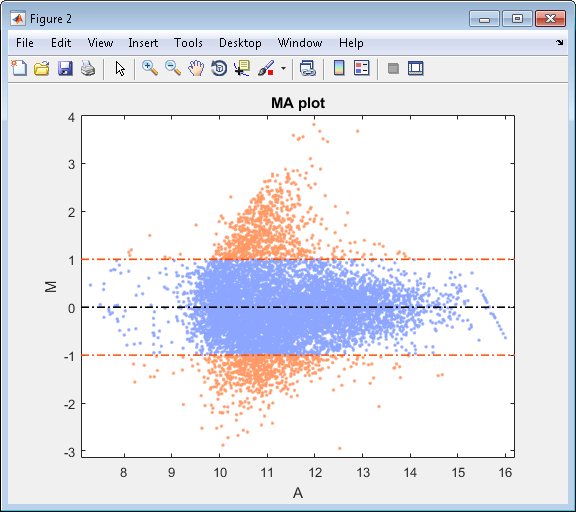
[intensities, ratios] = mairplot(cy5data, cy3data, 'Showplot', false);Создайте нормированный график MA cy3 и cy5 данных без компонентов пользовательского интерфейса.
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Type','MA','PlotOnly',true)
[1] Квакенбуш, J. (2002). Нормализация микроданных массива и преобразование. Генетика природы Suppl. 32, 496–501.
[2] Dudoit, S., Ян, Y.H., Затопляемый, M.J., и Скорость, T.P. (2002). Статистические методы для Идентификации Дифференцированно Описанных Генов в Реплицированных Экспериментах комплементарной ДНК Микромассивов. Statistica Sinica 12, 111–139.
maboxplot | magetfield | maimage | mainvarsetnorm | maloglog | malowess | manorm | mattest | mavolcanoplot