Приложение Genomics Viewer позволяет вам просмотреть и исследовать интегрированные геномные данные со встроенной версией Integrative Genomics Viewer (IGV) [1][2]. Геномные данные включают выравнивания чтения NGS, варианты генома и сегментированные данные о номере копии.
Первая часть этого примера дает краткий обзор приложения и поддерживаемых форматов файлов. Вторая часть примера исследует одно изменение нуклеотида цитохрома p450 ген (CYP2C19).
В командной строке ввести genomicsViewer. В качестве альтернативы кликните по значку приложения на вкладке Apps. Приложение требует интернет-соединения.
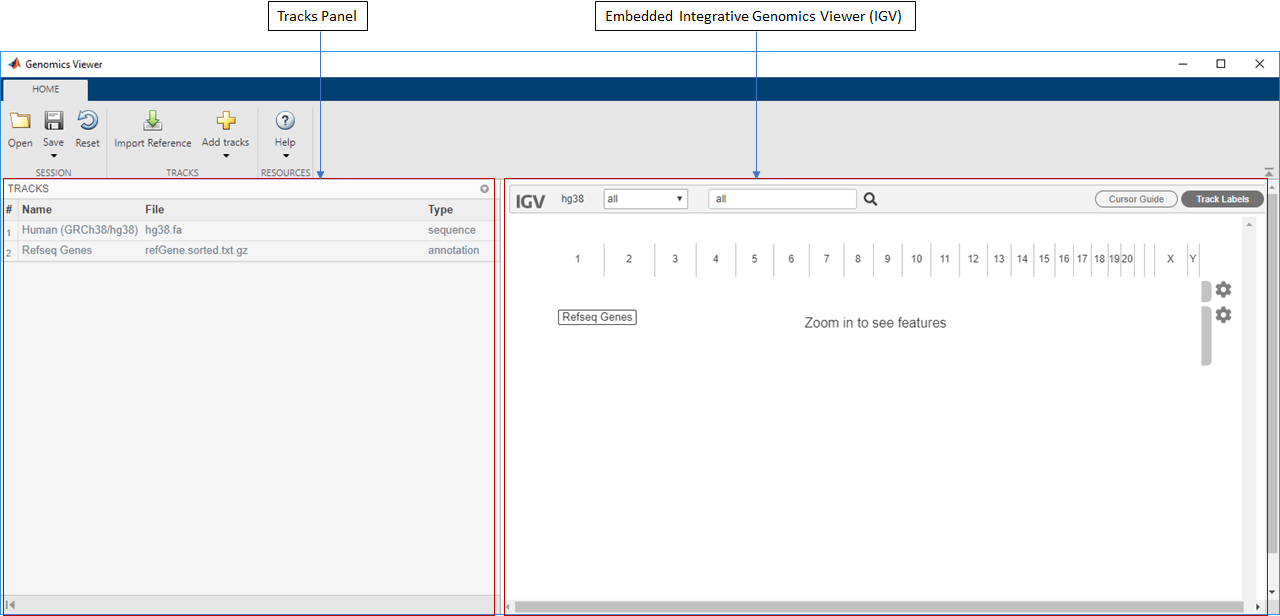
По умолчанию, Человек загрузок приложения (GRCh38/hg38) как ссылочная последовательность и Гены Refseq как файл аннотации. В приложении существует две основных панели. Левая панель является панелью Tracks, и правильная панель является встроенным веб-приложением IGV. Панель Tracks является областью только для чтения, отображающей имена дорожки, имена исходного файла и типы дорожки. Панель Tracks обновляется соответственно, когда вы конфигурируете дорожки во встроенном приложении IGV.
Кнопка Reset восстанавливает приложение к представлению по умолчанию с двумя дорожками (HG38 с Генами Refseq) и удаляет любые другие существующие дорожки. Перед сбросом можно сохранить текущее представление как сеанс (.json) файл и восстановление это позже.
Можно импортировать одну ссылочную последовательность. Ссылочная последовательность должна быть в файле FASTA. Выберите Import Reference на вкладке Home. Можно также импортировать соответствующий cytoband файл, который содержит цитогенетические данные G-соединения. Можно добавить локальные файлы или задать внешние URL. URL Должен запуститься или с https или с gs. Другие протоколы передачи файлов, такие как ftp, не поддерживаются.
Можно импортировать несколько наборов данных считанных данных о выравнивании последовательности. Данные о выравнивании должны быть файл CRAM или BAM. Не требуется, что у вас есть соответствующий индексный файл (.BAI или .CRAI) в том же месте как ваш BAM или файл CRAM. Однако отсутствие индексного файла сделает приложение медленнее.
Можно добавить файлы выравнивания чтения с помощью Add tracks from file и опций Add tracks from URL от кнопки Add tracks. Если вы задаете URL, URL должен запуститься или с https или с gs. Другие протоколы передачи файлов, такие как ftp, не поддерживаются.
Можно импортировать несколько наборов аннотаций функции из нескольких файлов, которые содержат данные для одной ссылочной последовательности. Поддерживаемые файлы аннотации: .BED, .GFF, .GFF3, и .GTF.
Можно также импортировать структурные варианты (.VCF) и визуализировать генетические изменения, такие как вставки и удаления.
Можно просмотреть сегментированные данные о номере копии (.SEG) и количественные геномные данные (.WIG, .BIGWIG, и .BEDGRAPH), такие как peaks ChIP и покрытие выравнивания.
Можно добавить аннотацию и геномные файлы данных с помощью Add tracks from file и опций Add tracks from URL от кнопки Add tracks. Если вы задаете URL, URL должен запуститься или с https или с gs. Другие протоколы передачи файлов, такие как FTP, не поддерживаются.
Ген CYP2C19 является членом цитохрома семейство генов P450. Ферменты, произведенные из цитохрома гены P450, вовлечены в метаболизм различных молекул и химикатов в ячейках. Фермент CYP2C19 играет роль в усваивании по крайней мере 10 процентов обычно прописываемых лекарств [3]. Полиморфизмы в цитохроме p450 семейство могут вызвать неблагоприятные ответы препарата в индивидуумах. Одним примером одного изменения нуклеотида является rs4986893 в положении chr10:94,780,653 где G заменяется A. Этот аллельный вариант также известен как CYP2C19*3. Следующие шаги показывают, как визуализировать такое изменение приложения с помощью и низкого покрытия и высоких данных о покрытии.
В целях этого примера начните с файла сеанса, который имеет некоторые предварительно загруженные дорожки. Чтобы загрузить файл, нажмите Open. Перейдите к matlabroot\examples\bioinfo\, где matlabroot является папкой, где вы установили MATLAB®. Выберите rs4986893.json.
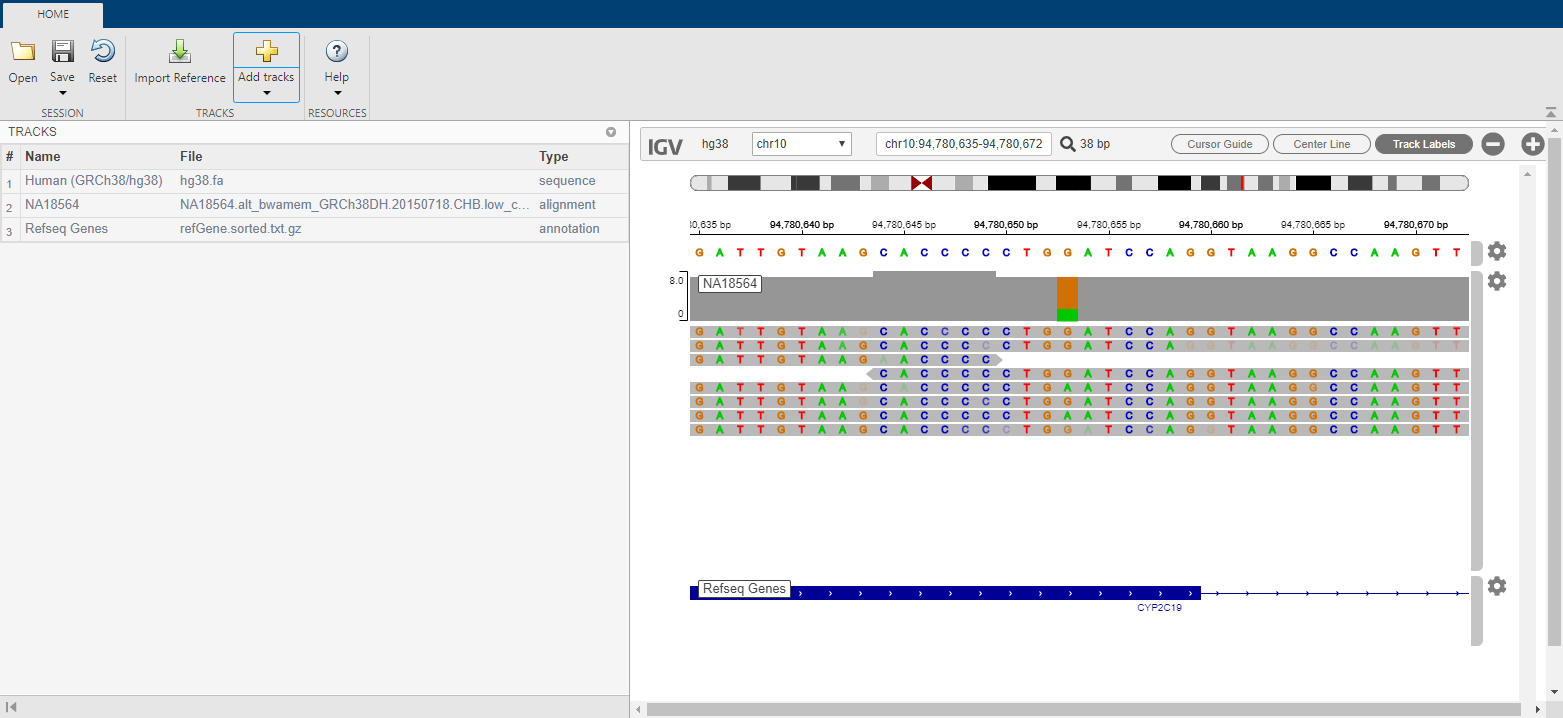
Сеанс содержит три дорожки:
Human (GRCh38/hg38) как ссылка
NA18564 как низкие данные о выравнивании покрытия
Гены Refseq
Низкие данные о выравнивании покрытия прибывают из розеточные ханьцы из Пекина, Китай. Демонстрационным ID является NA18564, и выборка была идентифицирована с мутацией CYP2C19*3 [4].
В этом файле сеанса данные о выравнивании были сосредоточены вокруг местоположения мутации на гене CYP2C19.
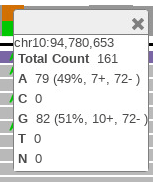
Кликните по оранжевой панели в зоне охвата, чтобы посмотреть на положение и информацию о распределении аллели.
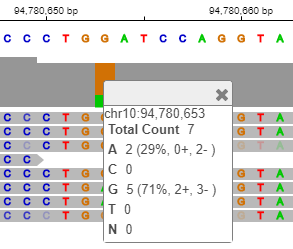
Это показывает, что 71% чтений имеет G, в то время как 29% имеют в местоположении chr10:94,780,653. Эти данные являются низкими данными о покрытии и не могут показать все случаи этой мутации. Высокие данные о покрытии будут исследованы позже в примере.
Закройте окно всплывающей подсказки.
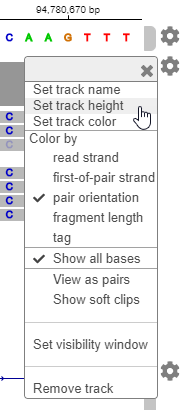
Можно настроить различные аспекты отображения данных в приложении. Например, можно изменить высоту дорожки, чтобы сделать больше комнаты для более поздних дорожек. Кликните по второму значку механизма. Выберите Set track height. Войдите 200.
Для получения дополнительной информации на встроенном приложении IGV и его доступных параметрах, посетите здесь.
Можно посмотреть на высокие данные о покрытии из той же выборки, чтобы видеть случаи этой мутации.
Перейдите к Международному Демонстрационному веб-сайту Ресурса Генома.
Ищите демонстрационный NA18564.
Загрузите файл выравнивания Exome, который находится в .CRAM формат.
Также загрузите соответствующий индексный файл, который находится в .CRAI формат. Сохраните файл в том же месте как источник .CRAM файл.
Щелкните (+) значок на вкладке Home. Выберите загруженный .CRAM файл и нажимает Open.
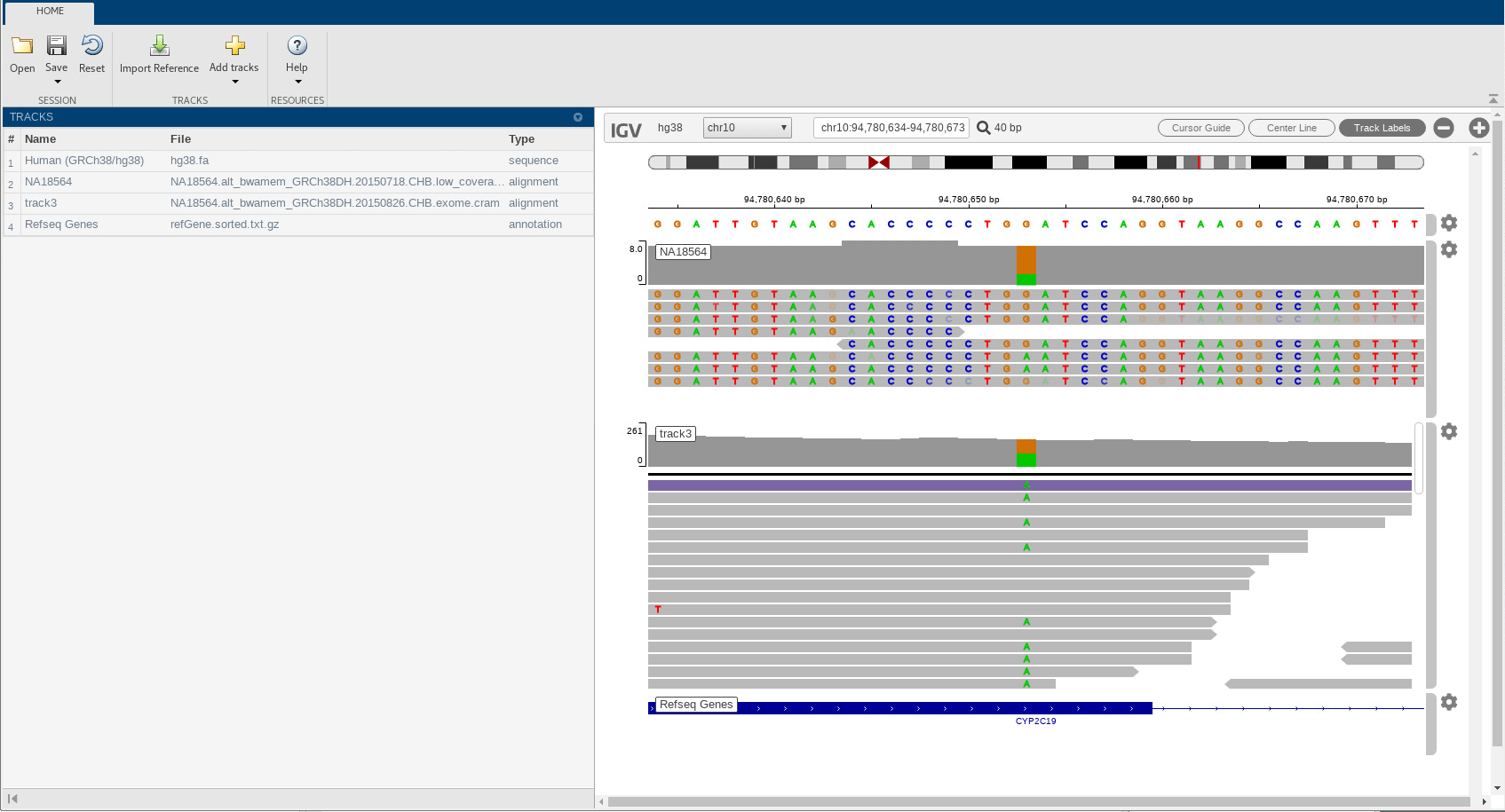
Высокие данные о покрытии появляются как track3. Можно теперь видеть много случаев мутации в нескольких чтениях.
Кликните по оранжевой панели в зоне охвата, чтобы видеть распределение аллели. Это показывает, что G заменяется почти в 50% времени.
[1] Робинсон, J., Х. Торволдсдоттир, В. Винклер, М. Гуттман, E. Высаживающийся на берег, Г. Гетц, Й. Месиров. 2011. Интегральный Genomics Viewer. Биотехнология природы. 29:24–26.
[2] Thorvaldsdóttir, H., Дж. Робинсон, Й. Месиров. 2013. Интегральный Genomics Viewer (IGV): высокоэффективная визуализация данных о геномике и исследование. Брифинги в Биоинформатике. 14:178–192.