Класс: geneont
Найдите условия, которые являются потомками заданного понятия Генной онтологии (GO)
DescendantIDs = getdescendants(GeneontObj, ID)
[DescendantIDs, Counts]
= getdescendants(GeneontObj, ID)
... = getdescendants(..., 'Depth', DepthValue,
...)
... = getdescendants(..., 'Relationtype', RelationtypeValue,
...)
... = getdescendants(..., 'Exclude', ExcludeValue,
...)
DescendantIDs = getdescendants(GeneontObj, ID)GeneontObj поисковых запросов, объект geneont, для ИДУТ условия, которые являются потомками ПОЙТИ термина (терминов), заданного ID, который является ПОЙТИ идентификатором термина или вектором идентификаторов. Это возвращает DescendantIDs, вектор ИДУТ идентификаторы термина включая ID. ID является неотрицательным целым числом или вектором, содержащим неотрицательные целые числа.
[ также возвращает число раз, которым найден каждый потомок. DescendantIDs, Counts]
= getdescendants(GeneontObj, ID)Counts является вектор-столбцом с тем же числом элементов как условия в GeneontObj.
Возвращаемое значение Counts полезно, когда вы соответствуете количествам в генных исследованиях обогащения. Для получения дополнительной информации смотрите Генное Обогащение Онтологии в Микроданных массива.
... = getdescendants(..., 'PropertyName', PropertyValue, ...) getdescendants с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
... = getdescendants(..., 'Depth', поисковые запросы вниз через конкретное количество уровней, DepthValue,
...)DepthValue, в генной онтологии. DepthValue является положительным целым числом. Значением по умолчанию является Inf.
... = getdescendants(..., 'Relationtype', поиски заданных типов связей, RelationtypeValue,
...)RelationtypeValue, в генной онтологии. RelationtypeValue является вектором символов. Выбором является 'is_a', 'part_of' или 'both' (значение по умолчанию).
... = getdescendants(..., 'Exclude', средства управления, исключая ExcludeValue,
...)ID, исходный запрошенный термин (термины), от вывода DescendantIDs, если термин не был найден при поиске генной онтологии. Выбором является true или false (значение по умолчанию).
GeneontObj | Объект geneont, такой, как создано функцией конструктора geneont. |
ID | ПОЙДИТЕ идентификатор термина или вектор идентификаторов. |
DepthValue | Положительное целое число, задающее количество уровней, чтобы искать вниз в генной онтологии. |
RelationtypeValue | Вектор символов, задающий типы связей, чтобы искать в генной онтологии. Выбор:
|
ExcludeValue | Средства управления, исключая ID, исходный запрошенный термин (термины), от вывода DescendantIDs, если термин не был достигнут при поиске генной онтологии. Выбором является true или false (значение по умолчанию). |
DescendantIDs | Вектор ИДЕТ идентификаторы термина включая ID. |
Counts | Вектор-столбец с тем же числом элементов как условия в GeneontObj, указывая на число раз каждый потомок найден. |
Загрузите текущую версию базы данных Gene Ontology с сети в объект geneont в MATLAB.
GO = geneont('LIVE', true)Программное обеспечение MATLAB создает объект geneont и отображает количество условий в базе данных.
Gene Ontology object with 27827 Terms.
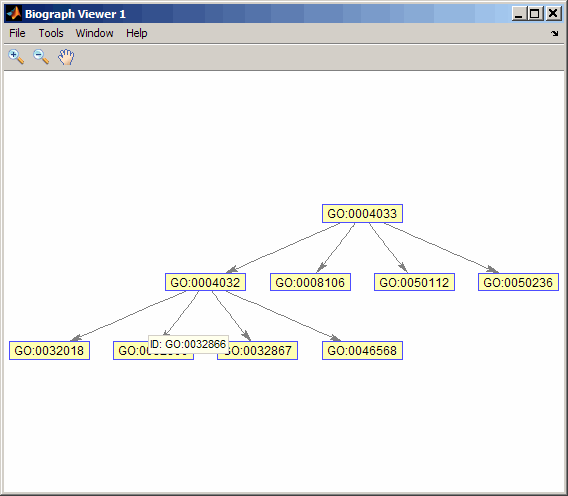
Получите потомков "aldo-keto, действие редуктазы" ИДУТ термин с ПОЙТИ идентификатором 4033.
descendants = getdescendants(GO,4033)
descendants =
4032
4033
8106
32018
32866
32867
46568
50112
50236Создайте зависимую Генную Онтологию.
subontology = GO(descendants) Gene Ontology object with 9 Terms.
Создайте и отобразите отчет зависимых Генных условий Онтологии, который включает ПОЙТИ идентификатор и имя.
rpt = [num2goid(cell2mat(get(subontology.terms,'id')))...
get(subontology.terms,'name')]';
disp(sprintf('%s --> %s \n',rpt{:}))
GO:0004032 --> aldehyde reductase activity
GO:0004033 --> aldo-keto reductase activity
GO:0008106 --> alcohol dehydrogenase (NADP+) activity
GO:0032018 --> 2-methylbutanal reductase activity
GO:0032866 --> xylose reductase activity
GO:0032867 --> arabinose reductase activity
GO:0046568 --> 3-methylbutanal reductase activity
GO:0050112 --> inositol 2-dehydrogenase activity
GO:0050236 --> pyridoxine 4-dehydrogenase activity Просмотрите отношения зависимой Генной Онтологии при помощи метода getmatrix, чтобы создать матрицу связи, чтобы передать функции biograph.
cm = getmatrix(subontology); BG = biograph(cm,rpt(1,:)); view(BG)
goannotread | num2goid | term