Считайте данные о масс-спектрометрии из файла NetCDF
mzCDFStruct = mzcdfread(File)
mzCDFStruct = mzcdfread(File,
...'TimeRange', TimeRangeValue, ...)
mzCDFStruct = mzcdfread(File,
...'ScanIndices', ScanIndicesValue, ...)
mzCDFStruct = mzcdfread(File,
...'Verbose', VerboseValue, ...)
File | Вектор символов или строка, содержащая имя файла, или путь и имя файла, файла NetCDF, который содержит данные о масс-спектрометрии и соответствует ANDI/MS или ASTM E2077-00 (2005) стандартная спецификация или более ранние спецификации. Если вы задаете только имя файла, тот файл должен быть на пути поиска файлов MATLAB® или в текущей папке. |
TimeRangeValue | Двухэлементный СоветЕдиницы измерения времени обозначаются в глобальных атрибутах NetCDF. Для итоговой информации об областях значений времени в файле NetCDF используйте функцию ПримечаниеЕсли вы задаете |
ScanIndicesValue | Положительное целое число, вектор целых чисел или двухэлементный числовой массив СоветДля получения информации об индексах сканирования в файле NetCDF проверяйте поле ПримечаниеЕсли вы задаете |
VerboseValue | Управляет отображением прогресса чтения |
mzCDFStruct | Структура MATLAB, содержащая информацию о масс-спектрометрии из файла NetCDF. Его поля соответствуют переменным и глобальным атрибутам в файле NetCDF. Если переменная NetCDF содержит локальные атрибуты, дополнительное поле создается с именем поля, являющегося именем переменной, добавленным с |
mzCDFStruct = mzcdfread(File)File, и затем создает структуру MATLAB, mzCDFStruct.
File является вектором символов или строкой, содержащей имя файла, или путь и имя файла, файла NetCDF, который содержит данные о масс-спектрометрии. Файл должен соответствовать ANDI/MS или ASTM E2077-00 (2005) стандартная спецификация или более ранние спецификации.
mzCDFStruct содержит поля, которые соответствуют переменным и глобальным атрибутам в файле NetCDF. Если переменная NetCDF содержит локальные атрибуты, дополнительное поле создается с именем поля, являющегося именем переменной, добавленным с _attributes. Номер и имена полей будут отличаться, в зависимости от массового программного обеспечения спектрометра, но обычно существуют поля mass_values и intensity_values.
Анализ данных LC/MS требует расширенных объемов памяти от операционной системы.
Если вы получаете ошибки, связанные с памятью, попробуйте следующее:
Увеличьте виртуальную память (область подкачки) для вашей операционной системы, как описано в Твердости “Из Памяти” Ошибки (MATLAB).
Если вы получаете ошибки, связанные с пространством "кучи" Java®, увеличиваете ваше пространство "кучи" Java:
Если у вас есть версия 7.10 (R2010a) MATLAB или позже, смотрите Настройки Java Heap Memory (MATLAB).
Если у вас есть версия 7.9 (R2009b) MATLAB или ранее, см. https://www.mathworks.com/support/solutions/en/data/1-18I2C/.
mzCDFStruct = mzcdfread(File, ...'PropertyName', PropertyValue, ...) mzcdfread с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
задает область значений времени в mzCDFStruct = mzcdfread(File,
...'TimeRange', TimeRangeValue, ...)File, чтобы читать. TimeRangeValue является двухэлементным числовым массивом [Start End]. Значение по умолчанию должно считать спектры изо всех случаев [0 Inf].
Единицы измерения времени обозначаются в глобальных атрибутах NetCDF. Для итоговой информации об областях значений времени в файле NetCDF используйте функцию mzcdfinfo.
Если вы задаете TimeRangeValue, вы не можете задать ScanIndicesValue.
mzCDFStruct = mzcdfread(File,
...'ScanIndices', ScanIndicesValue, ...)File, чтобы читать. ScanIndicesValue является положительным целым числом, вектором целых чисел или двухэлементным числовым массивом [Start_Ind End_Ind]. Start_Ind и End_Ind каждый положительные целые числа, указывающие на индекс сканирования. Start_Ind должен быть меньше, чем End_Ind. Значение по умолчанию должно считать все сканирования.
Для получения информации об индексах сканирования в файле NetCDF проверяйте поле NumberOfScans в структуру, возвращенную функцией mzcdfinfo.
Если вы задаете ScanIndicesValue, вы не можете задать TimeRangeValue.
mzCDFStruct = mzcdfread(File,
...'Verbose', VerboseValue, ...)File. Выбором является true (значение по умолчанию) или false.
В следующем примере файл не обеспечивается results.cdf.
Считайте файл NetCDF в программное обеспечение MATLAB как структура.
out = mzcdfread('results.cdf');
Просмотрите второе сканирование в файле NetCDF путем создания отдельных переменных, содержащих интенсивность и m/z значения, и затем строящих эти значения. Добавьте заголовок и x-и поля использования меток оси Y в выходной структуре.
idx1 = out.scan_index(2)+1;
idx2 = out.scan_index(3);
y = out.intensity_values(idx1:idx2);
z = out.mass_values(idx1:idx2);
stem(z,y,'marker','none')
title(sprintf('Time: %f',out.scan_acquisition_time(2)))
xlabel(out.mass_axis_units)
ylabel(out.intensity_axis_units)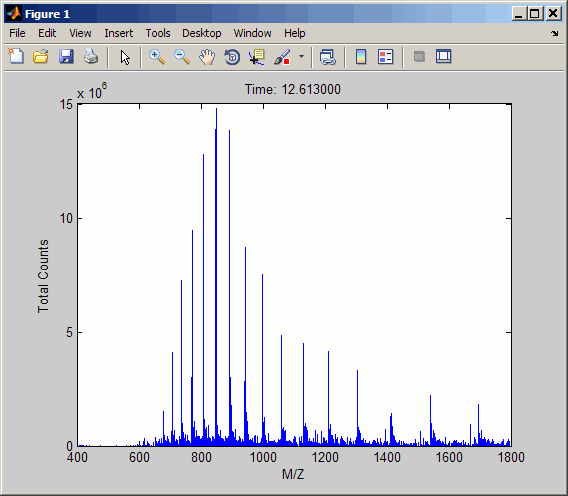
jcampread | mzcdf2peaks | mzcdfinfo | mzxmlread | tgspcread