CellsClosed, ClassesClosedИнтерпретация классов в графиках гистограммы
Блокноты MuPAD® будут демонтированы в будущем релизе. Используйте live скрипты MATLAB® вместо этого.
Live скрипты MATLAB поддерживают большую часть функциональности MuPAD, хотя существуют некоторые различия. Для получения дополнительной информации смотрите, Преобразуют Notebook MuPAD в Live скрипты MATLAB.
CellsClosed | Дополнительный | Left, или Right |
ClassesClosed | [[CellsClosed]] | Смотрите ниже |
| Объекты | Значения по умолчанию |
|---|---|
plot::Histogram2d |
|
CellsClosed определяет, являются ли классы, используемые в гистограмме, intepreted как полуоткрытыми интервалами, которые закрываются слева или правильный контур.
Графический примитивный график:: Histogram2d соответствует числовым данным в ячейки (“классы”), которые заданы атрибутом Cells = [a_1 .. b_1, a_2 .. b_2, dots]. По умолчанию эти классы интерпретированы как набор полуоткрытых интервалов![]() , которые закрываются на правильном контуре. Элемент данных
, которые закрываются на правильном контуре. Элемент данных x соответствуется в i-th ячейку, если она удовлетворяет ai < x ≤ bi. С опцией CellsClosed = Left или эквивалентный ClassesClosed = Left классы интерпретированы как полуоткрытые интервалы![]() , которые закрываются на левом контуре.
, которые закрываются на левом контуре.
Мы создаем выборку 15 точек данных:
data := [1, 2, 2, 3, 3, 3, 4, 4, 4, 4, 5, 5, 5, 5, 5]:
Этим данным нужно соответствовать в следующие ячейки (классы):
cells := [0 .. 1, 1 .. 2, 2 .. 3, 3 .. 4, 4 .. 5, 5 .. 6]:
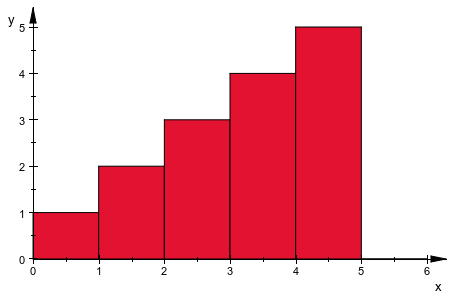
С настройкой по умолчанию CellsClosed = Right, эти 6 классов являются интервалами ![]()
![]()
![]() и т.д. Интервал
и т.д. Интервал![]() содержит один из элементов данных, интервал
содержит один из элементов данных, интервал![]() содержит два и т.д.:
содержит два и т.д.:
plot(plot::Histogram2d(data, Cells = cells))
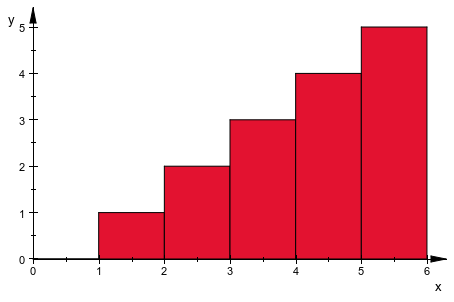
Используя CellsClosed = Left, эти 6 классов интерпретированы как интервалы![]() ,
,![]()
![]() и т.д. Теперь первый класс
и т.д. Теперь первый класс![]() не содержит ни один из элементов данных, второй класс
не содержит ни один из элементов данных, второй класс![]() содержит один элемент и т.д.:
содержит один элемент и т.д.:
plot(plot::Histogram2d(data, Cells = cells, CellsClosed = Left))
delete data, cells: