Отобразите и управляйте 3-D структурой молекулы
molviewer
molviewer(File)
molviewer(pdbID)
molviewer(pdbStruct)
FigureHandle =
molviewer(...)
File | Вектор символов или строка, задающая одно из следующего:
Файл, на который ссылаются, является файлом модели молекулы, таким как Банк данных белка (PDB) - отформатированный файл (текстовый ASCII-файл). Типы правильного файла включают:
|
pdbID | Вектор символов или строка, задающая уникальный идентификатор для структуры белка, записывают в базе данных PDB. ПримечаниеКаждая структура в базе данных PDB представлена алфавитно-цифровым идентификатором с четырьмя символами. Например, |
pdbStruct | Структура, содержащая поле для каждой записи PDB, такой, как возвращено getpdb или pdbread функция. |
FigureHandle | Изобразите указатель на Molecule Viewer. |
molviewer открывает приложение Molecule Viewer. Можно отобразить 3-D молекулярные структуры путем выбора File > Open, File > Load PDB ID или File > Open URL.
molviewer( считывает данные в файле модели молекулы, File)File, и открывает приложение Molecule Viewer, отображающее 3-D молекулярную структуру для просмотра и манипуляции.
molviewer( получает структурные данные белка, pdbID)pdbID, от базы данных PDB и открывает приложение Molecule Viewer, отображающее 3-D молекулярную структуру для просмотра и манипуляции.
molviewer( считывает данные из pdbStruct)pdbStruct, структура, содержащая поле для каждой записи PDB, и, открывает приложение Molecule Viewer, отображающее 3-D молекулярную структуру для просмотра и манипуляции.
FigureHandle =
molviewer(...)
Можно передать FigureHandle к evalrasmolscript функция, которая отправляет команды скрипта RasMol в окно Molecule Viewer.
Если вы получаете какие-либо ошибки, связанные с памятью или пространством "кучи" Java®, попытайтесь увеличить свое пространство "кучи" Java, аналогичное описанному в https://www.mathworks.com/support/solutions/en/data/1-18I2C/.
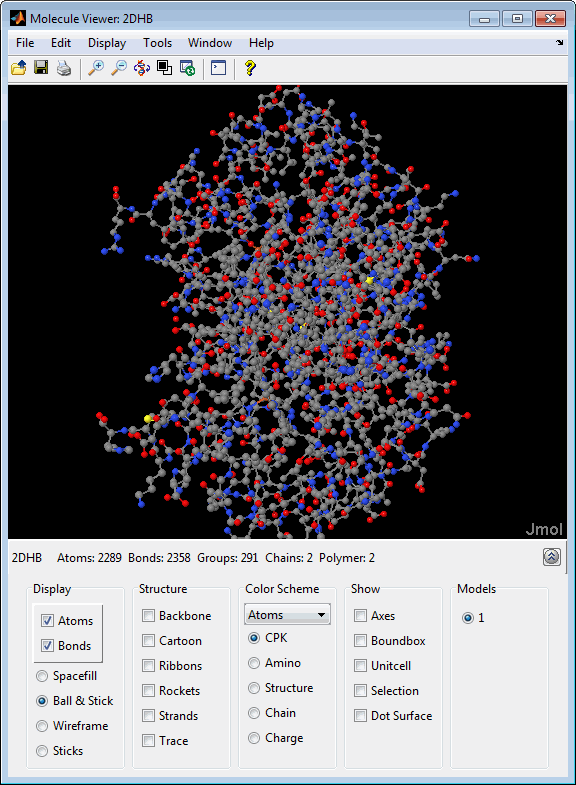
После отображения 3-D структуры молекулы вы можете:
Наведите мышь на субкомпонент молекулы, чтобы отобразить идентификационную метку для него.
Вращайте и вращайте молекулу под различными углами перетаскиванием нажатия кнопки это.
Вращайте молекулу в x-z плоскости путем нажатия![]() .
.
Вращайте молекулу в x-y плоскости путем нажатия и удержания клавиши Shift, затем левое и правое перетаскивание нажатия кнопки.
Увеличение stepless вида путем нажатия и удержания клавиши Shift, затем перетаскивание нажатия кнопки вверх и вниз.
Увеличение пошагового вида путем нажатия на фигуру, затем превращения колесика прокрутки мыши, или путем нажатия следующих кнопок:
![]() или
или ![]()
Переместите молекулу путем нажатия и содержания Ctrl + Alt, затем перетаскивание нажатия кнопки.
Измените цвет фона между черным цветом и белым цветом путем нажатия![]() .
.
Сбросьте положение молекулы путем нажатия![]() .
.
Покажите или скройте Панель управления путем нажатия![]() .
.
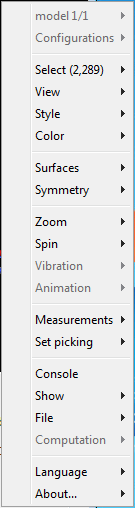
Управляйте и аннотируйте 3-D структуру путем выбора опций в Панели управления или, для полного списка опций, путем щелчка правой кнопкой по окну Molecule Viewer, чтобы выбрать команды:
Отобразите Консоль Скрипта Jmol путем нажатия![]() .
.
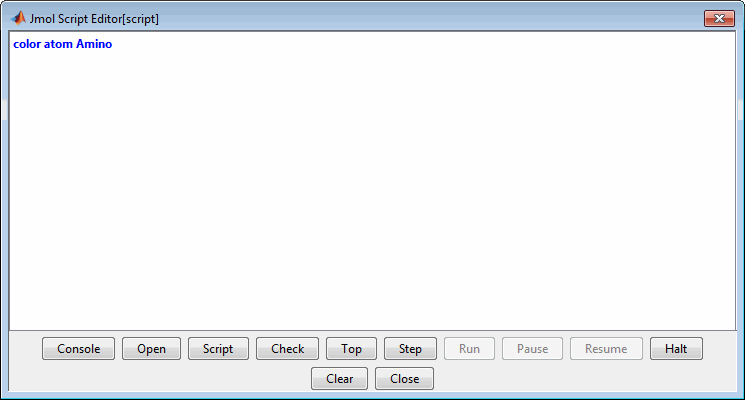
Существует известная ошибка с кнопкой Open редактора сценариев, который предотвращает загрузку скрипта Rasmol в интерактивном режиме. Вместо этого используйте evalrasmolscript функция, которая отправляет команды скрипта RasMol в приложение Molecule Viewer. Кроме того, можно скопировать и вставить команды скрипта в консоль скрипта.
Просмотрите ацетилсалициловую кислоту (аспирин) молекула, структурная информация которой содержится в файле молекулы Elsevier MDL aspirin.mol.
molviewer('aspirin.mol')Просмотрите вирусную молекулу гемагглютинина гриппа H5N1, структурная информация которой расположена в www.rcsb.org/pdb/files/2FK0.pdb.gz.
molviewer('http://www.rcsb.org/pdb/files/2FK0.pdb.gz') Просмотрите молекулу с идентификатором PDB 2DHB.
molviewer('2DHB')Просмотрите молекулу с идентификатором PDB 4hhb, и создайте указатель фигуры для Molecule Viewer.
FH = molviewer('4hhb')Используйте getpdb функция, чтобы получить данные о структуре белка от базы данных PDB и создать структуру MATLAB. Затем просмотрите молекулу белка.
pdbstruct = getpdb('1vqx')
molviewer(pdbstruct) evalrasmolscript | getpdb | pdbread | pdbsuperpose | pdbtransform | pdbwrite