Создайте агломерационные кластеры из данных
T = clusterdata(X,cutoff)X, учитывая порог cutoff для сокращения агломерационного иерархического дерева, что linkage функция генерирует от X.
clusterdata поддерживает агломерационную кластеризацию и включает pdist, linkage, и cluster функции, которые можно использовать отдельно в более детальном анализе. Дополнительную информацию см. в Описании Алгоритма.
T = clusterdata(X,Name,Value)'Maxclust',5 найти максимум пяти кластеров.
Если 'Linkage' 'centroid' или 'median', затем linkage может произвести кластерное дерево, которое не является монотонным. Этот результат происходит, когда расстояние от объединения двух кластеров, r и s, к третьему кластеру меньше расстояния между r и s. В этом случае, в древовидной схеме, чертившей с ориентацией по умолчанию, путь от листа до корневого узла делает некоторые нисходящие шаги. Чтобы избежать этого результата, задайте другое значение для 'Linkage'. Следующее изображение показывает немонотонное кластерное дерево.
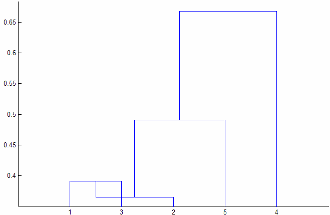
В этом случае к кластеру 1 и кластеру 3 соединяют в новый кластер, в то время как расстояние между этим новым кластером и кластером 2 меньше расстояния между кластером 1 и кластером 3.
Если вы задаете значение c для cutoff входной параметр, затем T = clusterdataXC
Создайте вектор Евклидова расстояния между парами наблюдений в X при помощи pdist.
Y = pdistX, 'евклидов')
Создайте агломерационное иерархическое кластерное дерево из Y при помощи linkage с 'single' метод для вычисления кратчайшего расстояния между кластерами.
Z = linkage(Y, 'single')
Если 0 < c< 2 , используйте cluster задавать кластеры от Z когда противоречивые значения меньше c.
T = cluster(Z, 'Сокращение', c)
Если c целочисленное значение ≥ 2, используйте cluster найти максимум c кластеры от Z.
T = кластер (Z, 'MaxClust', c)
Если у вас есть иерархический кластерный древовидный Z (выход linkage функция для матрицы входных данных X), можно использовать cluster выполнять агломерационную кластеризацию на Z и возвратите кластерное присвоение для каждого наблюдения (строка) в X.
cluster | dendrogram | inconsistent | kmeans | linkage | pdist