Сценарии симуляции
SimBiology.Scenarios объект, который позволяет вам сгенерировать различные сценарии симуляции на основе различных демонстрационных значений количеств модели. Можно объединить эти количества с различными дозами или вариантами и симулировать различные сценарии, чтобы исследовать поведения модели при различных экспериментальных условиях и режимах дозирования.
sObj = SimBiology.Scenarios(name,content)Scenarios объект sObj с одной записью. name имя количества модели или имя группы вариантов или доз для генерации сценария. content содержит соответствующие числовые значения для количества модели или вектора из различных объектов или вектора из объектов дозы.
sObj = SimBiology.Scenarios(quantityNames,probDist,Name,Value)quantityNames от объединенного вероятностного распределения probDist. Задайте дополнительные опции для вероятностных распределений и метода выборки с помощью одного или нескольких аргументов пары "имя-значение". Чтобы задать вероятностные распределения, у вас должен быть Statistics and Machine Learning Toolbox™.
name — Имя записиИмя записи в виде вектора символов или строки.
Можно определить имя записи к имени количества модели (разновидности, параметр или отсек). В качестве альтернативы можно задать имя для группы доз или вариантов, которые будут включены в выборку (сценарии) генерация.
Пример: "k1"
Типы данных: char | string
content — Значения количества модели или вектор из доз или вариантовRepeatDose или ScheduleDose объекты | вектор из различных объектовЗначения количества модели или вектор из доз или вариантов в виде числового вектора, вектора из RepeatDose или ScheduleDose объекты или вектор из variant объекты.
Если вы указываете, что количество называет для name входной параметр, набор content к числовому вектору.
Если вы задаете имя для группы доз или вариантов, устанавливаете content к вектору из объектов дозы или вектору из различных объектов.
Пример: [0.5,1,1.5]
quantityNames — Имена количеств моделиИмена количеств модели для выборки (сценарий) генерация в виде вектора символов, строки, представляют в виде строки вектор или массив ячеек из символьных векторов.
Пример: ["k12","k21"]
Типы данных: char | string | cell
probDist — Вероятностные распределенияВероятностные распределения, чтобы сгенерировать демонстрационные значения для количеств модели в виде вектора из объектов вероятностного распределения, вектора символов, строки, представляют в виде строки вектор или массив ячеек из символьных векторов, содержащий имена поддерживаемых вероятностных распределений. Чтобы задать вероятностные распределения, у вас должен быть Statistics and Machine Learning Toolbox.
Используйте makedist (Statistics and Machine Learning Toolbox) функция, чтобы создать объекты распределения. Для списка поддерживаемых распределений см. distname (Statistics and Machine Learning Toolbox).
Пример: [pd1,pd2]
Задайте дополнительные разделенные запятой пары Name,Value аргументы. Name имя аргумента и Value соответствующее значение. Name должен появиться в кавычках. Вы можете задать несколько аргументов в виде пар имен и значений в любом порядке, например: Name1, Value1, ..., NameN, ValueN.
'Number',10 задает, чтобы сгенерировать 10 выборок.'Number' — Количество выборок[] (значение по умолчанию) | положительная скалярная величинаКоличество выборок, чтобы чертить от вероятностных распределений в виде разделенной запятой пары, состоящей из 'Number' и положительная скалярная величина. Значение по умолчанию [] средние значения, что функция выводит количество выборок от других записей. Если номер не может быть выведен, номер определяется к 2.
Пример: 'Number',5
'RankCorrelation' — Матрица порядковой корреляции[] (значение по умолчанию) | числовая матрицаМатрица порядковой корреляции для объединенного вероятностного распределения в виде разделенной запятой пары, состоящей из 'RankCorrelation' и числовая матрица. Поведение по умолчанию - это когда оба 'RankCorrelation' и 'Covariance' установлены в [], SimBiology.Scenarios чертит некоррелированые выборки от объединенного вероятностного распределения.
Вы не можете задать 'RankCorrelation' если 'Covariance' установлен. Количество столбцов в матрице должно совпадать с количеством заданных распределений. Матрица должна быть симметричной с диагональными значениями 1. Все его собственные значения должны также быть положительными.
Пример: 'RankCorrelation',[1 0.3;0.3 1]
'Mean' — Средние значенияСредние значения количеств в виде разделенной запятой пары, состоящей из 'Mean' и числовой вектор.
Можно задать средние значения для нормальных распределений только. Количество средних значений должно равняться количеству заданных вероятностных распределений.
Пример: 'Mean',[0.5,1.5]
'Covariance' — Ковариационная матрица[] (значение по умолчанию) | числовая матрицаКовариационная матрица для объединенного вероятностного распределения в виде разделенной запятой пары, состоящей из 'Covariance' и числовая матрица. Поведение по умолчанию - это если оба 'RankCorrelation' и 'Covariance' установлены в [], SimBiology.Scenarios чертит некоррелированые выборки от объединенного вероятностного распределения. Вы не можете задать 'Covariance' если вы задаете 'RankCorrelation'.
Можно задать ковариационную матрицу для нормальных распределений только. Количество столбцов в матрице должно совпадать с количеством заданных распределений. Все его собственные значения должны также быть неотрицательными.
Пример: 'Covariance',[0.25 0.15;0.15 0.25]
'SamplingMethod' — Выборка метода'random' (значение по умолчанию) | 'lhs' | 'copula' | 'sobol' | 'halton'Выборка метода в виде разделенной запятой пары, состоящей из 'SamplingMethod' и вектор символов или строка. В зависимости от ли вероятностные распределения с 'RankCorrelation' или нормальные распределения с 'Covariance' заданы, методы выборки отличаются.
Если запись содержит (объединенное) нормальное распределение с Covariance заданный, методы выборки:
'random' – Чертите случайные выборки от заданного использования нормального распределения mvnrnd (Statistics and Machine Learning Toolbox).
'lhs' – Чертите латинские выборки гиперкуба от заданного использования нормальных распределений lhsnorm (Statistics and Machine Learning Toolbox). Для получения дополнительной информации смотрите Генерирующиеся Квазислучайные числа (Statistics and Machine Learning Toolbox).
Если запись содержит (объединенное) распределение без Covariance заданный, методы выборки:
'random' – Чертите случайные выборки от заданного использования вероятностных распределений random (Statistics and Machine Learning Toolbox).
'lhs' – Чертите латинские выборки гиперкуба от заданных вероятностных распределений с помощью алгоритма, похожего на lhsdesign (Statistics and Machine Learning Toolbox). Этот подход является более систематическим заполняющим пространство подходом, чем случайная выборка. Для получения дополнительной информации смотрите Генерирующиеся Квазислучайные числа (Statistics and Machine Learning Toolbox).
'copula' – Чертите случайные выборки с помощью связки (Statistics and Machine Learning Toolbox). Используйте эту опцию, чтобы наложить корреляции между выборками с помощью связок.
'sobol' – Используйте sobol последовательность (sobolset (Statistics and Machine Learning Toolbox)), который преобразовывается с помощью обратной кумулятивной функции распределения (icdf (Statistics and Machine Learning Toolbox)) заданных вероятностных распределений. Используйте этот метод для очень систематического заполнения. Для получения дополнительной информации смотрите Генерирующиеся Квазислучайные числа (Statistics and Machine Learning Toolbox).
'halton' – Используйте последовательность Холтона (haltonset (Statistics and Machine Learning Toolbox)), который преобразовывается с помощью обратной кумулятивной функции распределения (icdf (Statistics and Machine Learning Toolbox)) заданных вероятностных распределений. Для получения дополнительной информации смотрите Генерирующиеся Квазислучайные числа (Statistics and Machine Learning Toolbox).
Если никакой Covariance задан, SimBiology.Scenarios по существу выполняет два шага. Первый шаг должен сгенерировать выборки с помощью одного из вышеупомянутых методов выборки. Для lhs, sobol, и halton методы, сгенерированные универсальные выборки преобразовываются к выборкам от заданного распределения с помощью обратной кумулятивной функции распределения icdf (Statistics and Machine Learning Toolbox). Затем как второй шаг, выборки коррелируются с помощью алгоритма Iman-Коновера если RankCorrelation задан. Для random, выборки чертятся непосредственно от заданных распределений, и выборки затем коррелируются с помощью алгоритма Iman-Коновера.
Пример: 'SamplingMethod','lhs'
Expression — Выражение комбинацииЭто свойство доступно только для чтения.
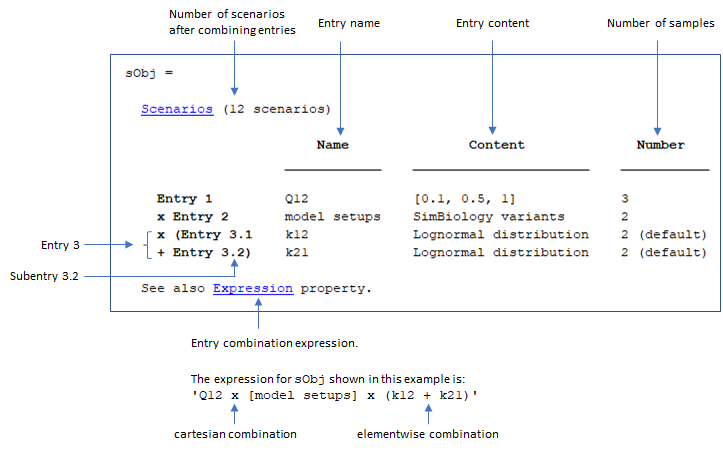
Выражение комбинации, обобщающее комбинацию записей в объекте в виде вектора символов. Плюс + знак указывает на поэлементную комбинацию и перекрестный x знак указывает на декартову комбинацию. Для получения дополнительной информации см. Сценарии Симуляции Объединения в SimBiology.
Пример:
'(k1 + k2 + k3) x doses'
Типы данных: char
NumberOfEntries — Количество записейКоличество записей в сценариях возражает в виде положительного целого числа.
Пример 4
Типы данных: double
RandomSeed — Отберите или утвердите для генерации случайных чисел[] (значение по умолчанию) | неотрицательное целое число, меньшее, чем 232 | структура, возвращенная rngОтберите для генерации случайных чисел, чтобы получить восстанавливаемые сценарии в виде неотрицательного целого числа, меньшего, чем 232 или структура, возвращенная rng это задает случайное состояние. Значение по умолчанию [] средние значения, что сгенерированные сценарии будут отличаться каждый раз generate функция вызвана, если вы не устанавливаете случайный seed прежде, чем вызвать функцию или используете восстанавливаемые последовательности, такие как Sobol или Холтон.
Пример:
10
Типы данных: double | struct
add | Добавьте значения количества, дозы или варианты к SimBiology.Scenarios объект |
getEntry | Получите содержимое записи от SimBiology.Scenarios объект |
updateEntry | Обновите содержимое записи от SimBiology.Scenarios объект |
rename | Переименуйте запись от SimBiology.Scenarios объект |
remove | Удалите записи из SimBiology.Scenarios объект |
verify | Проверьте SimBiology.Scenarios объект |
generate | Сгенерируйте сценарии от SimBiology.Scenarios возразите и возвратите таблицу |
getNumberScenarios | Возвратите количество сценариев от SimBiology.Scenarios объект |
Загрузите модель ответа инсулина глюкозы. Для получения дополнительной информации о модели, смотрите раздел Background в Симуляции Ответа Инсулина Глюкозы.
sbioloadproject('insulindemo','m1');
Модель содержит различные значения параметров и начальные условия, который представляет различные нарушения инсулина (такие как диабет 2 типа, низкая чувствительность инсулина, и так далее) сохраненный в пяти вариантах.
variants = getvariant(m1)
variants = SimBiology Variant Array Index: Name: Active: 1 Type 2 diabetic false 2 Low insulin se... false 3 High beta cell... false 4 Low beta cell ... false 5 High insulin s... false
Подавите информационное предупреждение, которое выдано во время симуляций.
warnSettings = warning('off','SimBiology:DimAnalysisNotDone_MatlabFcn_Dimensionless');
Выберите дозу, которая представляет одну еду 78 граммов глюкозы.
singleMeal = sbioselect(m1,'Name','Single Meal');
Создайте Scenarios объект представлять различные начальные условия, объединенные дозой. Таким образом, создайте scenario возразите, где каждый вариант соединен (или объединен) с дозой, для в общей сложности пяти сценариев симуляции.
sObj = SimBiology.Scenarios; add(sObj,'cartesian','variants',variants); add(sObj,'cartesian','dose',singleMeal)
ans =
Scenarios (5 scenarios)
Name Content Number
________ ___________________ ______
Entry 1 variants SimBiology variants 5
x Entry 2 dose SimBiology dose 1
See also Expression property.
sObj содержит две записи. Используйте generate функционируйте, чтобы объединить записи и сгенерировать пять сценариев. Функция возвращает таблицу сценариев, где каждая строка представляет сценарий, и каждый столбец представляет запись Scenarios объект.
scenariosTbl = generate(sObj)
scenariosTbl=5×2 table
variants dose
________________________ ___________________________
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
Поменяйте имя записи первой записи.
rename(sObj,1,'Insulin Impairements')ans =
Scenarios (5 scenarios)
Name Content Number
____________________ ___________________ ______
Entry 1 Insulin Impairements SimBiology variants 5
x Entry 2 dose SimBiology dose 1
See also Expression property.
Создайте SimFunction объект симулировать сгенерированные сценарии. Используйте Scenarios возразите как вход и задайте плазменные концентрации глюкозы и инсулина как reponses (выходные параметры функции, которая будет построена). Задайте [] для входного параметра дозы начиная с Scenarios объект уже имеет информацию о дозировании.
f = createSimFunction(m1,sObj,{'[Plasma Glu Conc]','[Plasma Ins Conc]'},[])f =
SimFunction
Parameters:
Name Value Type Units
____________________________ ______ _____________ ___________________________________________
{'Plasma Volume (Glu)' } 1.88 {'parameter'} {'deciliter' }
{'k1' } 0.065 {'parameter'} {'1/minute' }
{'k2' } 0.079 {'parameter'} {'1/minute' }
{'Plasma Volume (Ins)' } 0.05 {'parameter'} {'liter' }
{'m1' } 0.19 {'parameter'} {'1/minute' }
{'m2' } 0.484 {'parameter'} {'1/minute' }
{'m4' } 0.1936 {'parameter'} {'1/minute' }
{'m5' } 0.0304 {'parameter'} {'minute/picomole' }
{'m6' } 0.6469 {'parameter'} {'dimensionless' }
{'Hepatic Extraction' } 0.6 {'parameter'} {'dimensionless' }
{'kmax' } 0.0558 {'parameter'} {'1/minute' }
{'kmin' } 0.008 {'parameter'} {'1/minute' }
{'kabs' } 0.0568 {'parameter'} {'1/minute' }
{'kgri' } 0 {'parameter'} {'1/minute' }
{'f' } 0.9 {'parameter'} {'dimensionless' }
{'a' } 0 {'parameter'} {'1/milligram' }
{'b' } 0.82 {'parameter'} {'dimensionless' }
{'c' } 0 {'parameter'} {'1/milligram' }
{'d' } 0.01 {'parameter'} {'dimensionless' }
{'Stomach Glu After Dosing'} 78 {'parameter'} {'gram' }
{'kp1' } 2.7 {'parameter'} {'milligram/minute' }
{'kp2' } 0.0021 {'parameter'} {'1/minute' }
{'kp3' } 0.009 {'parameter'} {'(milligram/minute)/(picomole/liter)' }
{'kp4' } 0.0618 {'parameter'} {'(milligram/minute)/picomole' }
{'ki' } 0.0079 {'parameter'} {'1/minute' }
{'[Ins Ind Glu Util]' } 1 {'parameter'} {'milligram/minute' }
{'Vm0' } 2.5129 {'parameter'} {'milligram/minute' }
{'Vmx' } 0.047 {'parameter'} {'(milligram/minute)/(picomole/liter)' }
{'Km' } 225.59 {'parameter'} {'milligram' }
{'p2U' } 0.0331 {'parameter'} {'1/minute' }
{'K' } 2.28 {'parameter'} {'picomole/(milligram/deciliter)' }
{'alpha' } 0.05 {'parameter'} {'1/minute' }
{'beta' } 0.11 {'parameter'} {'(picomole/minute)/(milligram/deciliter)'}
{'gamma' } 0.5 {'parameter'} {'1/minute' }
{'ke1' } 0.0005 {'parameter'} {'1/minute' }
{'ke2' } 339 {'parameter'} {'milligram' }
{'Basal Plasma Glu Conc' } 91.76 {'parameter'} {'milligram/deciliter' }
{'Basal Plasma Ins Conc' } 25.49 {'parameter'} {'picomole/liter' }
Observables:
Name Type Units
_____________________ ___________ _______________________
{'[Plasma Glu Conc]'} {'species'} {'milligram/deciliter'}
{'[Plasma Ins Conc]'} {'species'} {'picomole/liter' }
Dosed:
TargetName TargetDimension
__________ _____________________
{'Dose'} {'Mass (e.g., gram)'}
Симулируйте модель в течение 24 часов и постройте данные моделирования. Данные содержат пять запусков, где каждый запуск представляет сценарий в объекте Scenarios.
sd = f(sObj,24); sbioplot(sd)

ans =
Axes (SbioPlot) with properties:
XLim: [0 25]
YLim: [0 450]
XScale: 'linear'
YScale: 'linear'
GridLineStyle: '-'
Position: [0.0744 0.1100 0.3901 0.8150]
Units: 'normalized'
Show all properties
Если у вас есть Statistics and Machine Learning Toolbox™, можно также чертить демонстрационные значения для количеств модели от различных вероятностных распределений. Например, предположите что параметры Vmx и kp3, которые известны низкой и высокой чувствительностью инсулина, следуют за логарифмически нормальным распределением. Можно сгенерировать демонстрационные значения для этих параметров от такого распределения и выполнить скан, чтобы исследовать поведение модели.
Задайте логарифмически нормальный объект вероятностного распределения для Vmx.
pd_Vmx = makedist('lognormal')pd_Vmx =
LognormalDistribution
Lognormal distribution
mu = 0
sigma = 1
По определению, параметр mu среднее значение логарифмических значений. Чтобы варьироваться значение параметров вокруг основного значения (модели) параметра, установите mu к log(model_value). Установите стандартное отклонение на 0,2.
Vmx = sbioselect(m1,'Name','Vmx'); pd_Vmx.mu = log(Vmx.Value); pd_Vmx.sigma = 0.2
pd_Vmx =
LognormalDistribution
Lognormal distribution
mu = -3.05761
sigma = 0.2
Так же задайте вероятностное распределение для kp3.
pd_kp3 = makedist('lognormal'); kp3 = sbioselect(m1,'Name','kp3'); pd_kp3.mu = log(kp3.Value); pd_kp3.sigma = 0.2
pd_kp3 =
LognormalDistribution
Lognormal distribution
mu = -4.71053
sigma = 0.2
Теперь задайте объединенное вероятностное распределение, чтобы чертить демонстрационные значения для Vmx и kp3 с порядковой корреляцией, чтобы задать некоторую корреляцию между этими двумя параметрами. Обратите внимание на то, что это предположение корреляции в целях рисунка этого примера только и не может быть биологически релевантным.
Сначала удалите запись вариантов (запись 1) от sObj.
remove(sObj,1)
ans =
Scenarios (1 scenarios)
Name Content Number
____ _______________ ______
Entry 1 dose SimBiology dose 1
See also Expression property.
Добавьте запись, которая задает объединенное вероятностное распределение с матрицей порядковой корреляции.
add(sObj,'cartesian',["Vmx","kp3"],[pd_Vmx, pd_kp3],'RankCorrelation',[1,0.5;0.5,1])
ans =
Scenarios (2 scenarios)
Name Content Number
____ ______________________ ___________
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 2 (default)
+ Entry 2.2) kp3 Lognormal distribution 2 (default)
See also Expression property.
По умолчанию номер выборок, чтобы чертить от совместного распределения определяется к 2. Увеличьте число выборок.
updateEntry(sObj,2,'Number',100)ans =
Scenarios (100 scenarios)
Name Content Number
____ ______________________ ______
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 100
+ Entry 2.2) kp3 Lognormal distribution 100
See also Expression property.
Проверьте что Scenarios объект может быть симулирован с моделью. verify функционируйте выдает ошибку, если какая-либо запись не решает исключительно к объекту в модели, или содержимое записи имеет противоречивые длины (объемы выборки). Функция выдает предупреждение, если многократные въезды решают к тому же объекту в модели.
verify(sObj,m1)
Сгенерируйте сценарии симуляции. Постройте демонстрационные значения с помощью plotmatrix. Вы видите значение Vmx варьируется вокруг его значения модели 0.047 и тот из kp3 приблизительно 0,009.
sTbl = generate(sObj); [s,ax,bigax,h,hax] = plotmatrix([sTbl.Vmx,sTbl.kp3]); ax(1,1).YLabel.String = "Vmx"; ax(2,1).YLabel.String = "kp3"; ax(2,1).XLabel.String = "Vmx"; ax(2,2).XLabel.String = "kp3";

Симулируйте сценарии с помощью того же SimFunction, который вы создали ранее. Вы не должны создавать новый объект SimFunction даже при том, что объект Scenarios был обновлен.
sd2 = f(sObj,24); sbioplot(sd2);

По умолчанию SimBiology использует случайный метод выборки. Можно изменить его в латинскую выборку гиперкуба (или sobol или Холтон) для более систематического заполняющего пространство подхода.
entry2struct = getEntry(sObj,2)
entry2struct = struct with fields:
Name: {'Vmx' 'kp3'}
Content: [2x1 prob.LognormalDistribution]
Number: 100
RankCorrelation: [2x2 double]
Covariance: []
SamplingMethod: 'random'
entry2struct.SamplingMethod = 'lhs'entry2struct = struct with fields:
Name: {'Vmx' 'kp3'}
Content: [2x1 prob.LognormalDistribution]
Number: 100
RankCorrelation: [2x2 double]
Covariance: []
SamplingMethod: 'lhs'
Можно теперь использовать обновленную структуру, чтобы изменить запись 2.
updateEntry(sObj,2,entry2struct)
ans =
Scenarios (100 scenarios)
Name Content Number
____ ______________________ ______
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 100
+ Entry 2.2) kp3 Lognormal distribution 100
See also Expression property.
Визуализируйте демонстрационные значения.
sTbl2 = generate(sObj); [s,ax,bigax,h,hax] = plotmatrix([sTbl2.Vmx,sTbl2.kp3]); ax(1,1).YLabel.String = "Vmx"; ax(2,1).YLabel.String = "kp3"; ax(2,1).XLabel.String = "Vmx"; ax(2,2).XLabel.String = "kp3";

Симулируйте сценарии.
sd3 = f(sObj,24); sbioplot(sd3);

Восстановите предупреждение настроек.
warning(warnSettings);
SimBiology.Scenarios ТерминологияЭтот раздел аннотирует отображение командной строки SimBiology.Scenarios возразите и объясняет условия, показанные в выходе. А именно, это объясняет эту терминологию: Scenarios, Entry, SubentryИмя, Content, NumberВыражение, inconsistent и Diagnosis.
Сопоставимый
Scenarios объект имеет записи, которые имеют правильное количество выборок так, чтобы записи могли быть объединены без ошибки. Пример сопоставимого Scenarios объект показывают затем.
Противоречивый объект Scenarios имеет одну или несколько записей с неправильным количеством выборок. Необходимо откорректировать эти записи, прежде чем можно будет использовать объект для симуляции. Пример противоречивого объекта показывают затем.
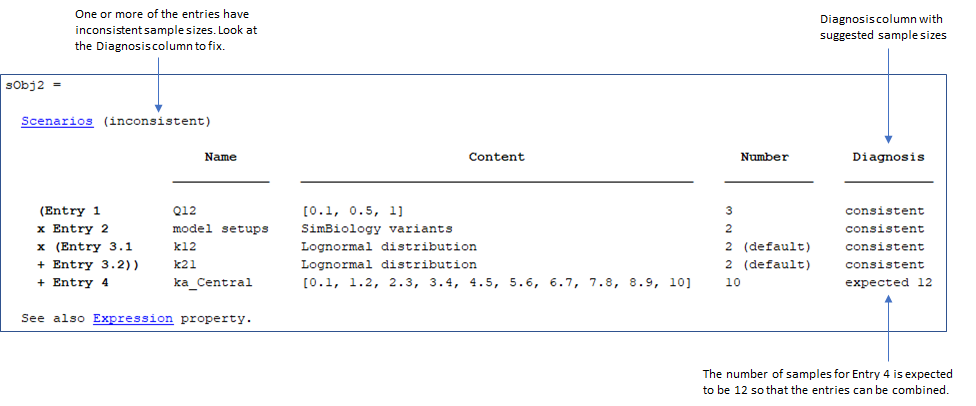
Diagnosis столбец предлагает который записи зафиксировать, чтобы иметь правильное количество выборок. Использование updateEntry, rename, и remove отредактировать записи.
[1] Iman, R. и В.Дж. Коновер. 1982. Подход без распределений к стимулированию порядковой корреляции среди входных переменных. Коммуникации в Статистике - Симуляция и Расчет. 11 (3):311–334.
У вас есть модифицированная версия этого примера. Вы хотите открыть этот пример со своими редактированиями?
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.