Вычислите количество чтений, сопоставленных с геномными функциями
T = featurecount(GTFfile,Inputfile)Inputfile та карта на геномные функции, как задано в отформатированном GTF файле GTFfile. GTFfile задает файл аннотации. Inputfile задает имена BAM или файлов SAM, чтобы рассмотреть. Выход T таблица, где строки соответствуют функциям, и столбцы соответствуют входным файлам. Элементы таблицы состоят из количества отображения чтений с каждой функцией данного входного файла.
[___] = featurecount(___, дополнительные опции использования заданы одним или несколькими Name,Value)Name,Value парные аргументы.
Считайте чтения из демонстрационного файла SAM, которые сопоставляют с функциями, включенными в файл GTF. По умолчанию, featurecount сопоставляет чтения с экзонами и обобщает общее количество чтений на генном уровне.
[t,s] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam');
Processing GTF file Dmel_BDGP5_nohc.gtf ... Processing SAM file rnaseq_sample1.sam ... Processing reference chr2L ... Processing reference chr2R ... Processing reference chr3L ... Processing reference chr3R ... Processing reference chr4 ... Processing reference chrX ... Done.
Отобразите первые 10 строк данных о количестве.
t(1:10,:)
ans=10×3 table
ID Reference rnaseq_sample1
_______________ _________ ______________
{'FBgn0002121'} {'chr2L'} 9
{'FBgn0067779'} {'chr2L'} 2
{'FBgn0005278'} {'chr2L'} 4
{'FBgn0031220'} {'chr2L'} 4
{'FBgn0025683'} {'chr2L'} 13
{'FBgn0053635'} {'chr2L'} 2
{'FBgn0016977'} {'chr2L'} 22
{'FBgn0086902'} {'chr2L'} 27
{'FBgn0031245'} {'chr2L'} 2
{'FBgn0024352'} {'chr2L'} 2
ID столбец содержит имена функций (гены в этом примере). Reference списки столбцов имена ссылочных последовательностей для функций. Третий столбец содержит общее количество чтений, сопоставленных с каждой функцией данного файла SAM, то есть, rnaseq_sample1.sam. По умолчанию таблица показывает только те функции (строки) и файлы SAM (столбцы) с ненулевыми количествами чтения. Установите 'ShowZeroCounts' на истину включать те строки и столбцы со всеми нулевыми количествами в выходной таблице.
s содержит итоговую статистику присвоенных и неприсвоенных чтений из каждого файла SAM. Например, TotalEntries строка указывает на общее количество записей выравнивания из данного файла SAM и Assigned строка включает количество чтений, которые присвоены функциям в файле GTF. Для получения дополнительной информации о каждой строке, обратитесь к разделу Output Arguments страницы с описанием.
s
s=9×1 table
rnaseq_sample1
______________
TotalEntries 33354
Assigned 16399
Unassigned_ambiguous 167
Unassigned_filtered 0
Unassigned_lowMappingQuality 0
Unassigned_multiMapped 0
Unassigned_noFeature 16788
Unassigned_supplementary 0
Unassigned_unmapped 0
Считайте чтения без любого резюмирования и отключите отображение сообщений о ходе выполнения.
[t2,s2] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'Summarization',false,'Verbose',false);
Заметьте, что столбец ID выходной таблицы теперь сообщает об атрибуте функции, сопровождаемом запуском, и остановите положения каждой функции, разделенной символами нижнего подчеркивания.
t2(1:10,:)
ans=10×3 table
ID Reference rnaseq_sample1
_____________________________ _________ ______________
{'FBgn0002121_12286_12928' } {'chr2L'} 3
{'FBgn0002121_13683_14874' } {'chr2L'} 1
{'FBgn0002121_14933_15711' } {'chr2L'} 3
{'FBgn0067779_67044_67507' } {'chr2L'} 2
{'FBgn0005278_108588_108809'} {'chr2L'} 1
{'FBgn0005278_110755_110877'} {'chr2L'} 1
{'FBgn0005278_112690_113369'} {'chr2L'} 1
{'FBgn0031220_117079_117759'} {'chr2L'} 2
{'FBgn0031220_118361_118874'} {'chr2L'} 1
{'FBgn0031220_118931_119076'} {'chr2L'} 1
Можно выбрать, как присвоить чтение конкретной функции, когда чтение перекрывается с несколькими функциями путем установки опции 'OverlapMethod'. Например, если вы хотите считать только для чтения, если это полностью перекрывает функцию, используйте 'полную' опцию.
[tFull, sFull] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'OverlapMethod','full','Verbose',false);
Если у вас есть данные парного конца, можно считать чтения как фрагменты.
[tFrag,sFrag] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'CountFragments',true,'Verbose',false);
Можно также считать фрагменты из нескольких файлов SAM.
[t2,s2] = featurecount('Dmel_BDGP5_nohc.gtf',... {'rnaseq_sample1.sam','rnaseq_sample2.sam'},'CountFragments',true, ... 'Verbose',false);
Используйте следующие опции, чтобы считать чтения парного конца, где по крайней мере один из помощников чтения выше определенного качественного порога отображения.
[t3,s3] = featurecount('Dmel_BDGP5_nohc.gtf',... 'rnaseq_sample1.sam','CountFragments',true,'MinMappingQuality',20, ... 'Verbose',false);
Если чтения прибывают из какого-либо специфичного для скрутки испытания, можно задать такую специфику скрутки во время подсчета. Например, если протокол скручивается, скрутка функции по сравнению со скруткой чтения. Затем только те чтения, которые имеют ту же скрутку как перекрытая функция, считаются.
[t4,s4] = featurecount('Dmel_BDGP5_nohc.gtf',... 'rnaseq_sample1.sam','StrandSpecificity','stranded','Verbose',false);
GTFfile — Отформатированное GTF имя файлаОтформатированное GTF имя файла в виде вектора символов или строки.
Пример: 'Dmel_BDGP5_nohc.gtf'
Inputfile — Отформатированное BAM или SAM-отформатированное имя файлаОтформатированное BAM или SAM-отформатированное имя файла в виде вектора символов, строки, представляет в виде строки вектор или массив ячеек из символьных векторов.
Пример: 'rnaseq_sample1.sam'
Задайте дополнительные разделенные запятой пары Name,Value аргументы. Name имя аргумента и Value соответствующее значение. Name должен появиться в кавычках. Вы можете задать несколько аргументов в виде пар имен и значений в любом порядке, например: Name1, Value1, ..., NameN, ValueN.
'CountFragments',true задает, чтобы считать чтения как пары помощников.'Feature' — Покажите тип'exon' (значение по умолчанию) | вектор символов | строкаПокажите тип в виде вектора символов или строки. Это используется, чтобы решить что функцию рассмотреть из файла GTF. Значением по умолчанию является 'exon'.
'Metafeature' — Припишите тип'gene_id' (значение по умолчанию) | вектор символов | строкаПрипишите тип в виде вектора символов или строки. Это используется, чтобы решить, какой атрибут рассмотреть из файла GTF для группировки функций в метафункции и суммирование чтения считают.
'Summarization' — Логическая переменная, указывающая, подвести ли итог на уровне метафункцииtrue (значение по умолчанию) | falseЛогическая переменная, указывающая, подвести ли итог на уровне метафункции в виде true или false.
Значением по умолчанию является true, значение функциональных функций групп в метафункции и отчеты чтение значит метафункции.
'Alias' — Имя файла, содержащего псевдонимы ссылочных именИмя файла, содержащего псевдонимы ссылочных имен в виде вектора символов или строки. Файл должен быть файлом с разделением табуляцией, где первый столбец соответствует ссылочным именам, используемым в файле GTF, и второй столбец соответствует ссылочным именам, используемым во входном файле (файлах). Имена являются чувствительными к регистру. Необходимо включать только ссылочные имена, которые отличаются в файле GTF и входном файле. Файл должен содержать только один термин псевдонима для любой ссылки, перечисленной во входном файле. По умолчанию ссылочные имена в файле GTF и те во входных файлах приняты, чтобы быть тем же самым.
'CountFragments' — Логическая переменная, указывающая, считать ли чтения как пары помощниковfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, считать ли чтения как фрагменты в виде true или false. Чтения парного конца должны иметь тот же ID для поля QNAME во входном файле и взаимном порядке помощников выведен соответствующим битом в FLAG поле во входном файле. Чтения, которые не имеют никакого допустимого помощника или потому что помощник не сопоставлен или отфильтрован входными критериями, все еще считаются, если они удовлетворяют перекрывающимся критериям.
Значением по умолчанию является false, то есть, чтения считаются, когда одно конец читает, и их информация о соединении проигнорирована.
'StrandSpecificity' — Скрутите специфику секвенирования протокола'unstranded' (значение по умолчанию) | 'stranded' | 'reverse'Скрутите специфику протокола секвенирования в виде 'unstranded' (значение по умолчанию), 'stranded', или 'reverse'.
Если 'unstranded', скрутка чтений (или фрагменты) проигнорирована.
Если 'stranded', скрутка чтений (или фрагменты) рассматривается, и только те, которые имеют ту же скрутку как функция, которую они перекрывают, считаются.
Если 'reverse', противоположное направление скрутки чтений (или фрагменты) рассматривается, и только те, которые имеют противоположную скрутку как функция, которую они перекрывают, считаются.
При подсчете фрагментов (чтения парного конца), скрутка первого помощника рассматривается как скрутку целого фрагмента. Взаимный порядок помощников (первый или второй) выведен из соответствующего бита в FLAG поле входного файла.
'MinOverlap' — Минимальное количество перекрытых базисов требуетсяМинимальное количество перекрытых базисов, требуемых присваивать чтение функции в виде положительного целого числа. При подсчете фрагментов сумма перекрытий от каждого конца используется в качестве минимального количества перекрытых базисов.
'MinMappingQuality' — Минимальное качество отображения для данного чтенияМинимальное качество отображения для данного чтения, которое будет рассмотрено для подсчета в виде неотрицательного целого числа. Это соответствует MAPQ поле во входном файле. При подсчете фрагментов по крайней мере один из помощников чтения должен удовлетворить этому критерию для того, чтобы быть рассмотренным для подсчета.
'CountMultiOverlap' — Логическая переменная, указывающая, считать ли чтения, перекрывающие несколько функцийfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, считать ли чтения, перекрывающие несколько функций в виде true или false (значение по умолчанию).
Если true, чтение (или фрагмент) перекрывающий несколько функций считается многократно. Во время резюмирования на уровне метафункции чтение (или фрагмент) считается только однажды, если это перекрывается с несколькими функциями, принадлежащими той же метафункции, пока это не перекрывается с другим metafeaures.
'CountMultiMapped' — Подсчет опции для чтений, имеющих несколько местоположений отображения во входном файле'primary' (значение по умолчанию) | 'none' | 'all'Подсчет опции для чтений, имеющих несколько местоположений отображения во входном файле в виде 'primary' (значение по умолчанию), 'none', или 'all'.
Если 'primary', только первичное выравнивание мультисопоставленного чтения рассматривается. Соответствующий бит во входном файле используется, чтобы идентифицировать первичные выравнивания.
Если 'none', все выравнивания мультисопоставленного чтения проигнорированы. Тег NH используется, чтобы идентифицировать мультисопоставленные чтения.
Если 'all', все выравнивания мультисопоставленного чтения рассматриваются и считаются многократно.
'BothEndsMapped' — Логическая переменная, указывающая, должен ли фрагмент иметь обоих помощников, сопоставилаfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, должен ли фрагмент иметь обоих помощников, сопоставленных в виде true или false. Помощник, сопоставляющий информацию, получен из FLAG поле во входном файле. Значением по умолчанию является false.
'ProperlyPaired' — Логическая переменная, указывающая, должен ли фрагмент быть правильно соединенfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, должен ли фрагмент быть правильно соединен в виде true или false. Помощник, соединяющий информацию, получен из FLAG поле во входном файле. Значением по умолчанию является false.
'ShowZeroCounts' — Логическая переменная, указывающая, сообщить ли о функциях или метафункциях с нулевым количествомfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, сообщить ли о функциях или метафункциях с нулем, значит каждый входной файл в выходной таблице в виде true или false.
Значением по умолчанию является false, то есть, только строки с ненулевыми количествами и столбцы с ненулевыми количествами включены в выходную таблицу.
'OverlapMethod' — Метод, чтобы использовать при присвоении данного чтения, чтобы метапоказать'partial' (значение по умолчанию) | 'full' | 'max' | 'hits'Метод, чтобы использовать при присвоении данного чтения, чтобы метапоказать в виде 'partial'полныйMax , или 'hits'. Если 'Summarization' установлен в false, затем чтения присвоены функциям, вместо метафункций, на основе заданного метода.
В следующей таблице R относится к чтению или фрагменту, и M относится к метафункции.
| Метод | Описание |
|---|---|
'partial' | R присвоен M, если R перекрывается (даже частично) только с M. В противном случае R рассматривается неоднозначным. |
'full' | R присвоен M, если R полностью сопоставлен только в M, то есть, полностью перекрывающийся только M. В противном случае R рассматривается неоднозначным |
'max' | R присвоен M, если R удовлетворяет перекрывающимся критериям только M, или если R удовлетворяет перекрывающимся критериям несколькими метафункциями, но перекрывается полностью только с M. |
'hits' | R присвоен M, если R перекрывает даже частично только M, или если M является единственной метафункцией с самым большим количеством функций, пораженных R; в противном случае R рассматривается неоднозначным. |
Следующая принципиальная схема и таблица иллюстрируют результат этих методов в сочетании с 'CountMultiOverlap' аргумент пары "имя-значение". На рисунке чтение относится к последовательности короткого чтения из входного файла, и функция A и функция B относятся к функциям, перечисленным в файле GTF.
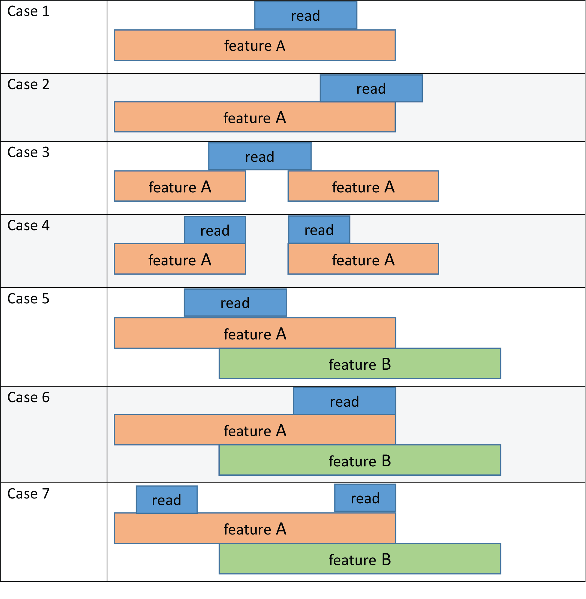
Каждые списки столбцов метода функция, что чтение присвоено на основе соответствующего метода. 'CountMultiOverlap' столбец указывает, установлена ли эта пара "имя-значение" в true или false и если это оказывает какое-либо влияние в результате каждого метода.
'CountMultiOverlap' | 'partial' | 'full' | 'max' | 'hits' | |
|---|---|---|---|---|---|
| Случай 1 | Никакой эффект начиная с чтения не сопоставляет только с одной функцией (покажите A). | покажите A | покажите A | покажите A | покажите A |
| Случай 2 | Никакой эффект начиная с чтения не сопоставляет только с одной функцией (покажите A). | покажите A | никакая функция | покажите A | покажите A |
| Случай 3 | Никакой эффект начиная с чтения не сопоставляет только с одной функцией (покажите A). | покажите A | никакая функция | покажите A | покажите A |
| Случай 4 | Никакой эффект начиная с чтения не сопоставляет только с одной функцией (покажите A). | покажите A | покажите A | покажите A | покажите A |
| Случай 5 | false | неоднозначный | покажите A | покажите A | неоднозначный |
true | покажите A, покажите B | покажите A | покажите A | покажите A, покажите B | |
| Случай 6 | false | неоднозначный | неоднозначный | неоднозначный | неоднозначный |
true | покажите A, покажите B | покажите A, покажите B | покажите A, покажите B | покажите A, покажите B | |
| Случай 7 | false | Неоднозначный | покажите A | покажите A | покажите A |
true | покажите A, покажите B | покажите A | покажите A | покажите A |
никакая функция не означает, что чтение не присвоено никакой функции. Если вы задали вторую выходную таблицу S, его Unassigned_noFeature строка постепенно увеличивается одной для такого вхождения. неоднозначный означает, что чтение не присвоено никакой функции, поскольку это удовлетворяет перекрывающимся критериям нескольких функций и Unassigned_ambiguous строка постепенно увеличивается одной для такого вхождения.
'UseParallel' — Логическая переменная, указывающая, вычислить ли параллельноfalse (значение по умолчанию) | trueЛогическая переменная, указывающая, вычислить ли параллельно в виде true или false.
Для того, чтобы выполнить расчет параллельно, у вас должен быть Parallel Computing Toolbox™. Если пул параллели MATLAB® не существует, каждый автоматически создается, когда опция автосоздания включена в ваших параллельных настройках. В противном случае расчет запускается в последовательном режиме.
Значением по умолчанию является false, то есть, последовательный режим.
'Verbose' — Логическая переменная, указывающая, отобразить ли прогресс расчетаtrue (значение по умолчанию) | falseЛогическая переменная, указывающая, отобразить ли прогресс расчета в виде true или false.
T — Результаты, содержащие чтения последовательности, сопоставленные с геномными функциямиРезультаты, содержащие чтения последовательности, сопоставленные с геномными функциями, возвращенными как таблица. Строки соответствуют функциям, и столбцы соответствуют входным файлам. Элементы таблицы состоят из количества чтений, сопоставленных с каждой функцией данного входного файла. Таблица также сообщает о ID каждой функции и ссылочной последовательности для функции.
Когда 'Summarization' установлен в ложь, столбец ID таблицы сообщает об атрибуте метафункции, сопровождаемом запуском и положениями остановки каждой функции, разделенной символами нижнего подчеркивания.
S — Сводные данные присвоенных и неприсвоенных записей выравниванияСводные данные присвоенных и неприсвоенных записей выравнивания, возвращенных как таблица. Каждый столбец таблицы соответствует каждому входному обеспеченному файлу. Таблица имеет следующие строки:
| 'RowNames' | Описание |
|---|---|
TotalEntries | Количество записей (или выравнивания) во входном файле |
Assigned | Количество чтений или фрагментов, которые были присвоены функциям |
Unassigned_ambiguous | Количество неприсвоенных чтений или фрагментов, перекрывающих несколько функций или метафункций |
Unassigned_filtered | Количество записей выравнивания отфильтровано входными критериями |
Unassigned_lowMappingQuality | Количество записей выравнивания отфильтровало из-за низкого качества отображения |
Unassigned_multiMapped | Количество записей выравнивания, не присвоенных из-за соответствующих чтений, сопоставленных с несколькими местоположениями |
Unassigned_noFeature | Количество чтений или фрагментов, не присвоенных любым функциям |
Unassigned_supplementary | Количество записей выравнивания не присвоило, потому что они отмечаются как дополнительные записи для фантастических выравниваний |
Unassigned_unmapped | Количество записей выравнивания не присвоило, потому что соответствующие чтения не сопоставлены |
Чтобы запуститься параллельно, установите 'UseParallel' к true.
Для получения дополнительной информации смотрите 'UseParallel' аргумент пары "имя-значение".
У вас есть модифицированная версия этого примера. Вы хотите открыть этот пример со своими редактированиями?
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.