Рисование вторичной структуры последовательности РНК
rnaplot(RNA2ndStruct)
ha = rnaplot(RNA2ndStruct)
[ha, H] = rnaplot(RNA2ndStruct)
rnaplot(RNA2ndStruct, ...'Sequence', SequenceValue, ...)
rnaplot(RNA2ndStruct, ...'Format', FormatValue, ...)
rnaplot(RNA2ndStruct, ...'Selection', SelectionValue, ...)
rnaplot(RNA2ndStruct, ...'ColorBy', ColorByValue, ...)
RNA2ndStruct | Вторичная структура последовательности РНК, представленной либо:
Совет Используйте |
SequenceValue | Строят график последовательности вторичной структуры РНК, определяемой одним из следующих способов:
RNA2ndStruct. Эта информация необходима, если вы указываете 'Diagram' формат или если необходимо выделить любой из следующих парных вариантов выбора: 'AU', 'UA', 'GC', 'CG', 'GU' или 'UG'. |
FormatValue | Символьный вектор или строка, задающая формат графика. Возможны следующие варианты:
Примечание При указании |
SelectionValue | Одно из следующих действий:
Примечание При указании |
ColorByValue | Символьный вектор или строка, задающая цветовую схему для графика. Возможны следующие варианты:
Примечание При указании Примечание Поскольку внутренние узлы дерева соответствуют парным остаткам, невозможно указать |
ha | Ручка к оси фигуры. |
H | Структура дескрипторов, содержащая подмножество следующих полей, на основе того, что вы указываете для 'Selection' и 'ColorBy' свойства:
|
rnaplot( рисует вторичную структуру РНК, указанную RNA2ndStruct)RNA2ndStructвторичная структура последовательности РНК, представленной символьным вектором или строкой, указывающей скобку, или матрицей связности.
ha = rnaplot(RNA2ndStruct)ha, ручку к оси фигуры.
[ также возвращает ha, H] = rnaplot(RNA2ndStruct)Hструктура дескрипторов, которую можно использовать для построения графиков элементов в окне MATLAB ® Figure .
Совет
Использовать дескрипторы, возвращенные в H для изменения свойств элементов графика, таких как цвет, размер маркера и тип маркера.
H содержит подмножество следующих полей, основанных на том, что вы указали для 'Selection' и 'ColorBy' свойства.
| Область | Описание |
|---|---|
Paired | Обрабатывает все парные остатки |
Unpaired | Обрабатывает все непарные остатки |
A | Обрабатывает все остатки A |
C | Обрабатывает все остатки C |
G | Обрабатывает все остатки G |
U | Обрабатывает все остатки U |
AU | Обрабатывает все пары AU или UA |
GC | Обрабатывает все пары GC или CG |
GU | Обрабатывает все пары GU или UG |
Selected | Обрабатывает все выбранные остатки |
rnaplot( требования RNA2ndStruct, ...'PropertyName', PropertyValue, ...)rnaplot с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
rnaplot( рисует вторичную структуру РНК, указанную RNA2ndStruct, ...'Sequence', SequenceValue, ...)RNA2ndStructи аннотирует его с последовательными позициями, поставляемыми SequenceValue, последовательность РНК, указанная символьным вектором, строкой или структурой, содержащей Sequence поле.
rnaplot( рисует вторичную структуру РНК, указанную RNA2ndStruct, ...'Format', FormatValue, ...)RNA2ndStruct, используя формат, указанный в FormatValue.
FormatValue - символьный вектор или строка, задающая формат графика. Выбор следующий.
| Формат | Описание |
|---|---|
'Circle' (по умолчанию) | Каждое основание представлено точкой на окружности круга произвольного размера. Линии соединяют основания, которые соединяются друг с другом.
|
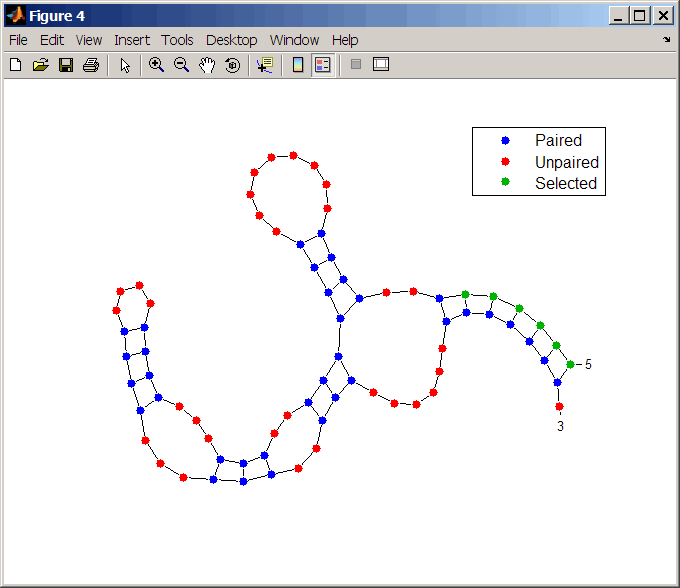
'Diagram' | Двумерное представление вторичной структуры РНК. Каждая основа представлена и идентифицирована буквой. Магистральные и водородные связи между парами оснований представлены линиями.
Примечание При указании |
'Dotdiagram' | Двумерное представление вторичной структуры РНК. Каждое основание представлено и идентифицировано точкой. Магистральные и водородные связи между парами оснований представлены линиями.
|
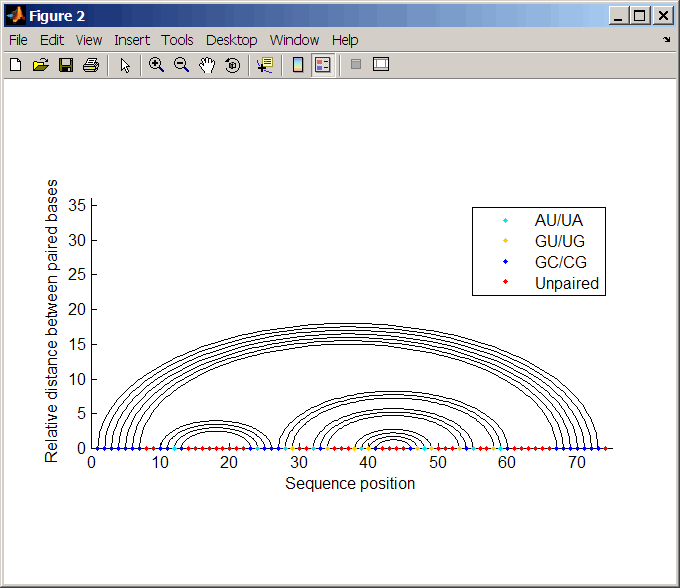
'Graph' | Основания отображаются в их позиции последовательности вдоль абсцисс (оси x) графа. Полуэллиптические линии соединяют основания, которые соединяются между собой. Высота линий пропорциональна расстоянию между парными основаниями.
|
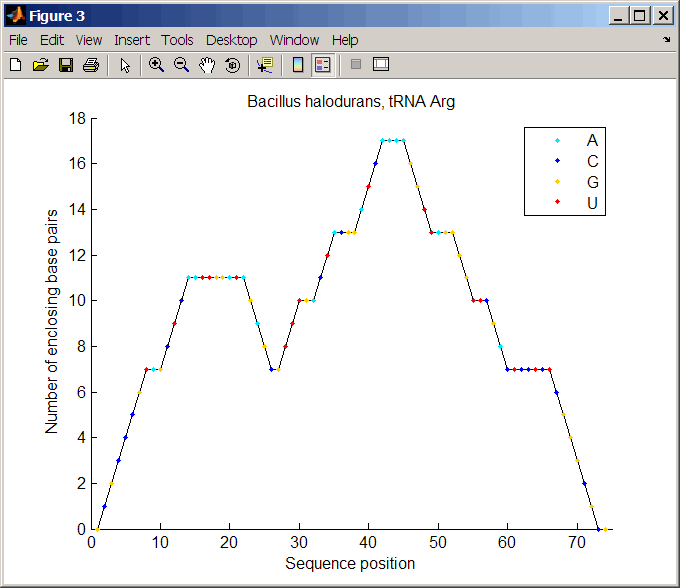
'Mountain' | Каждое основание представлено точкой на двумерном графике, где положение основания находится в абсциссе (ось x), а число пар оснований, охватывающих данное основание, находится в ординате (ось y).
|
'Tree' | Каждое основание представлено узлом в древовидном графе. Конечные узлы указывают непарные основания, в то время как каждый внутренний узел указывает базовую пару. Корень дерева - фиктивный узел, не связанный ни с одной базой во вторичной структуре.
|
rnaplot( рисует вторичную структуру РНК, указанную RNA2ndStruct, ...'Selection', SelectionValue, ...)RNA2ndStruct, выделяя подмножество остатков, указанных SelectionValue. SelectionValue может быть либо:
Числовой массив, определяющий индексы остатков для выделения на графике.
Символьный вектор или строка, задающая подмножество остатков для выделения на графике. Возможны следующие варианты:
'Paired'
'Unpaired'
'AU' или 'UA'
'GC' или 'CG'
'GU' или 'UG'
Примечание
При указании 'AU', 'UA', 'GC', 'CG', 'GU', или 'UG', вы также должны использовать 'Sequence' свойство обеспечивать последовательность РНК.
rnaplot( рисует вторичную структуру РНК, указанную RNA2ndStruct, ...'ColorBy', ColorByValue, ...)RNA2ndStruct, используя цветовую схему, заданную ColorByValue, символьный вектор или строка, указывающая цветовую схему. Возможны следующие варианты:
'State' (по умолчанию) - Цвет по состоянию пары: парные основания и непарные основания.
'Residue' - Цвет по типу остатка (A, C, G и U).
'Pair' - Цвет по типу пары (AU/UA, GC/CG и GU/UG).
Примечание
При указании 'Residue' или 'Pair', вы также должны использовать 'Sequence' свойство обеспечивать последовательность РНК.
Примечание
Поскольку внутренние узлы дерева соответствуют парным остаткам, невозможно указать 'Residue' при указании 'Tree' для 'Format' собственность.
Определите минимальную свободноэнергетическую вторичную структуру последовательности РНК и постройте ее график в формате круга:
seq = 'GCGCCCGUAGCUCAAUUGGAUAGAGCGUUUGACUACGGAUCAAAAGGUUAGGGGUUCGACUCCUCUCGGGCGCG';
ss = rnafold(seq);
rnaplot(ss)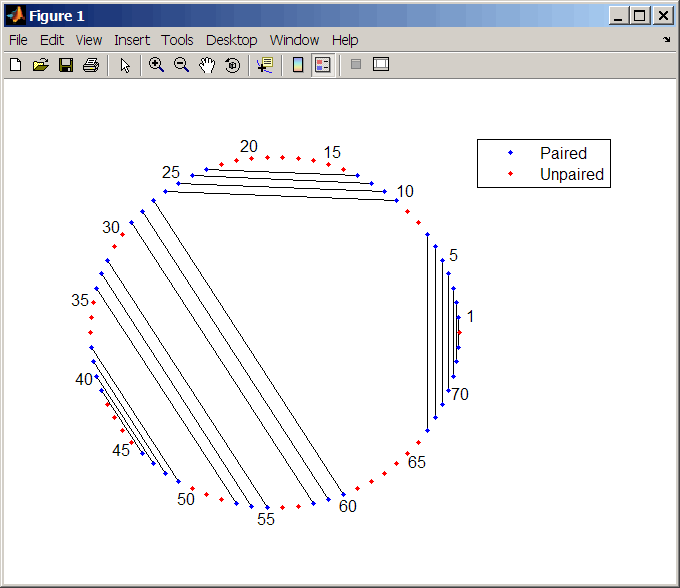
Постройте график вторичной структуры последовательности РНК в формате графа и раскрасьте его по типу пары.
rnaplot(ss, 'sequence', seq, 'format', 'graph', 'colorby', 'pair')
Постройте график вторичной структуры РНК-последовательности в горном формате и раскрасьте ее по типу остатка. Используйте маркер перемещения для добавления заголовка к графику.
ha = rnaplot(ss, 'sequence', seq, 'format', 'mountain',... 'colorby', 'residue') title(ha, 'Bacillus halodurans, tRNA Arg')
Мутируйте первые шесть положений в последовательности и наблюдайте влияние изменения на вторичную структуру, выделяя первые шесть остатков.
seqMut = seq; seqMut(1:6) = 'AAAAAA'; ssMut = rnafold(seqMut); rnaplot(ss, 'sequence', seq, 'format', 'dotdiagram', 'selection', 1:6); rnaplot(ssMut, 'sequence', seqMut, 'format', 'dotdiagram', 'selection', 1:6);
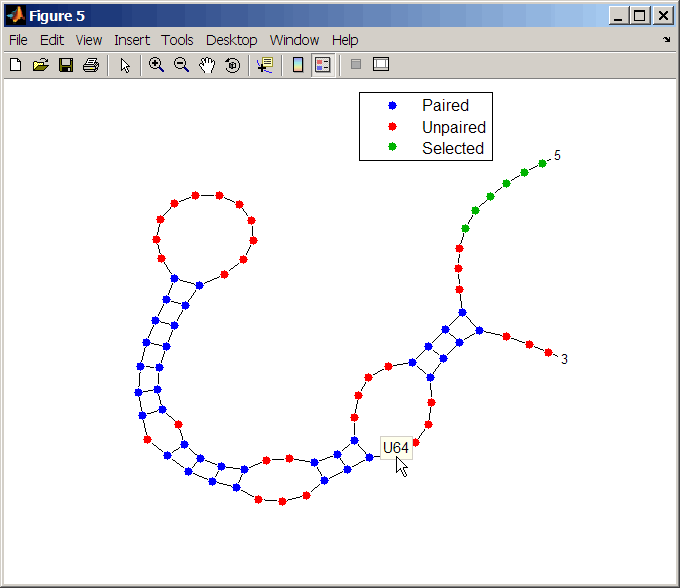
Совет
При необходимости перетащите легенду, удерживая нажатой кнопку мыши, чтобы она не охватывала график. Щелкните основание на графике, чтобы отобразить всплывающую подсказку с информацией об этом основании.