Чтобы начать моделирование, можно:
Создайте модель PK с помощью мастера построения модели, который позволяет указать количество отсеков, маршрут администрирования и тип исключения.
Расширение любой модели для построения моделей с более высокой точностью.
Создайте или загрузите собственную модель. Загрузите проект SimBiology ® или модель SBML.
На следующем рисунке сравнивается модель, как обычно представлено в фармакокинетике, с той же моделью, показанной на диаграмме модели SimBiology. Для этого сравнения предположим, что вы моделируете введение препарата с использованием двухкамерной модели с любым вводом дозирования и линейной кинетикой элиминации. (Структура модели остается прежней для любого типа дозирования.)
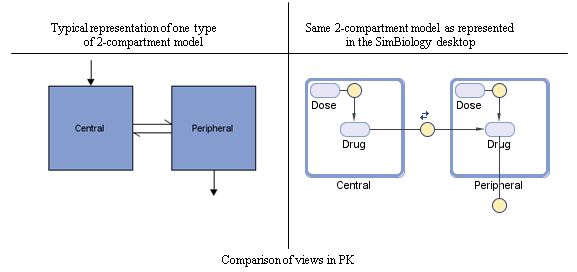
Обратите внимание на следующее:
SimBiology представляет концентрацию или количество лекарственного средства в данном отделении или объеме видовым объектом, содержащимся внутри отделения.
SimBiology представляет обмен или поток лекарственного средства между компартментами и элиминацию лекарственного средства реакциями.
SimBiology представляет межкомпартментальный клиренс по параметру (Q), который определяет зазор между отсеками.
SimBiology управляет графиком дозирования с помощью комбинации видов (Drug и/или Dose) и реакциями (Dose -> Drug), в зависимости от того, следует ли введение в отделение болюсу, кинетике дозирования нулевого порядка, инфузии или первого порядка. Дополнительные сведения о добавленных компонентах и оцененных параметрах см. в разделе Типы дозирования.
Эту модель можно также рассматривать как функцию регрессии. y = f(k,u), где y - прогнозируемое значение, заданные значения входного значения u, и значения параметров k. В SimBiology модель представляет fи модель используется для генерации регрессионной функции, если y, k, и u определяются в модели.
Для создания модели ПК с заданным количеством отсеков, типом дозирования и способом устранения:
Создать PKModelDesign объект. PKModelDesign объект позволяет указать количество отсеков, путь введения и способ устранения, который SimBiology использует для построения модельного объекта с необходимыми отсеками, видами, реакциями и правилами.
pkm = PKModelDesign;
Добавьте отсек с указанием имени отсека и, при необходимости, типа дозирования и метода исключения. Также укажите, содержат ли данные переменную ответа, измеренную в этом отделении, и имеют ли дозы задержки по времени. Например, при использовании набора данных тобрамицина [1] укажите отсек с именемCentral, с Bolus для DosingType собственность, linear-clearance для EliminationType собственность, и true для HasResponseVariable собственность.
pkc1 = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true);Описание других DosingType и EliminationType значения свойств см. в разделах Типы дозирования и Типы исключения.
Для описания HasResponseVariable свойство, см. HasResponseVariable. По крайней мере один отсек в модели должен иметь отклик. Хотя SimBiology поддерживает несколько ответов на отсек, при добавлении отсеков к PKModelDesign объект, вы ограничены одним откликом на отсек.
Примечание
Чтобы добавить отсек, который имеет временной лаг, связанный с любой дозой, которая нацелена на него, установите HasLag свойство для true:
pkc_lag = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true, 'HasLag', true);Или после добавления отсека установите его HasLag свойство для true:
pkc1.HasLag = true;
При необходимости добавьте второй отсек с именем Peripheral, без дозирования, без элиминации и без временной задержки. Установите HasResponseVariable свойство для true. Если используется набор данных тобрамицина [1], пропустите этот шаг и используйте только один отсек.
pkc2 = addCompartment(pkm, 'Peripheral', 'HasResponseVariable', true);
Процесс построения модели добавляет необходимые параметры, включая параметр, представляющий межкомпартментный зазор. Q. Можно добавить дополнительные отсеки, повторив этот шаг. Добавление каждого отсека создает цепочку отсеков в порядке добавления отсека, с двунаправленным потоком препарата между отсеками в модели.
Рукояткой к отсеку (pkc1 или pkc2), для изменения свойств отсека.
Создайте объект модели SimBiology.
[modelObj, PKModelMapObj] = pkm.construct
construct возвращает объект модели SimBiology (modelObj) и PKModelMap объект (PKModelMapObj), которая содержит сопоставление компонентов модели с элементами функции регрессии.
Примечание
При изменении PKModelDesign , необходимо создать новый объект модели с помощью construct способ. Изменения в PKModelDesign не распространяются автоматически на ранее построенный объект модели.
Выполните подгонку параметров, как показано в разделе Выполнение подгонки данных с использованием моделей PK/PD.
Объект модели и PKModelMap object являются входными аргументами для sbionlmefit, sbionlmefitsa и sbionlinfit функции, используемые при подборе параметров.
| Для получения информации о... | Видишь... |
|---|---|
| Типы дозирования | Типы дозирования |
| Виды исключения | Виды исключения |
| Фитинг параметров | Выполнение подгонки данных с использованием моделей PK/PD |
| Моделирование модели и описание наборов конфигурации | Моделирование модели |
При создании моделей SimBiology создает следующие компоненты модели для каждого отсека модели, независимо от типа дозирования:
Два вида (Drug_CompartmentName и Dose_CompartmentName) для каждого отсека.
Реакция (Dose_CompartmentName -> Drug_CompartmentName) для каждого отделения, определяемого кинетикой массового действия.
Параметр (ka_CompartmentName) для каждого отделения, представляющего скорость абсорбции лекарственного средства, когда абсорбция следует кинетике первого порядка. Это параметр прямой скорости для Dose_CompartmentName -> Drug_CompartmentName реакция.
Параметр (Tk0_CompartmentName) для каждого отделения, представляющего продолжительность абсорбции лекарственного средства, когда абсорбция следует кинетике нулевого порядка.
Параметр (TLag_CompartmentName) для каждого отделения, представляющего временную задержку для любой дозы, которая нацелена на это отделение, а также которая указана как имеющая временную задержку.
Для типов дозирования, которые имеют фиксированную продолжительность инфузии или абсорбции (infusion и zero-order), вы можете использовать перекрывающиеся дозы. Дозы являются аддитивными.
В следующей таблице описаны типы дозирования, параметры по умолчанию для оценки и перечислены компоненты модели, созданные и используемые для дозирования.
| Тип дозатора | Описание | Используемые компоненты модели SimBiology | Параметры по умолчанию для оценки |
|---|---|---|---|
''(пустой символьный вектор) | Без дозы | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Предполагает, что количество препарата увеличивается мгновенно во время дозы. В модели SimBiology начальная концентрация лекарственного средства основана на количестве дозы и объеме отделения, содержащего лекарственное средство. | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Предполагается, что количество вносимого лекарственного средства увеличивается с постоянной известной скоростью абсорбции (или инфузии) в течение известной продолжительности. Импортированный набор данных должен содержать скорость, а не продолжительность вливания. SimBiology использует эту информацию для изменения концентрации видов с постоянной скоростью в течение продолжительности, указанной в наборе данных. | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Предполагает, что препарат добавляется с постоянной скоростью в течение фиксированной, но неизвестной продолжительности. |
|
|
Приложение SimBiology - Командная строка - | Предполагает, что скорость всасывания препарата не является постоянной. Предполагается, что в модели SimBiology скорость поглощения определяется кинетикой массового действия. |
|
|
| Вид исключения | Описание | Создание компонентов модели SimBiology | Параметры по умолчанию для оценки |
|---|---|---|---|
Приложение SimBiology - Командная строка - | Предполагает простую кинетику массового действия при элиминации препарата. В модели SimBiology элиминация задается кинетикой массового действия с константой скорости элиминации, заданной параметром прямой скорости (ke). |
|
|
Приложение SimBiology - Командная строка - | Предполагает простую кинетику массового действия при элиминации препарата. В модели SimBiology аналогично Linear {Elimination Rate, Volume}. Но, кроме того, эта опция позволяет указать модель с точки зрения зазора (Cl) где, Cl = ke * volume). |
|
|
Приложение SimBiology - Командная строка - | Предполагает, что устранение регулируется кинетикой Михаэлиса-Ментена. |
|
|
Отсеки, создаваемые при создании модели SimBiology, образуют цепочку, и каждая пара связанных отсеков соединяется транспортной реакцией, аналогичной линейной элиминации. Добавление двух отсеков, C1 и C2, генерирует обратимую реакцию массового действия C1.Drug_C1 <-> C2.Drug. Параметр прямой скорости - это компартментальный зазор, Q12, деленное на объем C1. Параметр обратной скорости имеет значение Q12, деленное на объем C2.
Процесс добавления каждой пары отсеков в цепочку Cm и Cn создает следующие компоненты модели:
Параметр Qmn представляет собой разделительный зазор между этими двумя отсеками. Этот параметр добавляется в список оцениваемых параметров (Estimated имущество PKModelMap объект).
Параметр (kmn), представляющая скорость переноса препарата из Cm кому Cn, где kmn = Qmn/Vm.
Параметр (knm), представляющий собой скорость Cn кому Cm, где knm = Qmn/Vn.
обратимая реакция массового действия между двумя отсеками, Cm.Drug_Cm <-> Cn.Drug_Cn, с параметром прямой скорости kmnи параметр обратной скорости knm.
Начальное правило назначения, инициализирующее значение параметра kmn, на основе исходных значений для Cm и Qmn.
Начальное правило назначения, инициализирующее значение параметра knm, на основе исходных значений для Cn и Qmn.
Преобразование единиц позволяет преобразовать соответствующие физические величины в одну согласованную систему единиц для их разрешения. Это преобразование готовится к правильному моделированию, но SimBiology возвращает физические величины в модели в указанных единицах.
Независимо от того, является ли преобразование единицей измерения on или off, необходимо выразить данные дозирования в количестве. По умолчанию для параметра «Преобразование единиц» установлено значение offпоэтому необходимо убедиться, что единицы измерения для данных и модели согласуются друг с другом. Если преобразование единиц измерения равно on, необходимо указать единицы измерения.
Параметры в модели имеют единицы измерения по умолчанию. Если преобразование единиц onможно изменять единицы измерения до тех пор, пока размеры являются непротиворечивыми. Эти единицы измерения по умолчанию, которые можно использовать для задания значений начального предположения, следующие.
| Физическая величина или параметр модели | Единица |
|---|---|
| Объем центрального или периферийного отсека (центральный или периферийный) | liter |
| Коэффициент исключения первого порядка (ke) | 1/second |
| Константа Михаэлиса (км) | milligram/liter |
| Максимальная скорость реакции, кинетика Михаэлиса-Ментена (Вм) | milligram/second |
| Зазор (Cl) | liter/second |
| Продолжительность поглощения (Tk0) | second |
| Скорость поглощения (ка) | 1/second |
Используйте опции настроек конфигурации для преобразования единиц измерения on или off. Дополнительные сведения см. в разделе Моделирование модели.
Дополнительные сведения о размерном анализе скоростей реакции см. в разделе Оценка скоростей реакции.