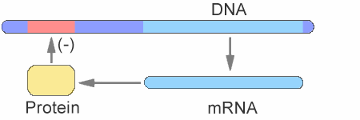
Можно визуализировать модель системной биологии с различными уровнями детализации. Один вид нарисовывает только основные виды и процессы. Эта модель является примером регуляции простых генов, где белковый продукт от трансляции контролирует транскрипцию. Можно создать более сложную модель путем добавления ферментов, коферментов, кофакторов, нуклеотидов и аминокислот, которые не включены в эту модель. Пример регуляции генов упрощает регуляторный механизм, не показывая вклада РНК-полимеразы и каких-либо кофакторов. Белковый продукт от экспрессии генов связывается с регуляторной областью на ДНК и подавляет транскрипцию.
Другой способ рассмотрения модели системной биологии - это список реакций в модели с процессами, которые они представляют.
DNA -> DNA + mRNA (transcription) mRNA -> mRNA + protein (translation) DNA + protein -> DNAProteinComplex (binding) DNAProteinComplex -> DNA + protein (unbinding) mRNA -> null (degradation) protein -> null (degradation)
Построение путей реакции поможет вам визуализировать отношения между реакциями и видами. В примере регуляции генов, когда количество белка увеличивается, белок образует комплекс с геном, ответственным за его экспрессию, и замедляет выработку белка.

Уравнения реакции определяют модель биологии систем на уровне детализации, необходимом для программной программы, чтобы симулировать динамическое поведение модели. Следующие реакции транскрипции, трансляции, связывания и деградации описывают простой механизм регулирования генов.
Транскрипция. Транскрипция - это где РНК-полимераза и кофакторы связываются с молекулой ДНК. Затем РНК-полимераза перемещается вдоль ДНК и объединяет нуклеотиды для создания мРНК. Простая модель транскрипции показывает только ДНК и мРНК.

Эта модель упрощает транскрипцию и синтез мРНК с помощью одной реакции.
| Реакция | DNA -> DNA + mRNA |
| Скорость реакции | v = k1 * DNA молекула/сек |
| Разновидности | DNA = 50 молекула |
mRNA = 0 молекула | |
| Параметры | k1 = 0.20 второй-1 |
Перевод. После того, как мРНК переходит от ядра к цитоплазме, она может связываться с рибосомами. Рибосомы перемещаются вдоль мРНК и создают белки с помощью тРНК, связанных с аминокислотами. Простая модель трансляции показывает только мРНК и белковый продукт.
Синтез белка моделируется как единая реакция.
| Реакция | mRNA -> mRNA + protein |
| Скорость реакции | v = k2 * mRNA молекула/сек |
| Разновидности | mRNA = 0 молекула |
protein = 0 молекула | |
| Параметры | k2 = 20 второй-1 |
Генная репрессия. Транскрипция ДНК к мРНК регулируется связыванием белкового продукта от трансляции к ДНК. Поскольку продуцируется больше белка, ДНК связана с белком чаще, и меньше времени доступно для транскрипции с несвязанной ДНК.
Прямая реакция (связывание)
| Реакция | DNA + protein -> DNAProteinComplex |
| Скорость реакции | v = k3 * DNA * protein молекула/сек |
| Разновидности | DNA = 50 молекула |
protein = 0 молекула | |
| Параметры | k3 = 0.2 1/( молекула * секунда) |
Обратная реакция (демпфирование)
| Реакция | DNAProteinComplex -> DNA + protein |
| Скорость реакции | v = k3r * DNA_protein молекула/сек |
| Разновидности | DNAProteinComplex = 0 молекула |
| Параметры | k3r = 1 второй-1 |
Для этого руководства моделируйте связывающие и несвязывающие реакции как одну обратимую реакцию.
| Реакция | DNA + protein <-> DNA_protein |
| Скорость реакции | v = k3 * DNA * protein - k3r * DNA_protein молекула/сек |
| Разновидности | DNA = 50 молекула |
protein = 0 молекула | |
| Параметры | k3 = 0.2 1/( вторая * молекула) |
k3r = 1 второй-1 |
Деградация. Деградация белка и мРНК являются важными реакциями для регулирования экспрессии генов. Установившийся уровень этих соединений поддерживается балансом между реакциями синтеза и деградации. Белки гидролизуются до аминокислот с помощью протеаз, а нуклеиновые кислоты деградируют до нуклеотидов.
![]()
деградация мРНК моделируется как преобразование в null вид.
| Реакция | mRNA -> null |
| Скорость реакции | v = k4 * mRNA молекула/сек |
| Разновидности | mRNA = 0 молекула |
| Параметры | k4 = 1.5 второй-1 |
Точно так же деградация белка также моделируется как преобразование в null вид. Вид null является предопределенным видовым именем в SimBiology® модели.
![]()
| Реакция | protein -> null |
| Скорость реакции | v = k5 * protein молекула/сек |
| Разновидности | protein = 0 молекула |
| Параметры | k5 = 1 второй-1 |
Модель SimBiology является набором объектов, которые структурированы иерархически. Объект модели нужен, чтобы содержать все другие объекты.
Создайте модель SimBiology с именем cell.
Mobj = sbiomodel('cell')Mobj =
SimBiology Model - cell
Model Components:
Compartments: 0
Events: 0
Parameters: 0
Reactions: 0
Rules: 0
Species: 0
Observables: 0
Добавьте отсек с именем comp на модель и установите модуль вместимости отсека.
compObj = addcompartment(Mobj,'comp'); compObj.CapacityUnits = 'liter';
Добавьте реакционную DNA -> DNA + mRNA в модель. SimBiology автоматически добавляет вид DNA и mRNA в модель с начальной суммой по умолчанию 0.
Robj1 = addreaction(Mobj,'DNA -> DNA + mRNA');Примечание
Поскольку этот пример имеет только один отсек, вам не нужно указывать отсек, к которому относится каждый вид. Если существует несколько отсеков, вот пример синтаксиса реакции:
Robj1 = addreaction(Mobj, 'nucleus.DNA -> nucleus.DNA + cytoplasm.mRNA');
nucleus и cytoplasm - имена отсеков.Отобразите добавленные виды модели.
Mobj.Species
ans = SimBiology Species Array Index: Compartment: Name: Value: Units: 1 comp DNA 0 2 comp mRNA 0
Установите начальное количество DNA на 50 а также количество модулей для обоих видов.
Mobj.Species(1).InitialAmount = 50; Mobj.Species(1).InitialAmountUnits = 'molecule'; Mobj.Species(2).InitialAmountUnits = 'molecule';
Задайте кинетику реакции, которая будет массовым действием, создав объект кинетического закона действия масс, Kobj1.
Kobj1 = addkineticlaw(Robj1,'MassAction');Для невозвратной реакции MassAction кинетика определяет выражение скорости реакции как forward rate constant * reactants.
Кинетический закон служит картой между параметрами и видами, необходимыми для экспрессии скорости реакции и параметров и видов в модели. Чтобы увидеть параметры и виды, которые должны быть сопоставлены, извлеките ParameterVariables и SpeciesVariables свойства Kobj1.
Kobj1.ParameterVariables
ans = 1x1 cell array
{'Forward Rate Parameter'}
Kobj1.SpeciesVariables
ans = 1x1 cell array
{'MassAction Species'}
Поскольку кинетический закон требует параметра прямой скорости, создайте параметр, k1, и установите его значение равным 0.2. Сопоставьте k1 параметра в параметр прямой скорости путем установки ParameterVariablesNames свойство Kobj1 на k1.
Pobj1 = addparameter(Kobj1,'k1'); Pobj1.Value = 0.2; Pobj1.ValueUnits = '1/second'; Kobj1.ParameterVariableNames = 'k1';
Для кинетики действующих масс SpeciesVariables автоматически назначаются реагентам. Поэтому SpeciesVariablesNames свойство Kobj1 автоматически устанавливается на DNA. Экспрессия скорости реакции теперь определяется следующим образом.
Robj1.ReactionRate
ans = 'k1*DNA'
Простая модель трансляции показывает только мРНК и белковый продукт. Для получения дополнительной информации смотрите Перевод.
Введите реактивную mRNA -> mRNA + protein и установите его кинетический закон в массовое действие. Также установите модуль измерения величины вида protein.
Robj2 = addreaction(Mobj,'mRNA -> mRNA + protein'); Mobj.Species(3).InitialAmountUnits = 'molecule'; Kobj2 = addkineticlaw(Robj2,'MassAction');
Задайте константу скорости реакции k2 для реакции.
Pobj2 = addparameter(Kobj2,'k2'); Pobj2.Value = 20; Pobj2.ValueUnits = '1/second'; Kobj2.ParameterVariableNames = 'k2';
Скорость реакции теперь определяется следующим образом.
Robj2.ReactionRate
ans = 'k2*mRNA'
Транскрипция ДНК к мРНК регулируется связыванием белкового продукта от трансляции к ДНК. Поскольку продуцируется больше белка, ДНК связана с белком чаще, и меньше времени доступно для транскрипции с несвязанной ДНК. Для получения дополнительной информации смотрите Gene Repression.
Вступает в обратимую реакцию связывания и разблокирования ДНК и белка. Добавьте параметр k3 как константа прямой скорости, и k3r как обратная константа скорости.
Robj3 = addreaction(Mobj,'DNA + protein <-> DNAProteinComplex'); Mobj.Species(4).InitialAmountUnits = 'molecule'; Kobj3 = addkineticlaw(Robj3,'MassAction'); Pobj3 = addparameter(Kobj3,'k3','Value',0.2,'ValueUnits','1/(molecule*second)'); Pobj3r = addparameter(Kobj3,'k3r','Value',1.0,'ValueUnits','1/second'); Kobj3.ParameterVariableNames = {'k3','k3r'};
Отобразите скорость реакции.
Robj3.ReactionRate
ans = 'k3*DNA*protein - k3r*DNAProteinComplex'
Деградация белка и мРНК являются важными реакциями для регулирования экспрессии генов. Установившийся уровень соединений поддерживается балансом между реакциями синтеза и деградации. Белки гидролизуются до аминокислот с помощью протеаз, в то время как нуклеиновые кислоты деградируют до нуклеотидов.
Вступает в реакцию на разложение мРНК до нуклеотидов. Добавьте параметр k4 как константа прямой скорости.
Robj4 = addreaction(Mobj,'mRNA -> null'); Kobj4 = addkineticlaw(Robj4, 'MassAction'); Pobj4 = addparameter(Kobj4,'k4','Value',1.5,'ValueUnits','1/second'); Kobj4.ParameterVariableNames = 'k4';
Скорость реакции разложения мРНК.
Robj4.ReactionRate
ans = 'k4*mRNA'
Вступите в реакцию на деградацию белка до аминокислот. Добавьте параметр k5 как константа прямой скорости для реакции.
Robj5 = addreaction(Mobj,'protein -> null'); Kobj5 = addkineticlaw(Robj5,'MassAction'); Pobj5 = addparameter(Kobj5,'k5','Value',1.0,'ValueUnits','1/second'); Kobj5.ParameterVariableNames = 'k5';
Скорость реакции деградации белка.
Robj5.ReactionRate
ans = 'k5*protein'
Симулируйте модель, чтобы увидеть ее динамическое поведение.
Сначала включите дополнительную функцию модуля. Эта функция автоматически преобразует модули измерения физических величин в одну последовательную систему. Это преобразование готовится к правильной симуляции, но количества видов возвращаются в модуле измерения, которую вы указали (molecule в этом примере).
configset = getconfigset(Mobj); configset.CompileOptions.UnitConversion = true;
Запустите симуляцию.
[t, simdata, names] = sbiosimulate(Mobj);
Постройте график результатов.
plot(t,simdata) legend(names,'Location','NorthEastOutside') title('Gene Regulation'); xlabel('Time'); ylabel('Species Amount');
