Чтобы начать моделирование, можно:
Создайте модель PK с помощью мастера конструкции модели, который позволяет вам задать количество отсеков, маршрут администрирования и тип исключения.
Расширьте любую модель, чтобы создать модели более высокой точности.
Создайте или загрузите свою собственную модель. Загрузка SimBiology® модель project или SBML.
Следующий рисунок сравнивает модель, обычно представленную в фармакокинетике, с той же моделью, показанной на диаграмме модели SimBiology. Для этого сравнения предположим, что вы моделируете введение препарата с помощью двухкамерной модели с любым входным дозированием и линейной кинетикой элиминации. (Структура модели остается прежней с любым типом дозирования.)
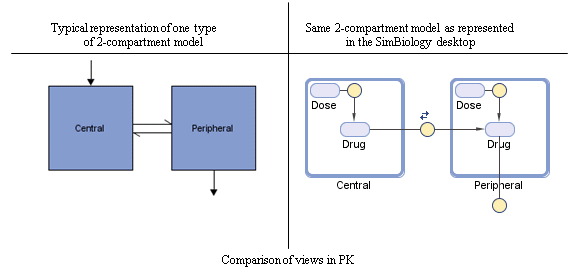
Обратите внимание на следующее:
SimBiology представляет концентрацию или количество лекарственного средства в заданном отсеке или объеме видовым объектом, содержащимся в отсеке.
SimBiology представляет обмен или поток препарата между отделениями и устранение препарата реакциями.
SimBiology представляет межкомпартментарное клиренс по параметру (Q), который определяет зазор между отсеками.
SimBiology управляет графиком дозирования с помощью комбинации видов (Drug и/или Dose) и реакции (Dose -> Drug), в зависимости от того, следует ли введение в отделение болюсу, нулевому порядку, инфузии или кинетике дозирования первого порядка. Для получения дополнительной информации о добавленных компонентах и оцененных параметрах смотрите Типы дозирования.
Можно также просмотреть эту модель как регрессионую функцию, y = f(k,u), где y - предсказанное значение, заданные значения входа u, и значения параметров k. В SimBiology модель представляет f, и модель используется, чтобы сгенерировать регрессионую функцию, если y, k, и u идентифицируются в модели.
Для создания модели ПК с заданным количеством отсеков, типом дозирования и методом устранения:
Создайте PKModelDesign объект. The PKModelDesign позволяет вам задать количество отсеков, путь введения и метод удаления, которые SimBiology использует для построения объекта модели с необходимыми отсеками, видами, реакциями и правилами.
pkm = PKModelDesign;
Добавить отсек с указанием имени отсека и, опционально, типа дозирования и способа удаления. Также укажите, содержат ли данные переменную отклика, измеренную в этом отсеке, и имеют ли доза (дозы) временные лаги. Для примера, если используется набор данных тобрамицина [1], задайте отсек с именем Central, с Bolus для DosingType свойство, linear-clearance для EliminationType свойство, и true для HasResponseVariable свойство.
pkc1 = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true);Описание других DosingType и EliminationType значения свойств, см. «Типы дозирования» и «Типы устранения».
Описание HasResponseVariable свойство, см. HasResponseVariable. По крайней мере, один отсек в модели должен иметь ответ. Хотя SimBiology поддерживает несколько откликов в каждом отсеке, при добавлении отсеков в PKModelDesign объект, вы ограничены одним ответом на каждый отсек.
Примечание
Чтобы добавить отсек, который имеет временную задержку, связанную с любой дозой, которая нацелена на него, установите HasLag свойство к true:
pkc_lag = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true, 'HasLag', true);Или после добавления отсека установите его HasLag свойство к true:
pkc1.HasLag = true;
Вы можете добавить второй отсек с именем Peripheralбез дозировки, без исключения и без временной задержки. Установите HasResponseVariable свойство к true. Если вы используете набор данных тобрамицина [1], пропустите этот шаг и используйте только один отсек.
pkc2 = addCompartment(pkm, 'Peripheral', 'HasResponseVariable', true);
Процесс конструкции модели добавляет необходимые параметры, включая параметр, представляющий межкомпартментальный клиренс Q. Можно добавить больше отсеков, повторив этот шаг. Сложение каждого отделения создает цепь отсеков в порядке сложения отсека с двунаправленным потоком лекарства между отсеками в модели.
Используйте указатель в отсек (pkc1 или pkc2), для изменения свойств отсека.
Создайте объект модели SimBiology.
[modelObj, PKModelMapObj] = pkm.construct
The construct метод возвращает объект модели SimBiology (modelObj) и a PKModelMap объект (PKModelMapObj), который содержит отображение компонентов модели с элементами регрессионной функции.
Примечание
Если вы меняете PKModelDesign объект, необходимо создать новый объект модели с помощью construct способ. Изменения в PKModelDesign не распространяются автоматически на ранее созданный объект модели.
Выполните аппроксимацию параметров, как показано на Perform Data Fitting with PK/PD Models.
Объект модели и PKModelMap объект являются входными параметрами для sbionlmefit, sbionlmefitsa и sbionlinfit функции, используемые в параметр подбора кривой.
| Для получения информации о... | Видите... |
|---|---|
| Типы дозирования | Типы дозирования |
| Типы исключения | Типы исключения |
| Параметрический подбор кривой | Выполните подбор данных с помощью моделей PK/PD |
| Симуляция модели и описание конфигураций модели | Симуляция модели |
При создании моделей SimBiology создает следующие компоненты модели для каждого отделения модели, независимо от типа дозирования:
Два вида (Drug_CompartmentName и Dose_CompartmentName) для каждого отсека.
Реакция (Dose_CompartmentName -> Drug_CompartmentName) для каждого отсека, регулируемого кинетикой действующих масс.
Параметр (ka_CompartmentName) для каждого отделения, представляющего скорость поглощения лекарственного средства, когда поглощение следует кинетике первого порядка. Это параметр прямой скорости для Dose_CompartmentName -> Drug_CompartmentName реакция.
Параметр (Tk0_CompartmentName) для каждого отделения, представляющего длительность поглощения лекарственного средства, когда поглощение следует кинетике нулевого порядка.
Параметр (TLag_CompartmentName) для каждого отделения, представляющего временную задержку для любой дозы, которая нацелена на это отделение, а также которая определяется как имеющая временную задержку.
Для типов дозирования, которые имеют фиксированную длительность инфузии или абсорбции (infusion и zero-order), вы можете использовать перекрывающиеся дозы. Дозы являются аддитивными.
В следующей таблице описаны типы дозирования, параметры по умолчанию для оценки и перечислены компоненты модели, созданные и используемые для дозирования.
| Тип дозирования | Описание | Используемые компоненты модели | Параметры по умолчанию для оценки |
|---|---|---|---|
''(пустой символьный вектор) | Доза отсутствует | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Принимает, что количество препарата увеличивается мгновенно во время дозы. В модели SimBiology начальная концентрация препарата основана на суммарной дозе и объеме отсека, содержащего препарат. | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Принимает, что количество введенного лекарственного средства увеличивается с постоянной известной скоростью абсорбции (или инфузии) в течение известной длительности. Импортированный набор данных должен содержать скорость, а не длительность инфузии. SimBiology использует эту информацию, чтобы изменить концентрацию видов с постоянной скоростью в течение длительности, указанной в наборе данных. | Вид ( | Ничего |
Приложение SimBiology - Командная строка - | Принимает, что препарат добавляется с постоянной скоростью в течение фиксированной, но неизвестной длительности. |
|
|
Приложение SimBiology - Командная строка - | Принимает, что скорость, с которой препарат поглощается, не является постоянной. В модели SimBiology скорость поглощения определяется кинетикой действующих масс. |
|
|
| Тип исключения | Описание | Созданные компоненты модели | Параметры по умолчанию для оценки |
|---|---|---|---|
Приложение SimBiology - Командная строка - | Принимает простую кинетику действующих масс в элиминации препарата. В модели SimBiology устранение задается кинетикой действующих масс с константой скорости исключения, заданной параметром прямой скорости (ke). |
|
|
Приложение SimBiology - Командная строка - | Принимает простую кинетику действующих масс в элиминации препарата. В модели SimBiology, подобной Linear {Elimination Rate, Volume}. Но, в сложение, эта опция позволяет вам задать модель с точки зрения зазора (Cl) где, Cl = ke * volume). |
|
|
Приложение SimBiology - Командная строка - | Принимает, что устранение регулируется михаэлис-Ментеном. |
|
|
Отсеки, созданные при генерации модели SimBiology, образуют цепь, и каждая пара связанных отсеков соединяется транспортной реакцией, подобной линейному исключению. Сложение двух отсеков, C1 и C2, генерирует обратимую реакцию массового действия C1.Drug_C1 <-> C2.Drug. Передний параметр скорости является отсекающим зазором, Q12, разделенная на объем C1. Параметр обратной скорости Q12, разделенная на объем C2.
Процесс добавления каждой пары отсеков в цепь Cm и Cn генерирует следующие компоненты модели:
A параметра Qmn представляет собой разрыв между этими двумя отсеками. Этот параметр добавляется к списку параметров, которые будут оценены (Estimated свойство PKModelMap объект).
Параметр (kmn), представляющий скорость передачи препарата от Cm на Cn, где kmn = Qmn/Vm.
Параметр (knm), представляющий скорость Cn на Cm, где knm = Qmn/Vn.
Обратимая реакция массового действия между двумя отделениями, Cm.Drug_Cm <-> Cn.Drug_Cn, с параметром прямой скорости kmn, и параметр обратной скорости knm.
Начальное правило назначения, которое инициализирует значение параметра kmn, на основе начальных значений для Cm и Qmn.
Начальное правило назначения, которое инициализирует значение параметра knm, на основе начальных значений для Cn и Qmn.
Модуль преобразования преобразует совпадающие физические величины в один последовательный модуль систему порядка чтобы разрешить их. Это преобразование готовится к правильной симуляции, но SimBiology возвращает физические величины в модели в модулях измерения, которые вы задаете.
Независимо от того, является ли модуль on или off, вы должны выразить данные дозирования в количестве. По умолчанию Unit Conversion есть offТаким образом, вы должны убедиться, что модули для данных и модели согласованы друг с другом. Если Unit Conversion onнеобходимо задать модули.
Параметры в модели имеют модули измерения по умолчанию. Если модуль измерения onможно изменять модули, пока размерности непротиворечивы. Эти модули измерения по умолчанию, которые можно использовать, чтобы задать значения для начального предположения, следующие.
| Физическая величина или параметр модели | Модуль |
|---|---|
| Объем центрального или периферийного отсека (Central или Peripheral) | liter |
| Коэффициент исключения из первого порядка (ke) | 1/second |
| Константа Михаэлиса (Km) | milligram/liter |
| Максимальная скорость реакции, михаэлис-Ментен (Vm) | milligram/second |
| Зазор (Cl) | liter/second |
| Длительность поглощения (Tk0) | second |
| Скорость поглощения (ka) | 1/second |
Используйте опции настроек строения, чтобы повернуть преобразование модулей on или off. Для получения дополнительной информации см. Симуляцию модели.
Для получения дополнительной информации о размерном анализе для скорости реакции смотрите Как оцениваются скорости реакции.