Используйте дифференциальные уравнения, кинетику действующих масс или кинетику Михаэлис-Ментен для определения ферментативных реакций.
Простая модель для реакций, катализируемых ферментом, запускает субстрат S обратимое связывание с ферментом E. Часть субстрата в комплексе субстрат/фермент превращается в продукт P с релизом фермента.
Эта простая модель может быть определена
Дифференциальные уравнения скорости. См. Ферментативные реакции с дифференциальными уравнениями скорости.
Реакции с кинетикой действующих масс. См. Ферментативные реакции с Кинетикой действующих масс.
Реакции с Кинетикой Генри-Михаэлиса-Ментен. См. Ферментативные реакции с необратимой Кинетикой Генри-Михаэлиса-Ментен.
Реакции для механизма ферментативной реакции с одним субстратом (см. Простая модель для одно-субстратных катализируемых реакций) могут быть описаны дифференциальными уравнениями скорости. Вы можете ввести дифференциальные уравнения скорости в программное обеспечение в качестве правила скорости.
reactions: none
reaction rate: none
rate rules: dS/dt = k1r*ES - k1*S*E
dE/dt = k1r*ES + k2*ES - k1*S*E
dES/dt = k1*S*E - k1r*ES - k2*ES
dP/dt = k2*ES
species: S = 8 mole
E = 4 mole
ES = 0 mole
P = 0 mole
parameters: k1 = 2 1/(mole*second)
k1r = 1 1/second
k2 = 1.5 1/secondПомните, что правило скорости dS/dt = f(x) написано на SimBiology® оцените выражение правила как S = f(x). Для получения дополнительной информации о правилах скорости см. «Правила скорости».

Кроме того, можно удалить правило скорости для ES, добавить новый вид Etotal для общего количества фермента и добавить алгебраическое правило 0 = Etotal - E - ES, где начальные суммы для Etotal и E равны.
reactions: none
reaction rate: none
rate rules: dS/dt = k1r*ES - k1*S*E
dE/dt = k1r*ES + k2*ES - k1*S*E
dP/dt = k2*ES
algebraic rule: 0 = Etotal - E - ES
species: S = 8 mole
E = 4 mole
ES = 0 mole
P = 0 mole
Etotal = 4 mole
parameters: k1 = 2 1/(mole*second)
k1r = 1 1/second
k2 = 1.5 1/secondОпределение дифференциальных уравнений скорости для реакций в модели является длительным процессом. Лучшим способом является ввод реакций для одно-субстратного механизма ферментативной реакции непосредственно в программное обеспечение. Следующий пример использует модели ферментативно катализируемой реакции с кинетикой действующих масс. Для описания модели реакции смотрите Простую модель для Одно-субстратных катализируемых реакций.
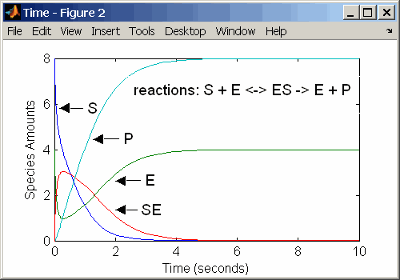
reaction: S + E -> ES
reaction rate: k1*S*E (binding)
reaction: ES -> S + E
reaction rate: k1r*ES (unbinding)
reaction: ES -> E + P
reaction rate: k2*ES (transformation)
species: S = 8 mole
E = 4 mole
ES = 0 mole
P = 0 mole
parameters: k1 = 2 1/(mole*second)
k1r = 1 1/second
k2 = 1.5 1/secondРезультаты для симуляции с использованием реакций идентичны результатам от использования дифференциальных уравнений скорости.
Представление реакции, катализируемой ферментом, с кинетикой действующих масс требует, чтобы вы знали константы скорости k1, k1r, и k2. Однако эти константы скорости редко сообщаются в литературе. Чаще всего можно задать константы скорости для кинетики Генри-Михаэлиса-Ментен с максимальной скоростью Vm=k2*E и постоянную Km = (k1r + k2)/k1. Скорость реакции для одно-субстратной ферментативной реакции, использующей Кинетику Генри-Михаэлиса-Ментен, приведена ниже. Для получения информации о модели смотрите Простая модель для Одно-субстратных катализируемых реакций.
В следующем примере моделируется реакция, катализируемая ферментом, с использованием кинетики Генри-Михаэлиса-Ментен с одной реакцией и уравнением скорости реакции. Введите реакцию, заданную ниже, в программное обеспечение и моделируйте.
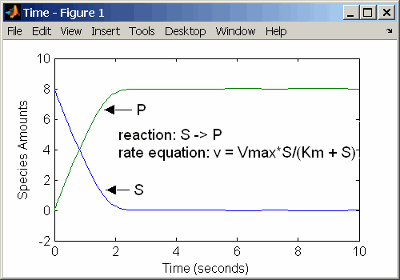
reaction: S -> P
reaction rate: Vmax*S/(Km + S)
species: S = 8 mole
P = 0 mole
parameters: Vmax = 6 mole/second
Km = 1.25 moleРезультаты показывают график, немного отличный от графика с помощью кинетики действующих масс. Различия связаны с допущениями, сделанными при выведении уравнения скорости Михаэлис-Ментен.