Этот пример показывает, как создать пользовательскую функцию и включить ее в симуляцию модели.
Этот пример предполагает, что вы имеете рабочие знания:
MATLAB® приложение
Создание и сохранение программ MATLAB
Этот пример использует модель, описанную в модели дрожжевого гетеротримерного цикла G-белка.
Эта таблица показывает реакции, используемые для моделирования цикла G-белка, и соответствующие параметры скорости (константы скорости) для каждой реакции. Для обратимых реакций сначала перечисляется параметр прямой скорости.
| Нет. | Имя | Реакция1 | Параметры скорости |
|---|---|---|---|
| 1 | Взаимодействие рецептор-лиганд | L + R <-> RL | kRL, kRLm |
| 2 | Гетеротримерное образование G-белка | Gd + Gbg -> G | kG1 |
| 3 | Активация G-белка | RL + G -> Ga + Gbg + RL | kGa |
| 4 | Синтез и деградация рецепторов | R <-> null | kRdo, kRs |
| 5 | Деградация рецептора-лиганда | RL -> null | kRD1 |
| 6 | Инактивация G-белка | Ga -> Gd | kGd |
| 1 Легенда видов: L = лиганд (альфа-фактор), R = альфа-фактор рецептор, Gd = неактивный G-альфа-ВВП, Gbg = свободные уровни G-бета: G-гамма комплекс, G = неактивный Gbg: Gd комплекс, Ga = активный G-альфа-ga | |||
Этот пример принимает, что:
Ингибитор (Inhib вид) замедляет инактивацию активного G-белка (реакция 6 выше, Ga –> Gd).
Изменение количества ингибитора (Inhib species) определяется в пользовательской функции, inhibvalex.
Ингибитор (Inhib вид) влияет на реакцию путем изменения величины параметра скорости kGd.
В этом примере показано, как создать и вызвать пользовательскую функцию в SimBiology® выражение. В частности, в нем показано, как использовать пользовательскую функцию в выражении правила.
Пользовательские функции можно использовать в:
Выражения скорости реакции (ReactionRate свойство)
Выражения правил (Rule свойство)
Выражения событий (EventFcns свойство или Trigger свойство)
Требования к использованию пользовательских функций в выражениях SimBiology:
Создайте пользовательскую функцию. Для получения дополнительной информации см. function.
Измените текущую папку на папку, содержащую пользовательский файл MATLAB. Сделайте это при помощи cd Команда или с помощью поля Текущая папка на панели инструментов рабочего стола MATLAB. Кроме того, добавьте папку, содержащую ваш файл, в путь поиска файлов. Сделайте это при помощи addpath или см. раздел «Изменение папок на Пути поиска файлов».
Вызовите пользовательскую функцию в выражении реакции, правила или события SimBiology.
Совет
Если ваше выражение скорости реакции не является непрерывным и дифференцируемым, см. Использование событий для устранения разрывов в выражениях правила и скорости реакции перед симуляцией модели.
Следующая процедура создает пользовательскую функцию, inhibvalex, что позволяет вам задать, как количество ингибитора изменяется с течением времени. Входами являются время, начальное количество ингибитора и параметр, который управляет количеством ингибитора. Выходом функции является количество ингибитора.
На рабочем столе MATLAB выберите File > New > Script, чтобы открыть РЕДАКТОРА MATLAB.
Скопируйте и вставьте следующее объявление функции:
% inhibvalex.m function Cp = inhibvalex(t, Cpo, kel) % This function takes the input arguments t, Cpo, and kel % and returns the value of the inhibitor Cp. % You can later specify the input arguments in a % SimBiology rule expression. % For example in the rule expression, specify: % t as time (a keyword recognized as simulation time), % Cpo as a parameter that represents the initial amount of inhibitor, % and kel as a parameter that governs the amount of inhibitor. if t < 400 Cp = Cpo*exp(-kel*(t)); else Cp = Cpo*exp(-kel*(t-400)); end
Сохраните файл (назовите файл inhibvalex.m) в директории, который находится в пути поиска файлов MATLAB или в директории, к которому вы можете получить доступ.
Если местоположение файла отсутствует в пути поиска файлов MATLAB, измените рабочую директорию на местоположение файла.
Загрузите gprotein пример проекта, который включает переменную m1, объект модели:
sbioloadproject gproteinThe m1 Объект модели появляется в Рабочем пространстве MATLAB.
Следующая процедура создает выражение правила, которое вызывает пользовательскую функцию, inhibvalex, и задает три входных значения для этой функции.
Добавьте повторное правило назначения к модели, которое задает три входных значения для пользовательской функции, inhibvalex:
rule1 = addrule(m1, 'Inhib = inhibvalex(time, Cpo, Kel)',... 'repeatedAssignment');
The time вход - ключевое слово SimBiology, распознаваемое как время симуляции
Создайте два параметра, используемых rule1 правило и присвоение им значений:
p1 = addparameter(m1, 'Cpo', 250); p2 = addparameter(m1, 'Kel', 0.01);
Создайте виды, используемые rule1 правило:
s1 = addspecies(m1.Compartments, 'Inhib');Значение параметра скорости kGd зависит от количества ингибитора, присутствующего в системе. Добавьте правило в модель, чтобы описать это действие, но сначала измените ConstantValue свойство параметра kGd так что это может быть изменено правилом.
Измените ConstantValue свойство kGd параметр в false.
p3 = sbioselect(m1, 'Type', 'parameter', 'Name', 'kGd'); p3.ConstantValue = false;
Добавьте повторное правило назначения к модели, чтобы определить, как kGd параметр зависит от Inhib вид.
rule2 = addrule(m1, 'kGd = 1/Inhib', 'repeatedAssignment');
Пользовательская функция, inhibvalex, вводит в модель разрыв, когда время = 400. Чтобы гарантировать точные результаты симуляции, добавьте событие в модель, чтобы сбросить решатель во время разрыва непрерывности. Установите событие на триггер во время разрыва (время = 400). Событие не должно изменять модель, поэтому создайте функцию события, которая умножает значение вида на 1.
addevent(m1, 'time>=400', 'G=1*G');
Настройте настройки симуляции (configset object) для m1 объект модели для регистрации всех состояний во время симуляции.
cs = getconfigset(m1);
cs.RuntimeOptions.StatesToLog = 'all';
Симулируйте модель.
simDataObj = sbiosimulate(m1);
Постройте график результатов.
sbioplot(simDataObj);
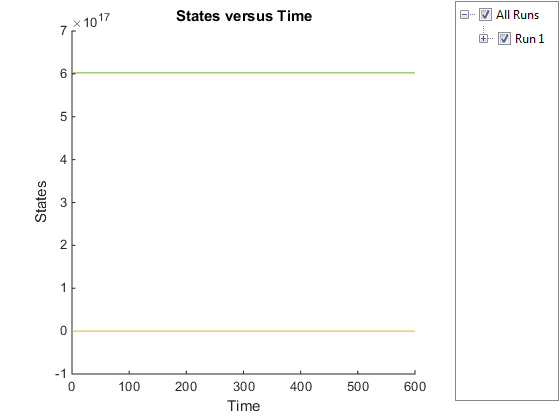
График не показывает интересующие виды из-за широкой области значений количеств/концентраций видов.
Постройте график только интересующих видов. Ga.
GaSimDataObj = selectbyname(simDataObj,'Ga');
sbioplot(GaSimDataObj);
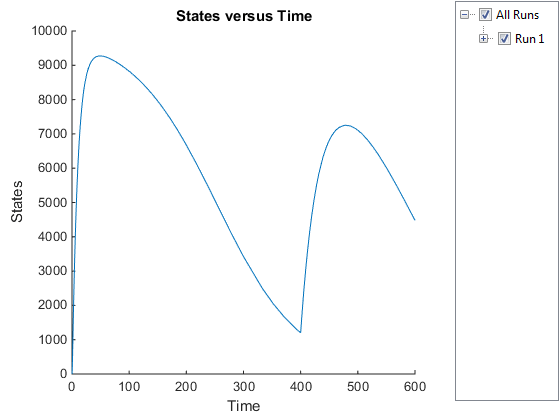
Заметьте изменение профиля видов Ga в момент времени = 400 секунд (время симуляции). Это время, когда количество ингибитора изменяется, чтобы отразить повторное добавление ингибитора к модели.
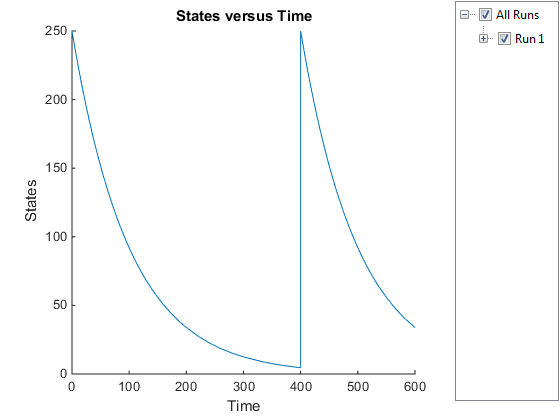
Постройте график только ингибитора (Inhib вид).
InhibSimDataObj = selectbyname(simDataObj,'Inhib');
sbioplot(InhibSimDataObj)