Можно использовать функции командной строки, приложение Wavelet Analyzer, или можно смешать обе для работы с вейвлетом деревьями пакетов (объекты WPTREE). Самые полезные команды:
Некоторые из этих функций можно увидеть в следующих примерах.
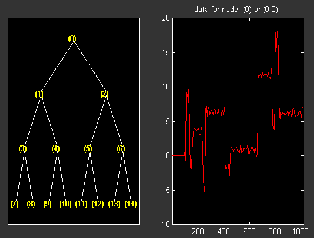
load noisbump x = noisbump; t = wpdec(x,3,'db2'); fig = plot(t);
Щелкните на узле 7.
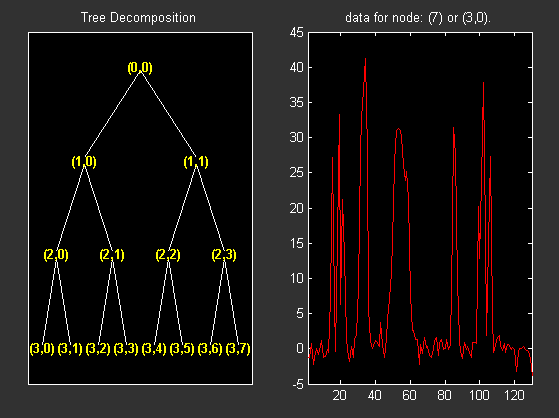
Измените Действие узла с Visualize на Split-Merge и объедините второй узел.
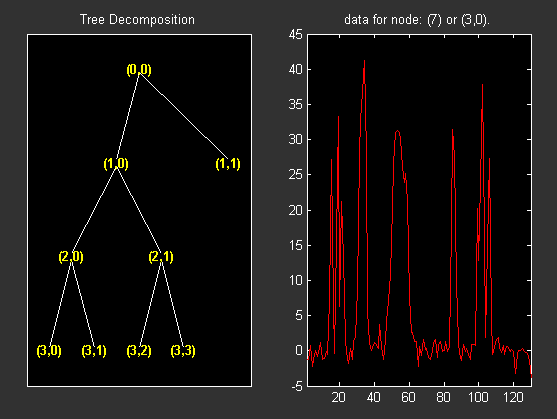
% From the command line, you can get the new tree. newt = plot(t,'read',fig); % The first argument of the plot function in the last command % is dummy. Then the general syntax is: % newt = plot(DUMMY,'read',fig); % where DUMMY is any object parented by an NTREE object. % DUMMY can be any object constructor name, which returns % an object parented by an NTREE object. For example: % newt = plot(ntree,'read',fig); % newt = plot(dtree,'read',fig); % newt = plot(wptree,'read',fig); % From the command line you can modify the new tree, % then plot it. newt = wpjoin(newt,3); fig2 = plot(newt); % Change Node Label from Depth_position to Index and % click the node (3). You get the following figure.
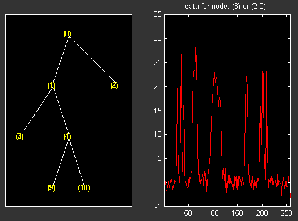
% Using plot(newt,fig), the plot is done in the figure fig, % which already contains a tree object. % You can see the colored wavelet packets coefficients using % from the command line, the wpviewcf function (type help % wpviewcf for more information). wpviewcf(newt,1) % You get the following plot, which contains the terminal nodes % colored coefficients.
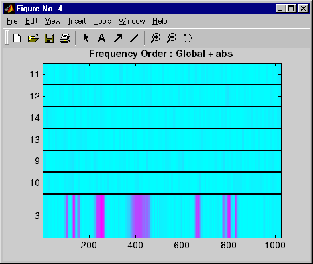
load noisbump x = noisbump; t = wpdec(x,3,'db2'); fig = drawtree(t); % The last command creates a GUI. % The same GUI can be obtained using waveletAnalyzer and: % - clicking the Wavelet Packet 1-D button, % - loading the signal noisbump, % - choosing the level and the wavelet % - clicking the decomposition button. % You get the following figure.
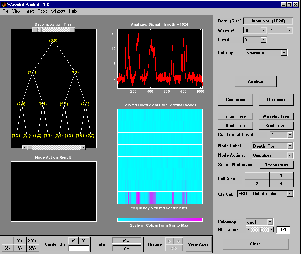
% From the app, you can modify the tree. % For example, change Node label from Depth_Position to Index, % change Node Action from Visualize to Split_Merge and % merge the node 2. % You get the following figure.
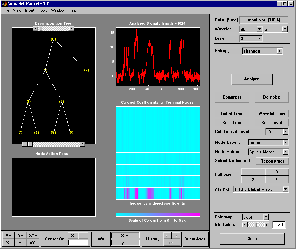
% From the command line, you can get the new tree. newt = readtree(fig); % From the command line you can modify the new tree; % then plot it in the same figure. newt = wpjoin(newt,3); drawtree(newt,fig);
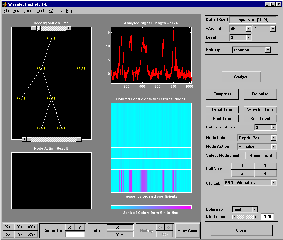
Можно чередовать предыдущие команды. Графический интерфейс пользователя, сопоставленная с plot команда проще и быстрее, но больше действий и информации доступно с помощью фрагментов приложения Wavelet Analyzer, связанных с вейвлет.
Методы, связанные с объектами WPTREE, позволяют вам совершать более сложные действия.
А именно, использование read и write методы, можно изменить коэффициенты терминального узла.
Проиллюстрируем эту точку следующим «забавным» примером.
load gatlin2 t = wpdec2(X,1,'haar'); plot(t); % Change Node Label from Depth_position to Index and % click the node (0). You get the following figure.
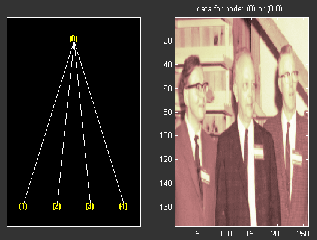
% Now modify the coefficients of the four terminal nodes. newt = t; NBcols = 40; for node = 1:4 cfs = read(t,'data',node); tmp = cfs(1:end,1:NBcols); cfs(1:end,1:NBcols) = cfs(1:end,end-NBcols+1:end); cfs(1:end,end-NBcols+1:end) = tmp; newt = write(newt,'data',node,cfs); end plot(newt) % Change Node Label from Depth_position to Index and % click on the node (0). You get the following figure.
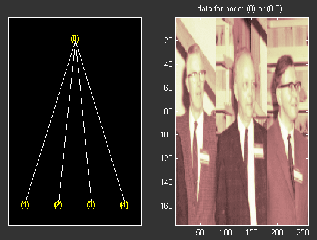
Можно использовать этот метод для более полезной цели. Давайте посмотрим обличающий пример.
load noisbloc x = noisbloc; t = wpdec(x,3,'sym4'); plot(t); % Change Node Label from Depth_position to Index and % click the node (0). You get the following plot.
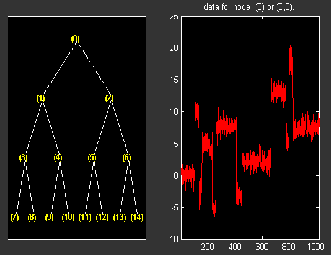
% Global thresholding.
t1 = t;
sorh = 'h';
thr = wthrmngr('wp1ddenoGBL','penalhi',t);
cfs = read(t,'data');
cfs = wthresh(cfs,sorh,thr);
t1 = write(t1,'data',cfs);
plot(t1)
% Change Node Label from Depth_position to Index and
% click the node (0). You get the following plot.
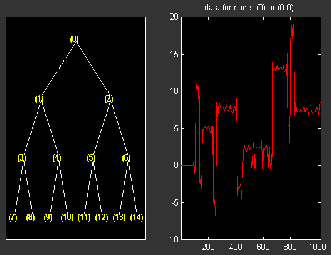
% Node by node thresholding.
t2 = t;
sorh = 's';
thr(1) = wthrmngr('wp1ddenoGBL','penalhi',t);
thr(2) = wthrmngr('wp1ddenoGBL','sqtwologswn',t);
tn = leaves(t);
for k=1:length(tn)
node = tn(k);
cfs = read(t,'data',node);
numthr = rem(node,2)+1;
cfs = wthresh(cfs,sorh,thr(numthr));
t2 = write(t2,'data',node,cfs);
end
plot(t2)
% Change Node Label from Depth_position to Index and
% click the node (0). You get the following plot.