Создайте значение по сравнению с отношением экспрессии гена (изменение сгиба) график рассеивания микроданных массива
mavolcanoplot(DataX, DataY,
PValues)
SigStructure = mavolcanoplot(DataX, DataY, PValues)
... mavolcanoplot(..., 'Labels', LabelsValue,
...)
... mavolcanoplot(..., 'LogTrans', LogTransValue,
...)
... mavolcanoplot(..., 'PCutoff', PCutoffValue, ...)
... mavolcanoplot(..., 'Foldchange', FoldchangeValue,
...)
... mavolcanoplot(..., 'PlotOnly', PlotOnlyValue,
...)
DataX, DataY | Объект DataMatrix, матрица или вектор из значений экспрессии гена от одного экспериментального условия. Если объект DataMatrix или матрица, каждая строка является геном, каждый столбец является выборкой, и среднее значение выражения вычисляется для каждого гена. Примечание Если значения в |
PValues | Любое из следующего:
|
LabelsValue | Массив ячеек из символьных векторов или вектор строки, содержащий метки (обычно названия генов или тестовые идентификаторы набора) для данных. После создания графика можно кликнуть по точке данных, чтобы отобразить метку, сопоставленную с ним. Если вы не обеспечиваете |
LogTransValue | Свойство управлять преобразованием данных в |
PCutoffValue | Позволяет вам задать p-значение сокращения, чтобы задать точки данных, которые являются статистически значительными. Это значение отображено графически как горизонтальная линия на графике. Значением по умолчанию является Примечание Можно также изменить сокращение p-значения в интерактивном режиме после создания графика. |
FoldchangeValue | Позволяет вам указать, что сгиб отношения изменяется, чтобы задать точки данных, которые дифференцированно описываются. Значением по умолчанию является Примечание Можно также изменить изменение сгиба в интерактивном режиме после создания графика. |
PlotOnlyValue | Управляет отображением графика вулкана без компонентов пользовательского интерфейса. Выбором является Примечание Если вы устанавливаете |
SigStructure | Структура, содержащая информацию для генов, которые считаются обоими статистически значительными (выше сокращения p-значения) и значительно дифференцированно описываются (за пределами значений изменения сгиба). Поля описаны ниже. |
mavolcanoplot( создает график рассеивания данных об экспрессии гена, строя значение по сравнению с изменением сгиба отношений экспрессии гена двух наборов данных, DataX, DataY,
PValues)DataX и DataY. Это строит значение как –log10 (p-значение) от входа, PValues. DataX и DataY могут быть векторы, матрицы или объекты DataMatrix. PValues объект DataMatrix или вектор-столбец.
SigStructure = mavolcanoplot(DataX, DataY, PValues)SigStructure сортируются по p-значению и включают:
Name
PCutoff
FCThreshold
GeneLabels
PValues
FoldChanges
Примечание
Поля PValues и FoldChanges будут или векторы или объекты DataMatrix в зависимости от типа входа PValues.
... mavolcanoplot (..., ' задает дополнительные свойства, которые используют имя свойства / пары значения в любом порядке. Это имя свойства / пары значения следующие:PropertyName', PropertyValue, ...)
... mavolcanoplot(..., 'Labels', позволяет вам обеспечить массив ячеек из символьных векторов или вектор строки, содержащий метки (обычно названия генов, или зондируйте идентификаторы набора) для данных. После создания графика можно кликнуть по точке данных, чтобы отобразить метку, сопоставленную с ним. Если вы не обеспечиваете LabelsValue,
...)LabelsValue, точки данных помечены номерами строк от DataX и DataY.
... mavolcanoplot(..., 'LogTrans', управляет преобразованием данных из LogTransValue,
...)DataX и DataY к шкале log2. Когда LogTransValue true, mavolcanoplot преобразует данные от естественного до шкалы log2. Значением по умолчанию является false, который принимает, что данные уже являются log2 шкалой.
... mavolcanoplot(..., 'PCutoff', позволяет вам задать сокращение p-значения, чтобы задать точки данных, которые являются статистически значительными. Это значение отображается графически как горизонтальная линия на графике. Значением по умолчанию является PCutoffValue, ...)0.05, который эквивалентен 1,3010 на –log10 (p-значение) шкала.
Примечание
Можно также изменить сокращение p-значения в интерактивном режиме после создания графика.
... mavolcanoplot(..., 'Foldchange', позволяет вам указать, что сгиб отношения изменяется, чтобы задать точки данных, которые дифференцированно описываются. Сверните отображение изменений графически как две вертикальных линии на графике. Значением по умолчанию является FoldchangeValue,
...)2, который соответствует отношению 1 и –1 на log2 (отношение) шкала.
Примечание
Можно также изменить изменение сгиба в интерактивном режиме после создания графика.
... mavolcanoplot(..., 'PlotOnly', управляет отображением графика вулкана без компонентов пользовательского интерфейса. Выбором является PlotOnlyValue,
...)true или false (значение по умолчанию).
Примечание
Если вы устанавливаете 'PlotOnly' свойство к true, можно все еще отобразить метки для точек данных путем нажатия на точку данных, и можно все еще настроить вертикальные линии изменения сгиба и горизонтальную линию сокращения p-значения перетаскиванием нажатия кнопки линии.
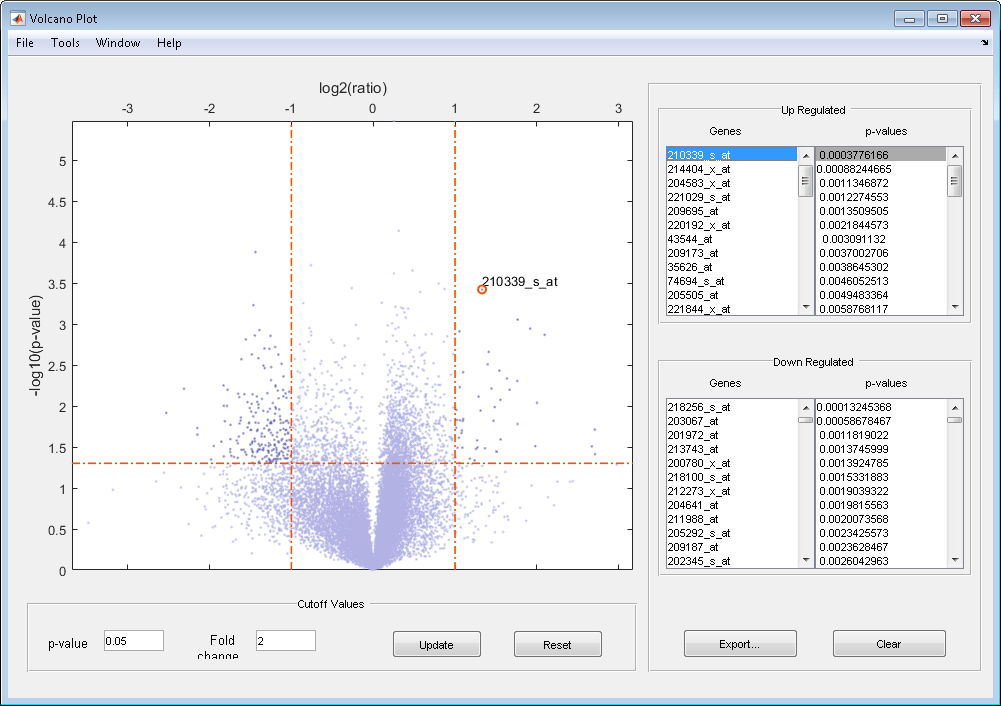
График вулкана отображает следующее:
– log10 (p-значение) по сравнению с log2 (отношение) график рассеивания генов
Два вертикальных сгиба изменяют линии на уровне изменения сгиба 2, который соответствует отношению 1 и –1 на log2 (отношение) шкала. (Линии будут на различных уровнях изменения сгиба, если вы использовали 'Foldchange' свойство.
Одна горизонтальная линия на 0,05 уровнях p-значения, которые эквивалентны 1,3010 на –log10 (p-значение) шкала. (Линия будет на различном уровне p-значения, если вы использовали 'PCutoff' свойство.
После того, как вы отобразите график рассеивания вулкана, вы можете в интерактивном режиме:
Настройте вертикальные линии изменения сгиба перетаскиванием нажатия кнопки одна линия или ввод значения в текстовом поле Fold Change.
Настройте горизонтальную линию сокращения p-значения перетаскиванием нажатия кнопки или вводом значения в текстовом поле p-value Cutoff.
Отобразите метки для точек данных путем нажатия на точку данных.
Выберите ген из Up Regulated или списка Down Regulated, чтобы подсветить соответствующую точку данных в графике. Нажмите и содержите Ctrl или Shift, чтобы выбрать несколько генов.
Масштабируйте график путем выбора Tools > Zoom In или Tools > Zoom Out.
Просмотрите списки значительно отрегулированных и вниз отрегулированных генов и их связанных p-значений, и опционально, экспортируйте метки, p-значения и изменения сгиба в структуре в MATLAB® Рабочая область путем нажатия на Export.
Загрузите MAT-файл, включенный с программным обеспечением Bioinformatics Toolbox™, которое содержит Affymetrix® переменные данных, включая dependentData и independentData, две матрицы значений экспрессии гена от двух экспериментальных условий.
load prostatecancerexpdataИспользуйте mattest функция, чтобы вычислить p-значения для значений экспрессии гена в этих двух матрицах.
pvalues = mattest(dependentData, independentData);
Используя эти две матрицы, pvalues вычисленный mattest, и probesetIDs вектор-столбец меток, если, используйте mavolcanoplot создать значение по сравнению с графиком рассеивания отношения экспрессии гена микроданных массива от двух экспериментальных условий.
mavolcanoplot(dependentData, independentData, pvalues,... 'Labels', probesetIDs)
Просмотрите график вулкана без компонентов пользовательского интерфейса.
mavolcanoplot(dependentData, independentData, pvalues,... 'Labels', probesetIDs,'Plotonly', true)
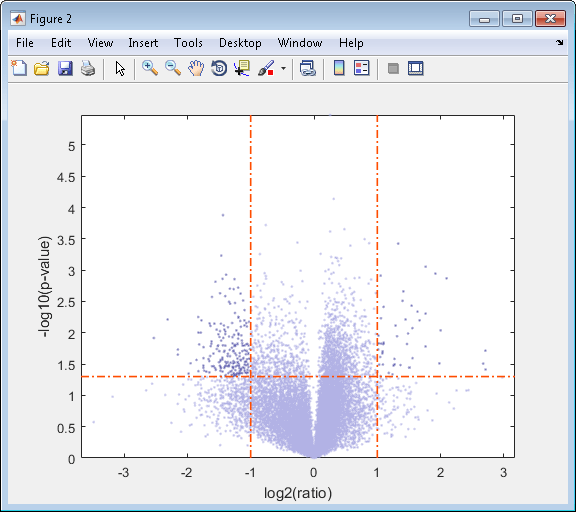
prostatecancerexpdata.mat файл, используемый в предыдущем примере, содержит данные из Лучшего и др., 2005.
[1] Цуй, X., Черчилль, G.A. (2003). Статистические тесты для дифференциального выражения в экспериментах комплементарной ДНК микромассивов. Биология генома 4, 210.
[2] Лучше всего, C.J.M., Гиллеспи, J.W., И, Y., Chandramouli, G.V.R., Perlmutter, M.A., Gathright, Y., Эриксон, H.S., Георгевич, L., Tangrea, M.A., Duray, P.H., Гонсалес, S., Веласко, A., Linehan, W.M., Matusik, R.J., Цена, D.K., Figg, W.D., Emmert-маркер, M.R., и Chuaqui, R.F. (2005). Молекулярные изменения при первичном раке простаты после терапии абляции андрогена. Клинические Исследования рака 11, 6823–6834.
maboxplot | maimage | mainvarsetnorm | mairplot | maloglog | malowess | manorm | mapcaplot | mattest