Выполните GC Устойчивое Среднее значение Мультимассивов (GCRMA) фоновая корректировка на данных тестового уровня Affymetrix микромассивов с помощью информации о последовательности
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
[PMMatrix_Adj, nsbStruct]
= gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
... = gcrmabackadj( ...'OpticalCorr', OpticalCorrValue,
...)
... = gcrmabackadj( ...'CorrConst', CorrConstValue,
...)
... = gcrmabackadj( ...'Method', MethodValue,
...)
... = gcrmabackadj( ...'TuningParam', TuningParamValue,
...)
... = gcrmabackadj( ...'AddVariance', AddVarianceValue,
...)
... = gcrmabackadj( ...'GSBCorr', GSBCorrValue,
...)
... = gcrmabackadj( ...'Showplot', ShowplotValue,
...)
... = gcrmabackadj( ...'Verbose', VerboseValue,
...)
PMMatrix | Матрица значений интенсивности, где каждая строка соответствует идеальной паре (PM), зонд и каждый столбец соответствуют файлу Affymetrix® CEL. (Каждый файл CEL сгенерирован от отдельного чипа. Все микросхемы должны иметь тот же тип.) СоветМожно использовать матрицу
|
MMMatrix | Матрица значений интенсивности, где каждая строка соответствует несоответствию (MM) зонд и каждый столбец, соответствует файлу Affymetrix CEL. (Каждый файл CEL сгенерирован от отдельного чипа. Все микросхемы должны иметь тот же тип.) СоветМожно использовать матрицу
|
AffinPM | Вектор-столбец PM зондирует сродство, такой, как возвращено функцией affyprobeaffinities. Каждая строка соответствует зонду. |
AffinMM | Вектор-столбец MM зондирует сродство, такой, как возвращено функцией affyprobeaffinities. Каждая строка соответствует зонду. |
OpticalCorrValue | Управляет использованием оптического фонового исправления на премьер-министре и значениях интенсивности зонда MM в PMMatrix и MMMatrix. Выбором является true (значение по умолчанию) или false. |
CorrConstValue | Значение, которое задает постоянную корреляцию, ро, для логарифмической фоновой интенсивности для каждой пары зонда PM/MM. Выбором является любое значение ≥ 0 и ≤ 1. Значением по умолчанию является 0.7. |
MethodValue | Вектор символов или строка, которая задает метод, чтобы оценить сигнал. Выбором является 'MLE', более быстрый, оперативный Оценочный метод Наибольшего правдоподобия, или 'EB', более медленный, более формальный, эмпирический метод Бейеса. Значением по умолчанию является 'MLE'. |
TuningParamValue | Значение, которое задает настраивающийся параметр, используемый оценочным методом. Этот настраивающий параметр устанавливает нижнюю границу значений сигналов с положительной вероятностью. Выбором является положительное значение. Значением по умолчанию является 5 (MLE) или 0.5 (EB).СоветДля получения информации об определении установки для этого параметра смотрите Ву и др., 2004. |
AddVarianceValue | Средства управления, добавляется ли отклонение сигнала к функции веса для сглаживания низкого ребра сигнала. Выбором является true или false (значение по умолчанию). |
GSBCorrValue | Задает, выполнить ли исправление специфичной для гена привязки (GSB) с помощью тестовых данных о сродстве. Выбором является true (значение по умолчанию) или false. Если нет никакой тестовой информации о сродстве, это свойство проигнорировано. |
ShowplotValue | Управляет отображением графика, показывающего log2 тестовых значений интенсивности из заданного столбца (чип) в MMMatrix, по сравнению с тестовым сродством в AffinMM. Выбором является true, false или I, целое число, задающее столбец в MMMatrix. Если установлено в true, первый столбец в MMMatrix построен. Значение по умолчанию:
|
VerboseValue | Управляет отображением отчета о выполнении работ, показывающего количество каждого чипа, когда это завершается. Выбором является true (значение по умолчанию) или false. |
PMMatrix_Adj | Матрица фона настроила PM (идеальная пара) значения интенсивности. |
nsbStruct | Структура, содержащая неопределенные обязательные фоновые параметры, оцененные от интенсивности и сродства зондов на массиве Affymetrix GeneChip®.
|
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)PMMatrix_Adj, матрица фона настроила PM (идеальная пара) значения интенсивности.
Если AffinPM и данные AffinMM не доступны, можно все еще использовать функцию gcrmabackadj путем ввода векторов пустого столбца для обоих из этих входных параметров в синтаксисе.
[ возвращает PMMatrix_Adj, nsbStruct]
= gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)nsbStruct, структура, содержащая неопределенные обязательные фоновые параметры, оцененные от интенсивности и сродства зондов на массиве Affymetrix GeneChip. nsbStruct включает следующие поля:
sigma
mu_pm
mu_mm
... = gcrmabackadj( ...'PropertyName', PropertyValue, ...) gcrmabackadj с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
... = gcrmabackadj( ...'OpticalCorr', управляет использованием оптического фонового исправления на премьер-министре и значениях интенсивности зонда MM в OpticalCorrValue,
...)PMMatrix и MMMatrix. Выбором является true (значение по умолчанию) или false.
... = gcrmabackadj( ...'CorrConst', задает постоянную корреляцию, ро, для логарифмической фоновой интенсивности для каждой пары зонда PM/MM. Выбором является любое значение CorrConstValue,
...)≥ 0 и ≤ 1. Значением по умолчанию является 0.7.
... = gcrmabackadj( ...'Method', задает метод, чтобы оценить сигнал. Выбором является MethodValue,
...)MLE, более быстрый, оперативный Оценочный метод Наибольшего правдоподобия, или EB, более медленный, более формальный, эмпирический метод Бейеса. Значением по умолчанию является MLE.
... = gcrmabackadj( ...'TuningParam', задает настраивающийся параметр, используемый оценочным методом. Этот настраивающий параметр устанавливает нижнюю границу значений сигналов с положительной вероятностью. Выбором является положительное значение. Значением по умолчанию является TuningParamValue,
...)5 (MLE) или 0.5 (EB).
Для получения информации об определении установки для этого параметра смотрите Ву и др., 2004.
... = gcrmabackadj( ...'AddVariance', средства управления, добавляется ли отклонение сигнала к функции веса для сглаживания низкого ребра сигнала. Выбором является AddVarianceValue,
...)true или false (значение по умолчанию).
... = gcrmabackadj( ...'GSBCorr', задает, выполнить ли исправление гена определенной привязки (GSB) с помощью тестовых данных о сродстве. Выбором является GSBCorrValue,
...)true (значение по умолчанию) или false. Если нет никакой тестовой информации о сродстве, это свойство проигнорировано.
... = gcrmabackadj( ...'Showplot', управляет отображением графика, показывающего log2 тестовых значений интенсивности из заданного столбца (чип) в ShowplotValue,
...)MMMatrix, по сравнению с тестовым сродством в AffinMM. Выбором является true, false или I, целое число, задающее столбец в MMMatrix. Если установлено в true, первый столбец в MMMatrix построен. Значение по умолчанию:
ложь Когда возвращаемые значения заданы.
tRUE Когда возвращаемые значения не заданы.
... = gcrmabackadj( ...'Verbose', управляет отображением отчета о выполнении работ, показывающего количество каждого чипа, когда это завершается. Выбором является VerboseValue,
...)true (значение по умолчанию) или false.
Загрузите MAT-файл, включенный с программным обеспечением Bioinformatics Toolbox™, которое содержит данные Affymetrix из исследования рака простаты. Переменные в MAT-файле включают seqMatrix, матрицу, содержащую информацию о последовательности для зондов PM, pmMatrix и mmMatrix, матриц, содержащих PM и значения интенсивности зонда MM и probeIndices, вектор-столбец, содержащий информацию об индексации зонда.
load prostatecancerrawdataВычислите PM Affymetrix и сродство зонда MM от их последовательностей и интенсивности зонда MM.
[apm, amm] = affyprobeaffinities(seqMatrix, mmMatrix(:,1),... 'ProbeIndices', probeIndices);
Выполните фоновую корректировку GCRMA на данных тестового уровня Affymetrix микромассивов, создавание матрицы фона настроило значения интенсивности PM. Кроме того, отобразите график, показывающий log2 тестовых значений интенсивности из столбца 3 (чип 3) в mmMatrix, по сравнению с тестовым сродством в amm.
pms_adj = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'showplot', 3);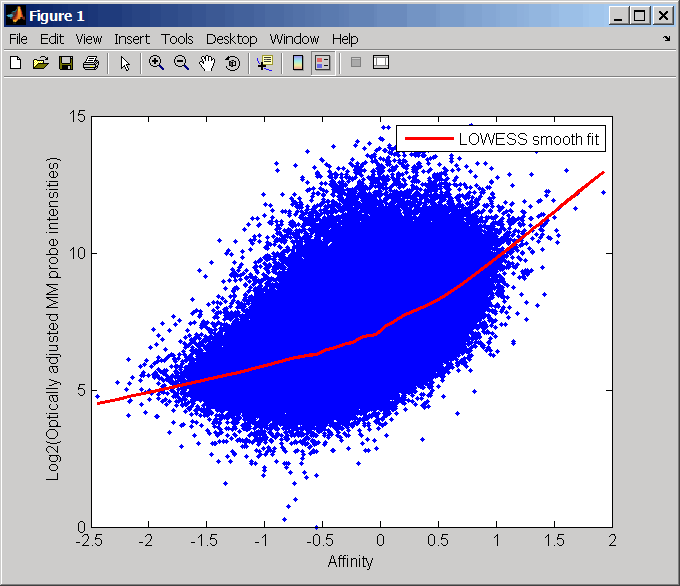
Выполните фоновую корректировку GCRMA снова, с помощью более медленного, более формального, эмпирического метода Бейеса.
pms_adj2 = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'method', 'EB');
Файл prostatecancerrawdata.mat, используемый в этом примере, содержит данные из Лучшего и др., 2005.
[1] Ву, Z., Irizarry, R.A., джентльмен, Р., Мурильо, F.M., и Спенсер, F. (2004). Основанная на модели фоновая корректировка к массивам выражения олигонуклеотида. Журнал американской статистической ассоциации 99 (468), 909–917.
[2] Ву, Z., и Irizarry, R.A. (2005). Стохастические модели, вдохновленные теорией гибридизации для коротких массивов олигонуклеотида. Продолжения RECOMB 2004. J Comput Biol. 12 (6), 882–93.
[3] Ву, Z., и Irizarry, R.A. (2005). Статистическая среда для анализа данных Тестового Уровня микромассивов. Университет Джонса Хопкинса, рабочие документы биостатистики 73.
[4] Ву, Z., и Irizarry, R.A. (2003). Основанная на модели фоновая корректировка к массивам выражения олигонуклеотида. Семинар RSS по экспрессии гена, Уаю, Англия, http://biosun01.biostat.jhsph.edu/%7Eririzarr/Talks/gctalk.pdf.
[5] Абд Рэббо, N.A., и Barakat, H.M. (1979). Проблемы оценки в двумерном логарифмически нормальном распределении. Индийский J. Чистая прикладная математика 10 (7), 815–825.
[6] Лучше всего, C.J.M., Гиллеспи, J.W., И, Y., Chandramouli, G.V.R., Perlmutter, M.A., Gathright, Y., Эриксон, H.S., Георгевич, L., Tangrea, M.A., Duray, P.H., Гонсалес, S., Веласко, A., Linehan, W.M., Matusik, R.J., Цена, D.K., Figg, W.D., Emmert-маркер, M.R., и Chuaqui, R.F. (2005). Молекулярные изменения при первичном раке простаты после терапии абляции андрогена. Клинические Исследования рака 11, 6823–6834.
affygcrma | affyprobeseqread | affyread | celintensityread | probelibraryinfo