Sequence Viewer интегрирует многие функции последовательности в тулбоксе Bioinformatics Toolbox™. Вместо того, чтобы ввести команды в Командном окне MATLAB®, можно выбрать и ввести опции с помощью приложения.
Первый шаг при анализе нуклеотида или последовательности аминокислот должен импортировать информацию о последовательности в среду MATLAB. Sequence Viewer может соединиться с базами данных Web, такими как NCBI и EMBL и считать информации в среду MATLAB.
Следующая процедура иллюстрирует, как получить информацию о последовательности из базы данных NCBI в сети. Этот пример использует инвентарный номер GenBank® NM_000520, который является человеческим геном HEXA, который сопоставлен с болезнью Тея-Сакса.
Данные в общедоступных репозиториях часто курируются и обновляются; поэтому, результаты этого примера могут немного отличаться, когда вы используете актуальные последовательности.
В Окне Команды MATLAB ввести
seqviewer
Также нажмите Sequence Viewer на вкладке Apps.
Sequence Viewer открывается без загруженной последовательности. Заметьте, что панели направо и нижняя часть являются пробелом.
Чтобы получить последовательность из базы данных NCBI, выберите File > Download Sequence from > NCBI.
Диалоговое окно Download Sequence from NCBI открывается.
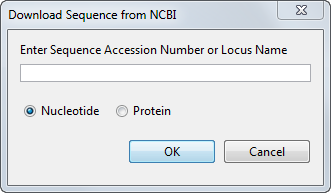
В поле Enter Sequence введите инвентарный номер для записи базы данных NCBI, например, NM_000520. Кликните по переключателю Nucleotide, и затем нажмите OK.
Программное обеспечение MATLAB получает доступ к базе данных NCBI в сети, информация о последовательности нуклеотида загрузок для инвентарного номера вы вошли и вычисляете некоторую базовую статистику.
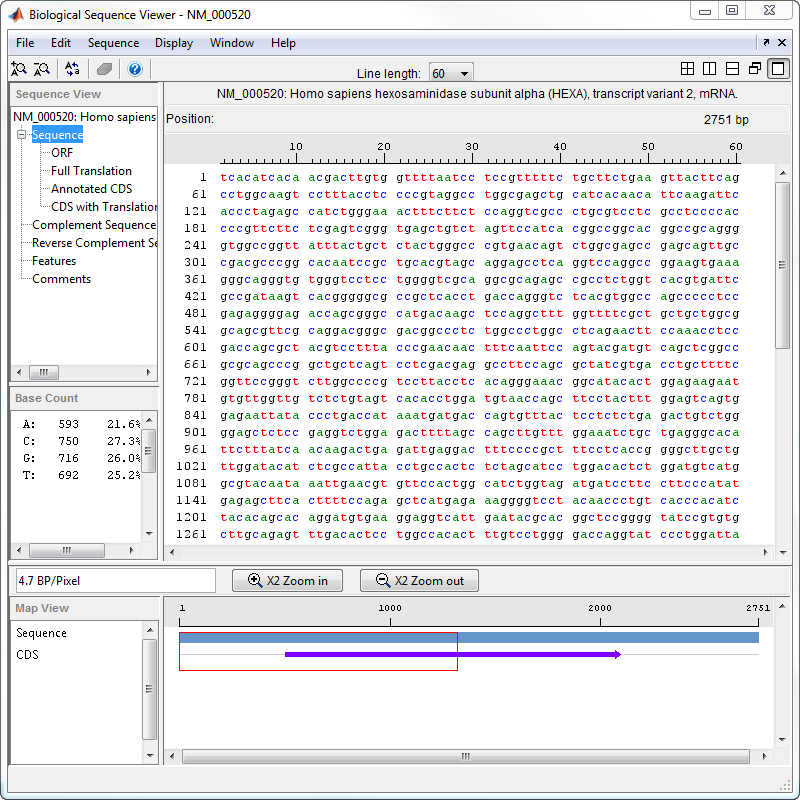
После того, как вы импортируете последовательность в приложение Sequence Viewer, можно считать информацию, хранившую с последовательностью, или можно просмотреть графические представления для ORFs и CDSs.
В дереве левой панели нажмите Comments. Правая панель отображает общую информацию о последовательности.
Теперь нажмите Features. Правая панель отображает информацию о функции NCBI, включая индексы для гена и любых последовательностей CDS.
Нажмите ORF, чтобы показать результаты поиска для ORFs в этих шести рамках считывания.
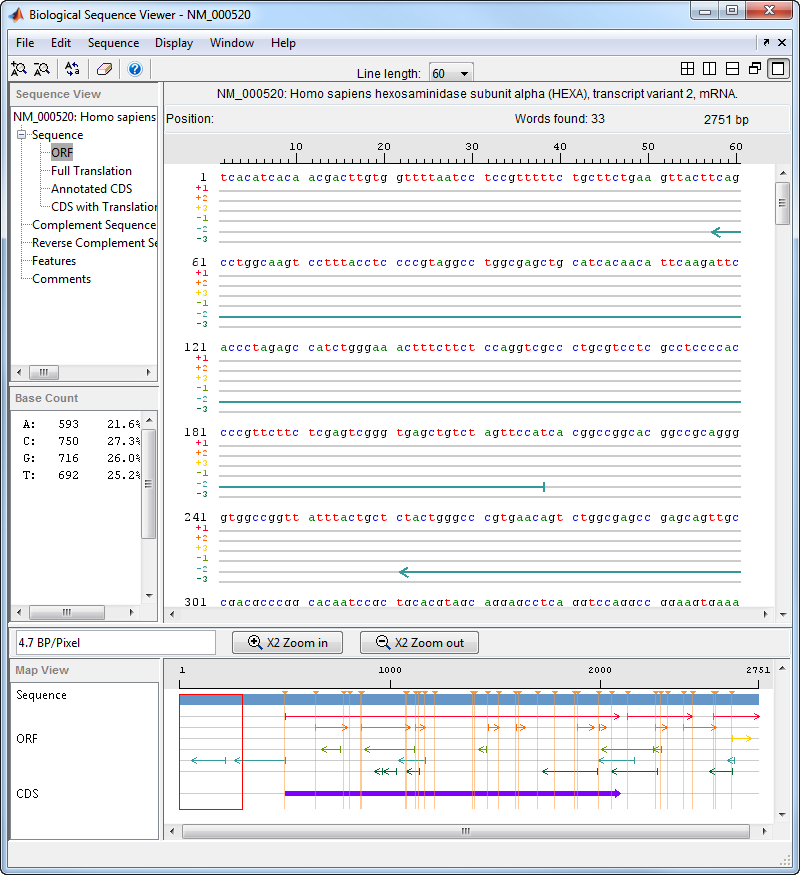
Нажмите Annotated CDS, чтобы показать часть кодирования белка последовательности нуклеотида.
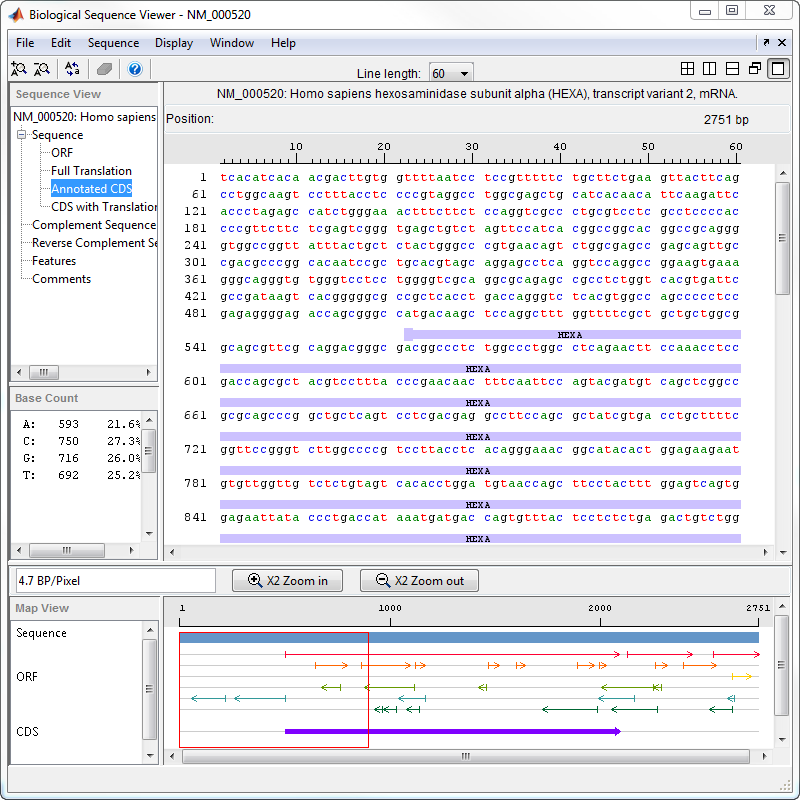
Можно также искать характеристические слова или шаблоны последовательности с помощью регулярных выражений. Можно ввести нуклеотид IUB/IUPAC и символы аминокислоты, которые автоматически преобразованы в соответствующие нуклеотиды и аминокислоты соответственно. Для получения дополнительной информации о том, как символы интерпретированы, см. Таблицы преобразования Преобразования и Аминокислоты Нуклеотида seq2regexp. Например, если вы ищете слово 'TAR' с установленным флажком Regular Expression, приложение подсвечивает все случаи 'TAA' и 'TAG' в последовательности начиная с R = [AG].
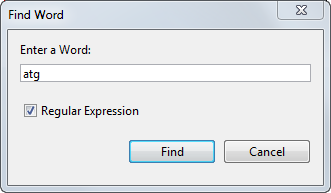
Выберите Sequence > Find Word.
В диалоговом окне Find Word введите слово последовательности или шаблон, например, atg, и затем нажмите Find.
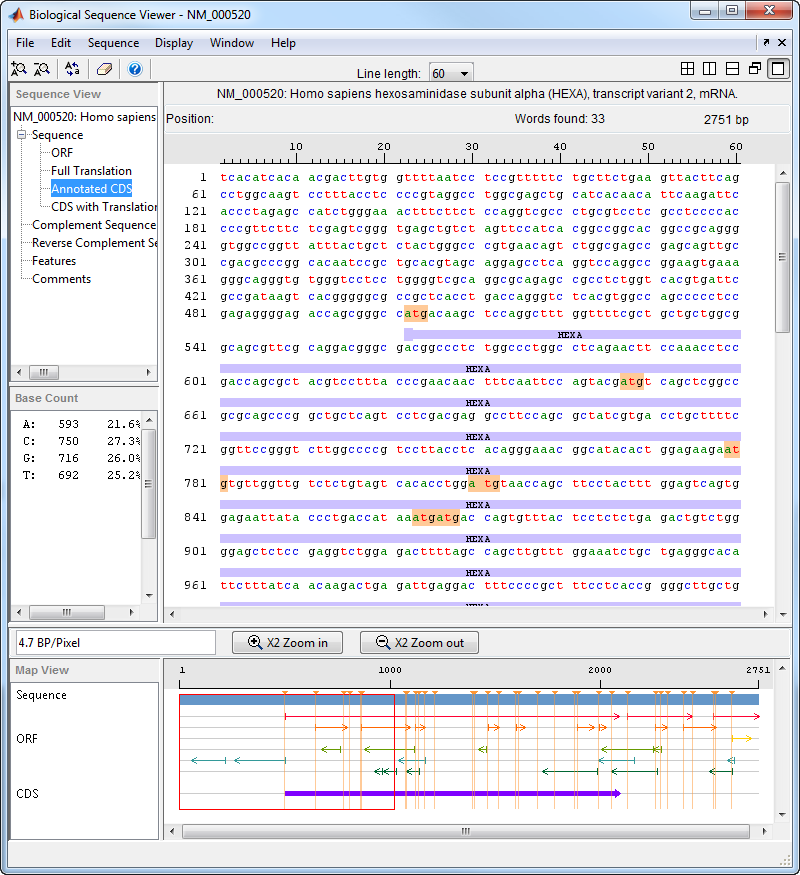
Sequence Viewer ищет и отображает местоположение выбранного слова.
Очистите отображение путем нажатия на Clear Word Selection button![]() на панели инструментов.
на панели инструментов.
Следующая процедура иллюстрирует, как идентифицировать часть кодирования белка последовательности нуклеотида и скопировать ее в новое представление. Идентификация разделов кодирования последовательности нуклеотида является общей задачей биоинформатики. После определения местоположения части кодирования последовательности можно скопировать его в новое представление, перевести его в последовательность аминокислот и продолжить анализ.
На левой панели нажмите ORF.
Sequence Viewer отображает ORFs для этих шести рамок считывания в нижней правой панели. Наведите курсор на кадр, чтобы отобразить информацию об этом.
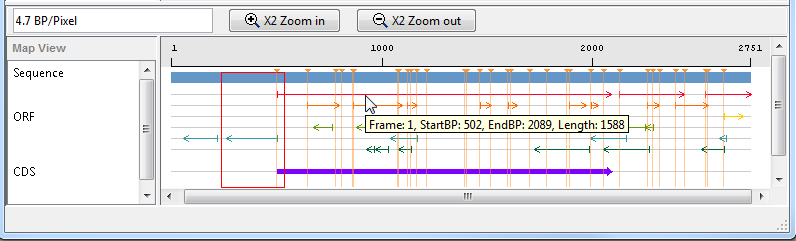
Кликните по самому длинному ORF на рамке считывания 2.
ORF подсвечен, чтобы указать на часть последовательности, которая выбрана.
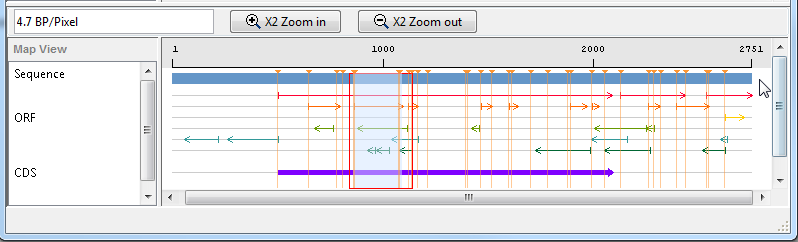
Щелкните правой кнопкой по выбранному ORF и затем выберите Export to Workspace. В диалоговом окне Export to MATLAB Workspace введите имя переменной, например, NM_000520_ORF_2, затем нажмите Export.
Переменная NM_000520_ORF_2 добавляется к рабочему пространству MATLAB.
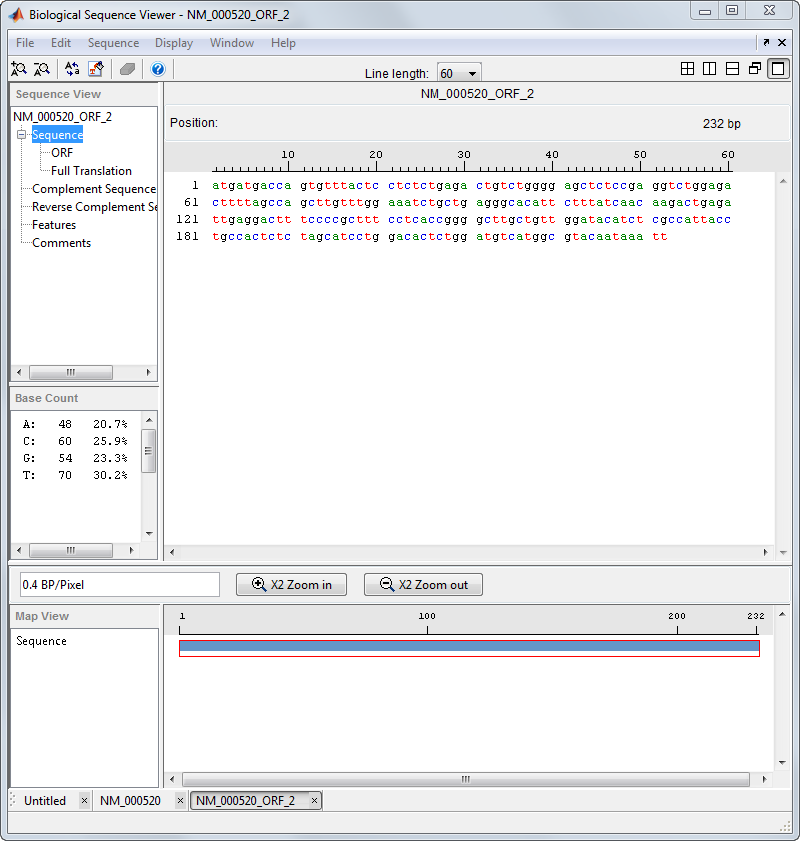
Выберите File > Import from Workspace. Введите имя переменной с экспортируемым ORF, например, NM_000520_ORF_2, и затем нажмите Import.
Sequence Viewer добавляет вкладку в нижней части для новой последовательности при оставлении исходной последовательности открытой.
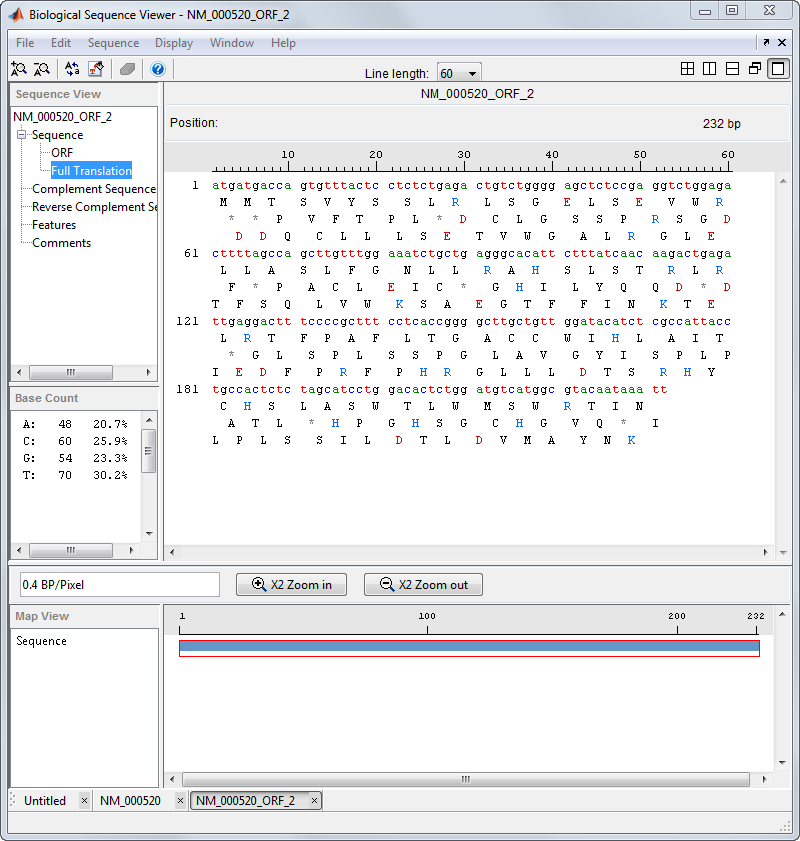
На левой панели нажмите Full Translation. Выберите Display > Amino Acid Residue Display > One Letter Code.
Sequence Viewer отображает последовательность аминокислот ниже последовательности нуклеотида.
Закройте Sequence Viewer из командной строки MATLAB с помощью следующего синтаксиса:
seqviewer('close')