Чтобы начать моделировать, вы можете:
Создайте модель PK с помощью мастера типовой конструкции, который позволяет вам задать количество отсеков, пути введения и типа устранения.
Расширьте любую модель, чтобы создать более высокие модели точности.
Создайте или загрузите свою собственную модель. Загрузите проект SimBiology® или модель SBML.
Следующая фигура сравнивает модель, как обычно представлено в фармакокинетике с той же моделью, показанной в диаграмме модели SimBiology. Для этого сравнения примите, что вы моделируете администрирование препарата с помощью модели 2D отсека с любым входом дозирования и линейной кинетикой устранения. (Структура модели остается то же самое с любым типом дозирования.)
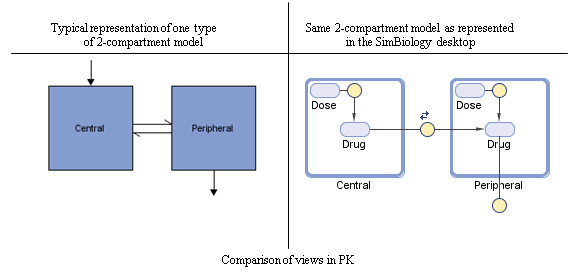
Отметьте следующее:
SimBiology представляет концентрацию или количество препарата в данном отсеке или объеме объектом разновидностей, содержавшим в отсеке.
SimBiology представляет обмен или поток препарата между отсеками и устранением препарата реакциями.
SimBiology представляет межразделенное на отсеки разрешение параметром (Q) который задает разрешение между отсеками.
SimBiology управляет расписанием дозирования с комбинацией разновидностей (Drug и/или Dose) и реакции (Dose -> Drug) в зависимости от того, следует ли администрирование в отсек за шариком, нулевым порядком, вливанием или кинетикой дозирования первого порядка. Для получения дополнительной информации о добавленных компонентах и оцененные параметры, смотрите Типы Дозирования.
Можно также просмотреть эту модель как функцию регрессии, y = f(k,u), где y ожидаемое значение, учитывая значения входа u, и значения параметров k. В SimBiology модель представляет f, и модель используется, чтобы сгенерировать функцию регрессии если yK, и u идентифицированы в модели.
Создать модель PK с конкретным количеством отсеков, дозируя тип и метод устранения:
Создайте PKModelDesign объект. PKModelDesign объект позволяет вам задать количество отсеков, пути введения и метода устранения, который использование SimBiology создать объект модели с необходимыми отсеками, разновидностями, реакциями и правилами.
pkm = PKModelDesign;
Добавьте отсек, задающий имя отсека, и опционально, тип дозирования и метод устранения. Также задайте, содержат ли данные переменную отклика, измеренную в этом отсеке и имеет ли доза (дозы) задержки. Например, при использовании tobramycin набора данных [1], задайте отсек под названием Central, с Bolus для DosingType свойство, linear-clearance для EliminationType свойство и true для HasResponseVariable свойство.
pkc1 = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true);Для описания другого DosingType и EliminationType значения свойств, смотрите Типы Дозирования и Типы Устранения.
Для описания HasResponseVariable свойство, смотрите HasResponseVariable. По крайней мере один отсек в модели должен иметь ответ. Несмотря на то, что SimBiology поддерживает множественные ответы на отсек при добавлении отсеков в PKModelDesign объект, вы ограничиваетесь одним ответом на отсек.
Примечание
Чтобы добавить отсек, которому сопоставили задержку с любой дозой, которая предназначается для него, устанавливает HasLag свойство к true:
pkc_lag = addCompartment(pkm, 'Central', 'DosingType', 'Bolus', ...
'EliminationType', 'linear-clearance', ...
'HasResponseVariable', true, 'HasLag', true);Или после добавления отсека, набор его HasLag свойство к true:
pkc1.HasLag = true;
Опционально, добавьте второй отсек под названием Peripheral, без дозирования, никакого устранения и никакой задержки. Установите HasResponseVariable свойство к true. Если вы используете tobramycin набор данных [1], пропускаете этот шаг и используете только один отсек.
pkc2 = addCompartment(pkm, 'Peripheral', 'HasResponseVariable', true);
Процесс типовой конструкции добавляет необходимые параметры, включая параметр, представляющий межразделенное на отсеки разрешение Q. Можно добавить больше отсеков путем повторения этого шага. Сложение каждого отсека создает цепь отсеков в порядке сложения отсека с двунаправленным потоком препарата между отсеками в модели.
Используйте указатель на отсек (pkc1 или pkc2), чтобы изменить свойства отсека.
Создайте объект модели SimBiology.
[modelObj, PKModelMapObj] = pkm.construct
construct метод возвращает объект модели SimBiology (modelObj) и PKModelMap объект (PKModelMapObj) это содержит отображение компонентов модели к элементам функции регрессии.
Примечание
Если вы изменяете PKModelDesign объект, необходимо создать новый объект модели с помощью construct метод. Изменения в PKModelDesign автоматически не распространяйте к ранее созданному объекту модели.
Выполните параметр, соответствующий как показано в, Выполняют Данные, Соответствующие Моделям PK/PD.
Объект модели и PKModelMap объект является входными параметрами для sbionlmefit, sbionlmefitsa и sbionlinfit функции используются в подборе кривой параметра.
| Для получения информации о... | Смотрите... |
|---|---|
| Дозирование типов | Дозирование типов |
| Типы устранения | Типы устранения |
| Подбор кривой параметра | Выполните данные, соответствующие моделям PK/PD |
| Симуляция модели и описания конфигураций модели | Симуляция модели |
При создании моделей SimBiology создает следующие компоненты модели для каждого отсека в модели, независимо от типа дозирования:
Две разновидности (Drug_CompartmentName и Dose_CompartmentName) для каждого отсека.
Реакция (Dose_CompartmentName -> Drug_CompartmentName) для каждого отсека, которым управляет кинетика действующих масс.
Параметр (ka_CompartmentName) для каждого отсека, представляя показатель поглощения препарата, когда поглощение следует за кинетикой первого порядка. Это - параметр форвардного курса для Dose_CompartmentName -> Drug_CompartmentName реакция.
Параметр (Tk0_CompartmentName) для каждого отсека, представляя длительность поглощения препарата, когда поглощение следует за кинетикой нулевого порядка.
Параметр (TLag_CompartmentName) для каждого отсека, представляя задержку для любой дозы, которая предназначается для того отсека и также который задан как наличие задержки.
Для дозирования типов, которые имеют фиксированную длительность вливания или поглощения (infusion и zero-order), можно использовать перекрывающиеся дозы. Дозы являются дополнением.
Следующая таблица описывает типы дозирования, параметры по умолчанию, чтобы оценить, и приводит компоненты модели, созданные и используемые для дозирования.
| Дозирование типа | Описание | Используемые компоненты модели SimBiology | Параметры по умолчанию, чтобы оценить |
|---|---|---|---|
''(пустой символьный вектор) | Никакая доза | Разновидности ( | 'none' |
Приложение SimBiology — Командная строка — | Принимает, что количество препарата увеличено немедленно во время дозы. В модели SimBiology начальная концентрация препарата основана на суммарной дозе и объеме отсека, содержащего препарат. | Разновидности ( | 'none' |
Приложение SimBiology — Командная строка — | Принимает, что вселенный препарат составляет увеличения при постоянном известном поглощении (или вливание) уровень по известной длительности. Импортированный набор данных должен содержать уровень и не длительность вливания. SimBiology использует эту информацию, чтобы изменить концентрацию разновидностей на постоянном уровне по длительности, заданной в наборе данных. | Разновидности ( | 'none' |
Приложение SimBiology — Командная строка — | Принимает, что препарат добавляется на постоянном уровне по фиксированной, но неизвестной длительности. |
|
|
Приложение SimBiology — Командная строка — | Принимает, что уровень, на котором поглощен препарат, не является постоянным. В модели SimBiology показатель поглощения принят, чтобы управляться кинетикой действующих масс. |
|
|
| Тип устранения | Описание | Созданные компоненты модели SimBiology | Параметры по умолчанию, чтобы оценить |
|---|---|---|---|
Приложение SimBiology — Командная строка — | Принимает простую кинетику действующих масс в устранении препарата. В модели SimBiology устранение задано кинетикой действующих масс с константой скорости устранения, заданной параметром форвардного курса (ke). |
|
|
Приложение SimBiology — Командная строка — | Принимает простую кинетику действующих масс в устранении препарата. В модели SimBiology, похожей на Linear {Elimination Rate, Volume}. Но кроме того, эта опция позволяет вам задать модель в терминах разрешения (Cl) где, Cl = ke * volume). |
|
|
Приложение SimBiology — Командная строка — | Принимает, что устранением управляет кинетика Михаэлиса-Ментен. |
|
|
Отсеки, созданные, когда вы генерируете модель SimBiology, формируют цепь, и каждая пара соединенных отсеков соединяются транспортной реакцией, похожей на линейное устранение. Сложение двух отсеков, C1 и C2, генерирует обратимую реакцию массовой акции C1.Drug_C1 <-> C2.Drug. Параметр форвардного курса является разделенным на отсеки разрешением, Q12, разделенный на объем C1. Противоположным параметром уровня является Q12, разделенный на объем C2.
Процесс добавления каждой пары отсеков в цепочечном Cm и Cn генерирует следующие компоненты модели:
Параметр Qmn представление разделенного на отсеки разрешения между теми двумя отсеками. Этот параметр добавляется к списку параметров, которые будут оценены (Estimated свойство PKModelMap объект.
Параметр (kmn) представление уровня передачи препарата от Cm к Cn, где kmn = Qmn/Vm.
Параметр (knm) представление уровня Cn к Cm, где knm = Qmn/Vn.
Обратимая реакция массовой акции между этими двумя отсеками, Cm.Drug_Cm <-> Cn.Drug_Cn, параметром форвардного курса kmn, и противоположный параметр уровня knm.
Первоначальное правило присвоения, которое инициализирует значение параметра kmn, на основе начальных значений для Cm и Qmn.
Первоначальное правило присвоения, которое инициализирует значение параметра knm, на основе начальных значений для Cn и Qmn.
Модульное преобразование преобразует соответствующие физические количества в одну сопоставимую модульную систему для того, чтобы разрешить их. Это преобразование при подготовке к правильной симуляции, но SimBiology возвращает физические количества в модели в модулях, которые вы задаете.
Независимо от того, является ли модульным преобразованием on или off, необходимо описать данные о дозировании в сумме. По умолчанию Unit Conversion является off, таким образом, необходимо гарантировать, что модули для данных и модели сопоставимы друг с другом. Если Unit Conversion является on, необходимо задать модули.
Параметры в модели имеют модули по умолчанию. Если модульным преобразованием является on, можно изменить модули, пока размерности сопоставимы. Эти модули по умолчанию, которые вы можете использовать, чтобы задать значения для исходного предположения, следующие.
| Физическое количество или параметр модели | Модуль |
|---|---|
| Центральный или периферийный объем отсека (Central или Peripheral) | liter |
| Уровень устранения первого порядка (ke) | 1/second |
| Michaelis, постоянный (Km) | milligram/liter |
| Максимальная скорость реакции, кинетика Михаэлиса-Ментен (Vm) | milligram/second |
| Разрешение (Cl) | liter/second |
| Длительность поглощения (Tk0) | second |
| Показатель поглощения (ka) | 1/second |
Используйте опции параметров конфигурации, чтобы повернуть модульное преобразование on или off. Для получения дополнительной информации смотрите Симуляцию модели.
Для получения дополнительной информации согласно размерному анализу для скоростей реакции, смотрите, Как Оценены Скорости реакции.