В этом примере показано, как идентифицировать важные параметры модели для модели [1] роста опухоли. В этом примере вы вычисляете локальную зависящую от времени чувствительность роста опухоли относительно параметров модели для лекарства от рака.
Модель, используемая в этом примере, является реализацией SimBiology® фармакокинетической/фармакодинамической модели (PK/PD) Simeoni и др. Это определяет количество эффекта лекарств от рака на кинетике роста опухоли от в естественных условиях исследований на животных. Фармакокинетика препарата описана моделью 2D отсека с дозированием болюсного внутривенного введения и линейным устранением (ke) из отсека Central. Рост опухоли является двухфазным процессом с начальным экспоненциальным ростом, сопровождаемым линейным ростом. Темп роста распространяющихся опухолевых клеток описан
L0, L1 и Ψ являются параметрами роста опухоли, x1 является весом распространяющихся опухолевых клеток, и w является общим весом опухоли. В отсутствие любых наркотиков опухоль состоит из распространяющихся ячеек только, то есть, w = x1. В присутствии противоракового агента часть распространяющихся ячеек преобразовывается в нераспространяющиеся ячейки. Уровень этого преобразования принят, чтобы быть функцией концентрации препарата в плазме и факторе эффективности k2. Нераспространяющиеся ячейки x2 проходят серию транзитных этапов (x3 и x4) и в конечном счете очищены от системы. Поток - через транзитных отсеков моделируется как процесс первого порядка с константой скорости k1.
Модель SimBiology вносит эти корректировки в фармакодинамику роста опухоли:
Вместо того, чтобы задать вес опухоли как сумму x1, x2, x3 и x4, модель задает вес опухоли реакцией под названием Increase, null → tumor_weight, со скоростью реакции .
tumor_weight является общим весом опухоли, x1 является весом распространяющихся опухолевых клеток и L0, и L1 является параметрами роста опухоли.
Точно так же модель задает уменьшение в весе опухоли реакцией под названием Decay, tumor_weight → null, со скоростью реакции k1*x4. Постоянный k1 является параметром форвардного курса, и x4 является последними разновидностями в ряду транзитных сокращений веса опухоли.
ke является функцией разрешения и объемом центрального отсека: ke = Cl_Central/Central.
Анализ чувствительности позволяет вам определить, какие константы скорости и концентрации в модели имеют значительное влияние на полное поведение модели. В этом примере показано, как заняться расследованиями, какие параметры модели чувствительны к росту опухоли с помощью встроенной программы анализа чувствительности. А именно, этот пример показывает вам, как вычислить чувствительность разновидностей tumor_weight относительно параметров модели можно следующим образом:
Предположим, что вы уже калибровали параметры модели роста опухоли к экспериментальным данным PK/PD и сохраненным оценкам параметра и начальным условиям как вариант под названием parameterEstimates в вашей модели. Как последующий анализ, вы хотите узнать, какие параметры модели чувствительны к росту опухоли. Следующие шаги показывают, как сделать настолько использующий встроенную программу анализа чувствительности.
В командной строке MATLAB® загрузите модель (m1) путем ввода:
sbioloadproject tumor_growth_vpop_sa.sbprojОткройте модель в приложении SimBiology Model Analyzer путем ввода:
simBiologyModelAnalyzer(m1)
В качестве альтернативы можно открыть приложение путем нажатия на SimBiology Model Analyzer на вкладке Apps. Затем загрузите модель из приложения путем выбора Model> Import Model from MATLAB.
На вкладке Home выберите Program> Calculate Sensitivities. Новая программа открывается.
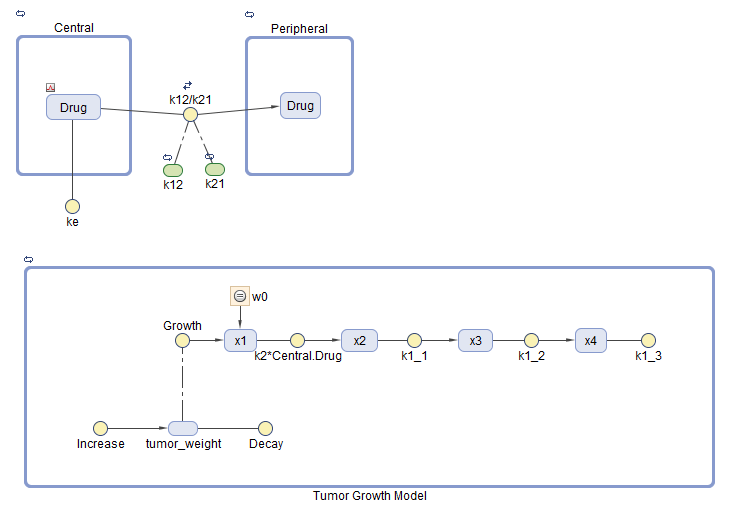
В разделе Variants шага Model (шаг настройки) программы, выберите parameterEstimates. Этот вариант содержит ранее оцененные значения параметров и начальные условия для препарата.
В разделе Doses выберите interval_dose. Эта повторная доза применяет 30 мг препарата каждые 4 дня, начиная в день 7 в течение в общей сложности пяти раз.
В разделе States To Log выберите [Tumor Growth Model].tumor_weight только.
Совет
Контекстные меню Variants, таблицы Doses и States To Log содержат опции, чтобы добавить соответствующие компоненты модели и показать детали этих компонентов в приложении Model Builder. Доступ к контекстному меню путем щелчка правой кнопкой по таблице.
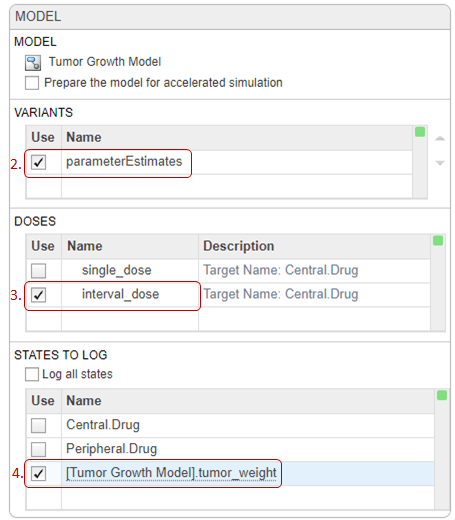
На шаге Simulation (шаг выполнения), по умолчанию, метод нормализации установлен в Full (full dedimensionalization), означая, что приложение полностью нормирует чувствительность так, чтобы они могли сравниться друг с другом. Для получения дополнительной информации смотрите Normalization.
Совет
Можно запустить каждый шаг выполнения программы отдельно. Шаг выполнения включает кнопку Run рядом с именем шаг. Выполнение отдельного шага особенно полезно, если программа содержит несколько шагов, и вы хотите видеть, что промежуточное звено следует из конкретного шага. Путем выполнения так, можно внести изменения по мере необходимости прежде, чем запустить следующий шаг или целую программу. Чтобы запустить целую программу, нажмите кнопку Run на вкладке Home.
В разделе Sensitivities to Compute щелкните правой кнопкой где угодно по таблице и выберите Add All Constant Parameters. Очистите ke, потому что он задан в зависимости от Central объем и Cl_Central, который уже является входом. Кликните по последней пустой строке и введите tumor. Затем выберите [Tumor Growth Model].tumor_weight из списка. Разновидность tumor_weight затем добавляется как выход.
Можно просмотреть начальные условия количеств модели, а именно, отсеки, разновидности и параметры, прежде, чем симулировать модель. Начальные условия являются значениями количества во времени симуляции = 0 после применения правил присвоения, вариантов и доз (если любой во время = 0). Для получения дополнительной информации смотрите Симуляцию модели. Можно использовать эту информацию, чтобы отладить модель и проверку, если значения количества инициализируются, как вы ожидаете прежде, чем симулировать модель.
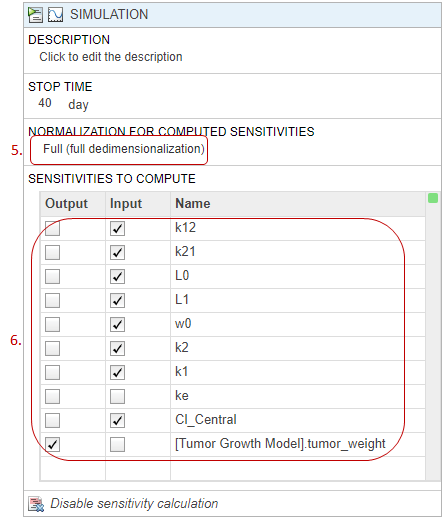
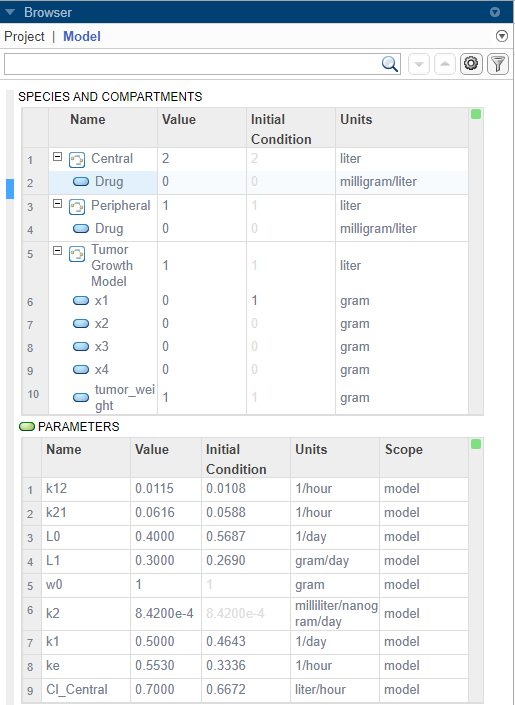
В панели Browser нажмите Model.
Щелкните правой кнопкой где угодно в рамках таблицы Species and Compartments или таблицы Parameters. Выберите Show Model Initial Conditions.
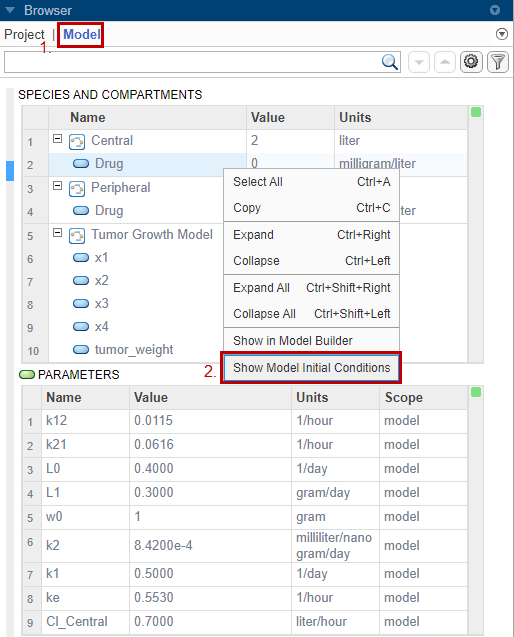
Приложение показывает столбец Initial Condition в обеих таблицах количества. Эти значения - то, что программа использует во время = 0, когда вы симулируете.
Затем запустите программу путем нажатия на Run на вкладке Home.
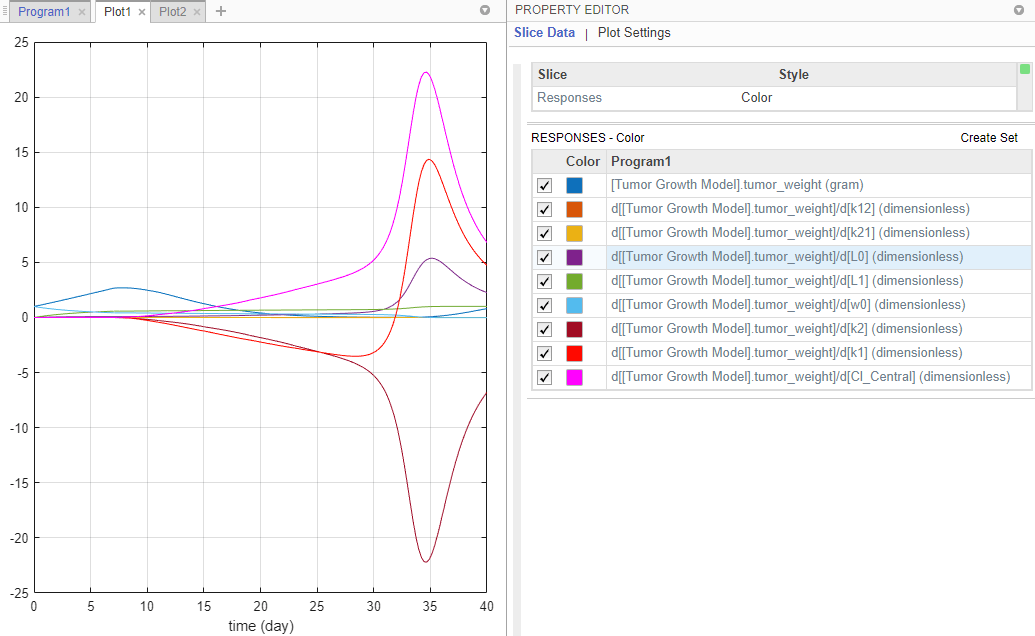
После анализа чувствительности программа автоматически генерирует два графика. Кликните по вкладке Plot1, чтобы просмотреть график курса времени веса опухоли и других значений чувствительности d[tumor_weight]/d[parameter]. Графики временной зависимости для d[tumor_weight]/d[k21] и d[tumor_weight]/d[k12] останьтесь относительно плоскими по сравнению с другой чувствительностью, указав, что рост опухоли не чувствителен к параметрам k12 и k21. Property Editor позволяет вам выбрать который графики отобразиться.
Совет
Графики поддерживаются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками состояния. Когда данные (или экспериментальные данные или результаты симуляции) удалены или изменены, графики также обновляются согласно изменениям в базовых данных.
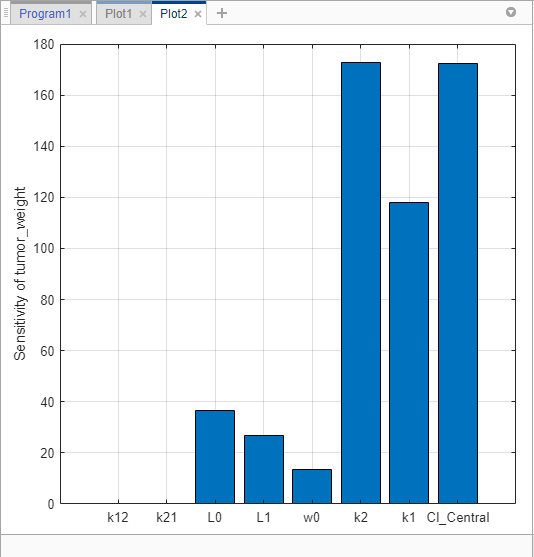
Можно также исследовать те же данные о чувствительности путем проверки величины вычисленной чувствительности, интегрируемой в зависимости от времени. Второй сгенерированный график (Plot2) показывает столбиковую диаграмму, содержащую такую информацию. График показывает, что вес опухоли чувствителен к L0, L1, w0, k2, k1 и Cl_Central, но не к k12 и k21.
Сохраните результаты анализа чувствительности в отдельной папке.
Нажмите Project в панели Browser. Расширьте папку Program1.
Щелкните правой кнопкой по папке LastRun. Выберите Save Data.
В диалоговом окне Save Data введите sa_data как имя данных.
Результаты анализа чувствительности показывают, что вес опухоли не чувствителен к значениям модели k12 и k21. Исследуйте, приводят ли, варьируясь эти значения параметров к различным результатам чувствительности.
Варьируйтесь k12. Во-первых, варьируйтесь значения для параметра k12.
Кликните по вкладке Program1. Кликните (+) плюс наверху программы и выберите Generate Samples.
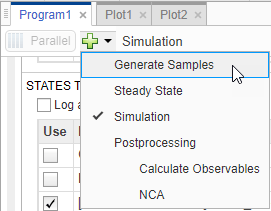
Шаг Generate Samples появляется. В разделе Parameter Set шага дважды кликните пустую ячейку в Component Name и введите k12. Установите следующие опции:
Ввод Область значений значений
Spacing — линейный
Min 1e-3
Max 1
# Of Steps — 25
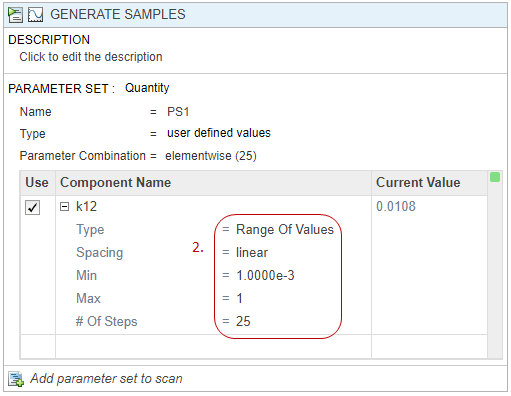
Отключите генерацию графика по умолчанию путем нажатия кнопки графика наверху шага.
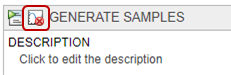
На шаге Simulation, под Sensitivities to Compute, очищают все входные параметры кроме k12. Сохраните tumor_weight как выход. Кроме того, отключите генерацию графика по умолчанию путем нажатия кнопки графика наверху шага.
На вкладке Home нажмите кнопку Run, чтобы запустить целую программу.
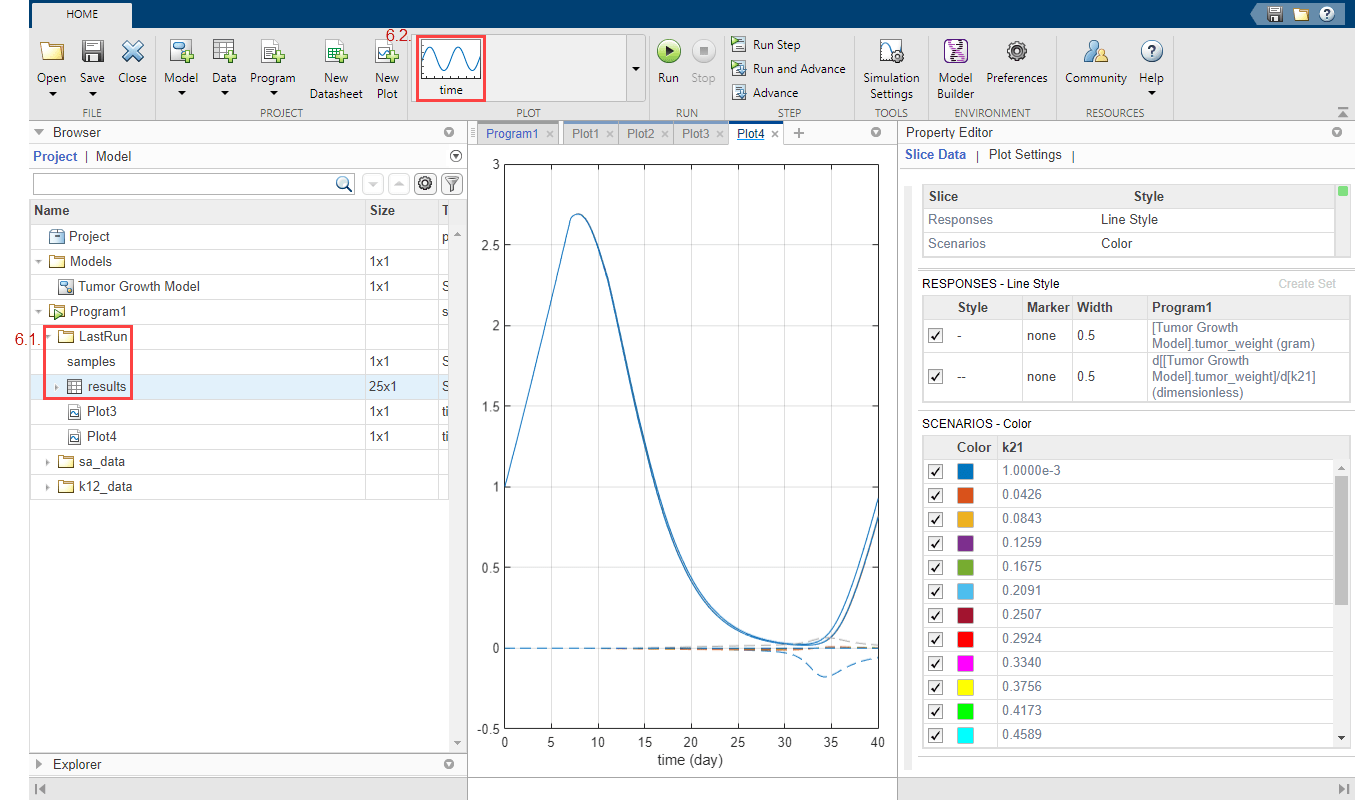
Если симуляция заканчивается, чувствительность, results хранится в папке LastRun. Расширьте папку LastRun. Нажмите results. Затем выберите time из раздела Plot по вкладке Home.
Responses в графике соответствует весу опухоли и чувствительности веса опухоли относительно k12. Каждый ответ построен с помощью различного стиля линии. Scenarios соответствует каждому скану параметра (сценарий симуляции). Каждый сценарий построен с помощью различного цвета.
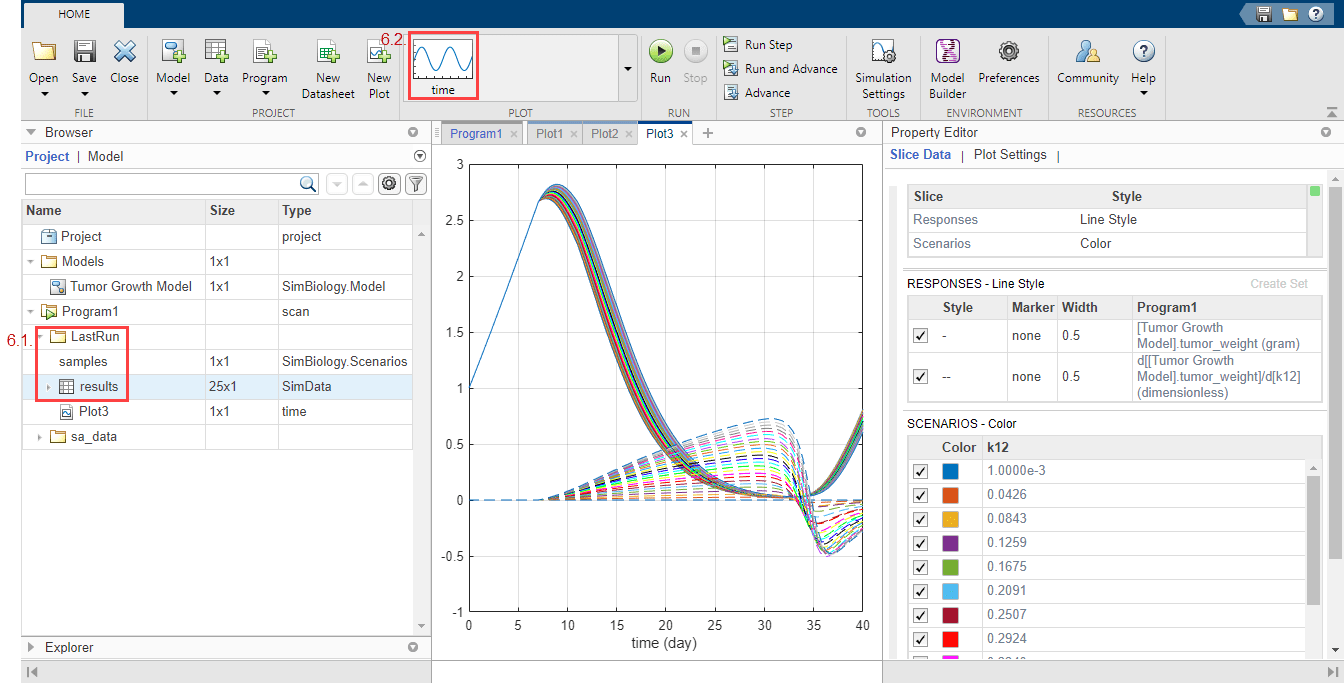
Настройте график так, чтобы график использовал различный цвет для каждого ответа вместо этого. В таблице Slice Data очистите Style Scenarios путем выбора пустой. Установите Responses на Color.
В таблице Responses очистите tumor_weight и сохраните только результаты чувствительности.
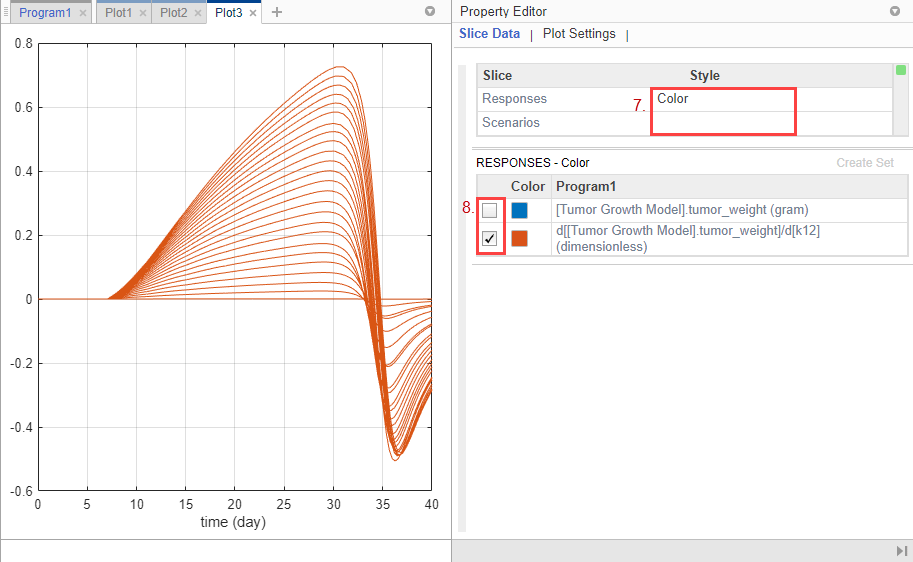
График предполагает, что вес опухоли чувствителен к некоторым значениям параметров k12, когда варианты модели, отличающиеся, чем предполагаемые значения параметров, исследуются.
Постройте чувствительность k12 вместе с предыдущими результатами чувствительности, насколько чувствительный рост опухоли к k12 относительно других параметров модели. В панели Browser расширьте папку sa_data что вы сохраненный ранее. Затем расширьте results.
Мультивыберите (Shift + Click) элементы данных чувствительности для L0, L1, w0, k2, k1 и Cl_Central. Затем перетащите их на графике чувствительности k12 Plot3.
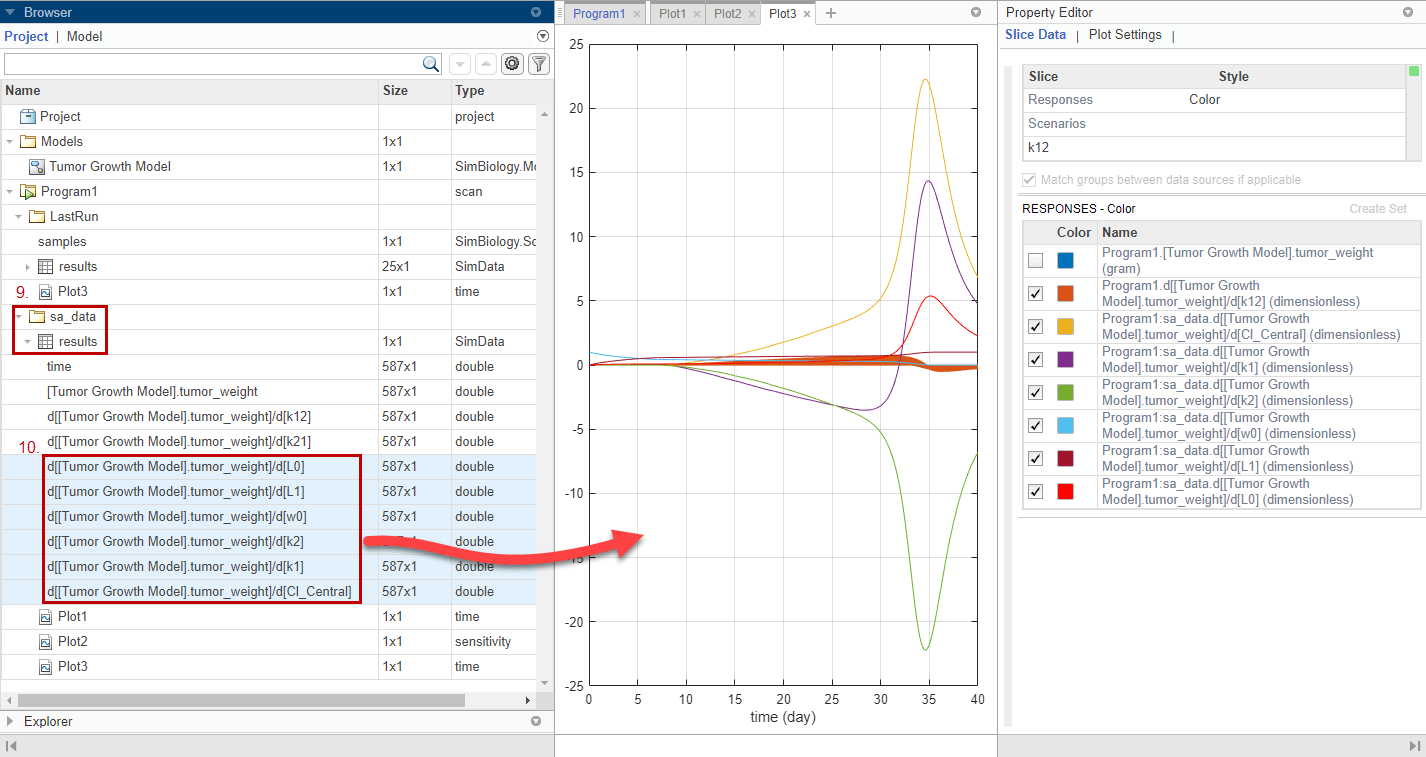
График предполагает, что рост опухоли еще более чувствителен к большинству других параметров, чем k12.
Сохраните результаты в отдельной папке. Щелкните правой кнопкой по LastRun и выберите Save Data. Введите k12_data как имя данных.
Варьируйтесь k21. Можно выполнить подобный анализ различным k21 и сравнив его результаты с другой чувствительностью параметра.
Возвратитесь к Program1. На шаге Generate Samples нажмите Add parameter set to scan. Появляется новый раздел Parameter Set.
Дважды кликните пустую ячейку в Component Name и введите k21. Установите те же опции как k12:
Ввод Область значений значений
Spacing — линейный
Min 1e-3
Max 1
# Of Steps — 25
Отключите первый набор параметров (PS1). Укажите на верхний угол раздела Parameter Set PS1 и кликните по кнопке действий. Затем выберите Disable Parameter Set.
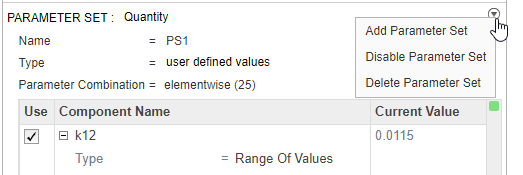
Parameter Set затем grayed, чтобы указать, что вы успешно отключили его.
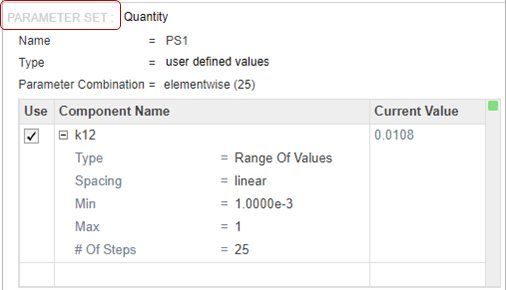
На шаге Simulation выберите k21 как единственный вход и tumor_weight как выход. Отключите генерацию графика по умолчанию путем нажатия кнопки графика наверху шага.
Поразите Run от вкладки Home, чтобы запустить программу снова.
Если симуляция заканчивается, чувствительность, results хранится в папке LastRun. Расширьте папку LastRun. Нажмите results. Затем выберите time из раздела Plot по вкладке Home.
В таблице Slice Data очистите Style Scenarios путем выбора пустой. Установите Responses на Color.
В таблице Responses очистите tumor_weight и сохраните только результаты чувствительности.
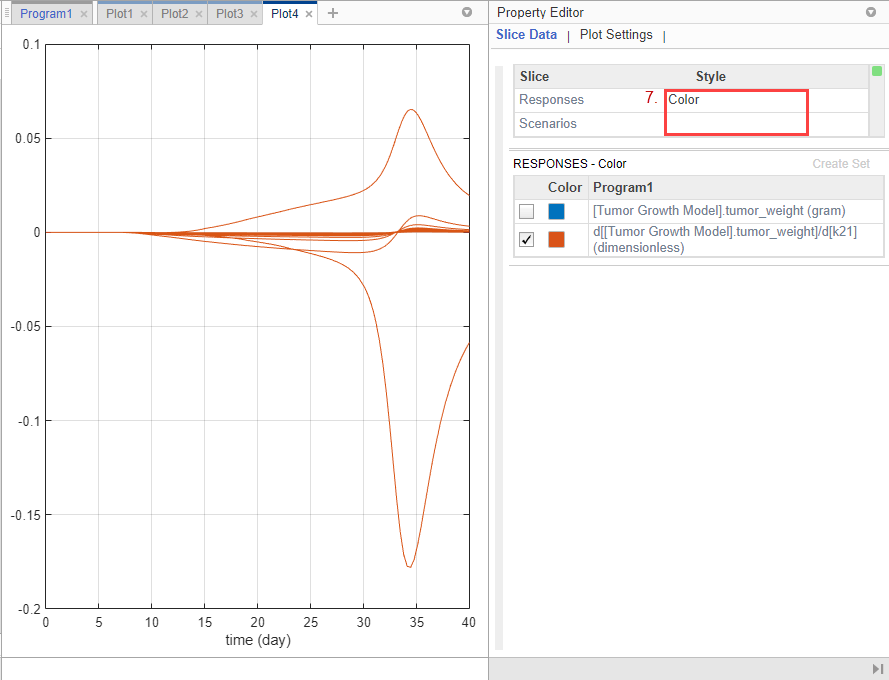
График предполагает, что вес опухоли чувствителен к некоторым значениям параметров k21, когда варианты модели, отличающиеся, чем предполагаемые значения параметров, исследуются.
Постройте чувствительность k21 вместе с предыдущими результатами чувствительности выдержать сравнение. Нажмите Plot3. В панели Browser расширьте папку LastRun. Затем расширьте results.
Перетащите данные о чувствительности k21 d[[Tumor Growth Model].tumor_weight]/d[k21] на Plot3.
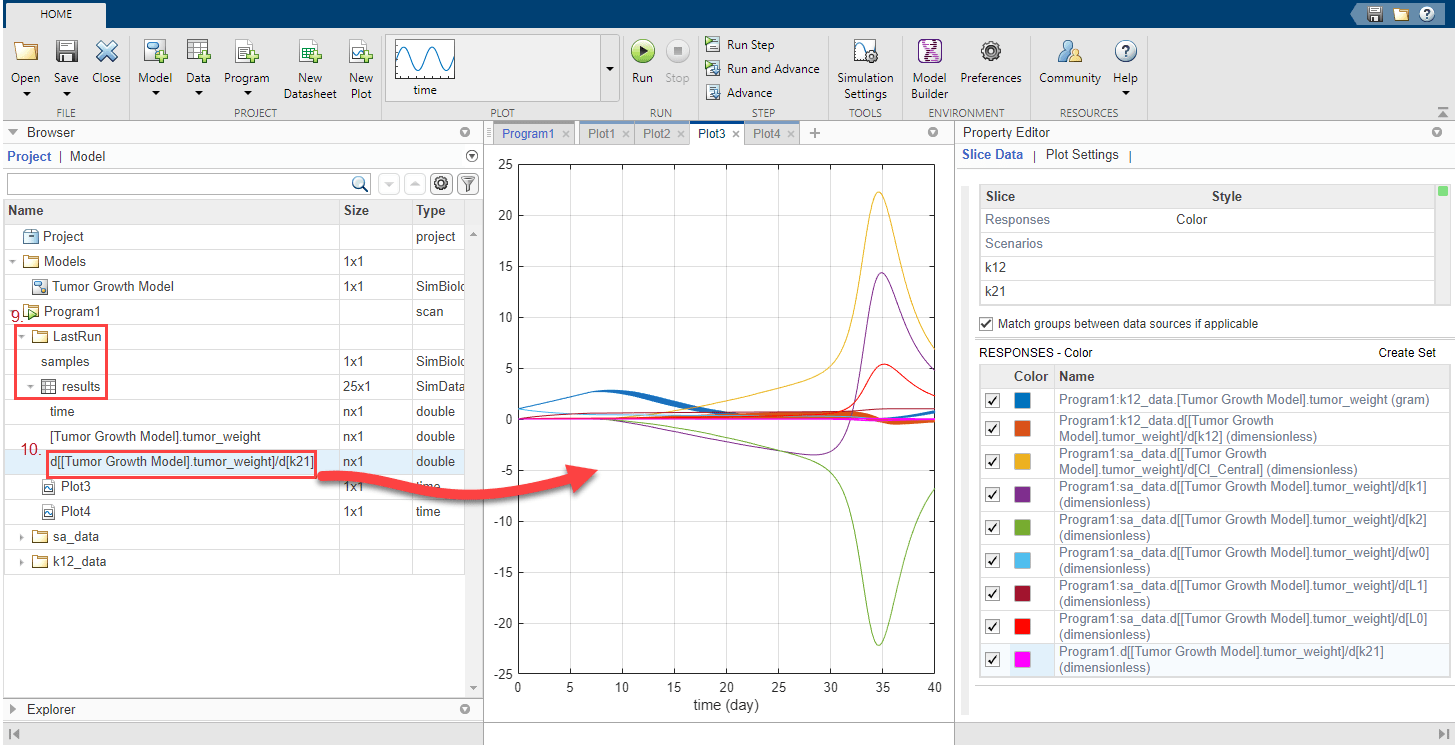
График предполагает, что рост опухоли относительно более чувствителен к большинству других параметров, чем k21 и k12.
В этом примере показано, как выполнить анализ чувствительности, чтобы найти важные параметры модели, к которым рост опухоли чувствителен. Начальный анализ приходит к заключению, что вес опухоли не чувствителен к некоторым параметрам. Пример затем исследует пространство параметров этих менее чувствительных параметров и сравнивает результаты чувствительности с теми из других параметров модели. Можно использовать такую информацию о чувствительности для других исследований, например, выполнить симуляции Монте-Карло путем варьирования чувствительных параметров, чтобы исследовать динамику модели и биологическую изменчивость.
[1] Simeoni, M., П. Магни, К. Кэммия, Г. Де Николао, V. Шафраны, Э. Пезенти, М. Джермани, я. Poggesi и М. Роккетти. 2004. Прогнозирующее фармакокинетическо-фармакодинамическое моделирование кинетики роста опухоли в моделях ксенотрансплантата после администрирования противораковых агентов. Исследования рака. 64:1094-1101.