В этом примере показано, как сгенерировать и симулировать виртуальных пациентов, использующих приложение SimBiology Model Analyzer. В этом примере виртуальный пациент представлен как одна реализация параметров модели. Пример использует модель [1] роста опухоли, чтобы исследовать изменчивость некоторых параметров модели, которые влияют на рост опухоли и исследуют различные режимы дозирования, чтобы управлять им.
Этот пример требует Statistics and Machine Learning Toolbox™.
Модель, используемая в этом примере, является реализацией SimBiology® фармакокинетической/фармакодинамической модели (PK/PD) Simeoni и др. Это определяет количество эффекта лекарств от рака на кинетике роста опухоли от в естественных условиях исследований на животных. Фармакокинетика препарата описана моделью 2D отсека с дозированием болюсного внутривенного введения и линейным устранением (ke) из отсека Central. Рост опухоли является двухфазным процессом с начальным экспоненциальным ростом, сопровождаемым линейным ростом. Темп роста распространяющихся опухолевых клеток описан
L0, L1 и Ψ являются параметрами роста опухоли, x1 является весом распространяющихся опухолевых клеток, и w является общим весом опухоли. В отсутствие любых наркотиков опухоль состоит из распространяющихся ячеек только, то есть, w = x1. В присутствии противоракового агента часть распространяющихся ячеек преобразовывается в нераспространяющиеся ячейки. Уровень этого преобразования принят, чтобы быть функцией концентрации препарата в плазме и факторе эффективности k2. Нераспространяющиеся ячейки x2 проходят серию транзитных этапов (x3 и x4) и в конечном счете очищены от системы. Поток - через транзитных отсеков моделируется как процесс первого порядка с константой скорости k1.
Модель SimBiology вносит эти корректировки в фармакодинамику роста опухоли:
Вместо того, чтобы задать вес опухоли как сумму x1, x2, x3 и x4, модель задает вес опухоли реакцией под названием Increase, null → tumor_weight, со скоростью реакции .
tumor_weight является общим весом опухоли, x1 является весом распространяющихся опухолевых клеток и L0, и L1 является параметрами роста опухоли.
Точно так же модель задает уменьшение в весе опухоли реакцией под названием Decay, tumor_weight → null, со скоростью реакции k1*x4. Постоянный k1 является параметром форвардного курса, и x4 является последними разновидностями в ряду транзитных сокращений веса опухоли.
ke является функцией разрешения и объемом центрального отсека: ke = Cl_Central/Central.
Виртуальное население в этом примере представлено виртуальными пациентами в виде отличных наборов терпеливо-специфичных значений параметров. Предположим, что, на основе предварительных знаний или анализа чувствительности модели, рост опухоли чувствителен к этим параметрам модели: L0, L1, w0, k1, k2 и Cl_Central. При предположении, что эти биологические параметры следуют за логарифмически нормальным распределением (и должно всегда быть положительным), можно сгенерировать виртуальных пациентов, которые представляют различные значения параметров, чертившие от объединенного логарифмически нормального распределения.
Логарифмически нормальное распределение использует эти параметры:
\mu Среднее значение логарифмических значений
\sigma Стандартное отклонение логарифмических значений
Для получения дополнительной информации смотрите Логарифмически нормальное Распределение (Statistics and Machine Learning Toolbox).
В этом примере sigma принят, чтобы быть 0.01 для всех параметров. Для маленького значения sigma среднее значение логарифмически нормального распределения приблизительно равно журналу значения модели. Следовательно, этот пример принимает, что mu для каждого параметра является журналом значения модели.
В этом примере лекарство от рака используется, чтобы управлять ростом опухоли. Каждый виртуальный пациент получает то же количество препарата в том же расписании. Ответ роста опухоли симулирован для каждого пациента. Суммарные дозы затем варьируются, чтобы найти область значений суммарных доз, которые могут подавить рост опухоли многих виртуальных пациентов в населении.
Следующие шаги показывают, как чертить демонстрационные значения от объединенного вероятностного распределения для параметров модели, которые чувствительны к росту опухоли, чтобы представлять группу виртуальных пациентов.
В командной строке MATLAB® загрузите модель (m1) путем ввода:
sbioloadproject tumor_growth_vpop_sa.sbprojОткройте модель в приложении SimBiology Model Analyzer путем ввода:
simBiologyModelAnalyzer(m1)
В качестве альтернативы можно открыть приложение путем нажатия на SimBiology Model Analyzer на вкладке Apps. Затем загрузите модель из приложения путем выбора Model> Import Model from MATLAB.
Создайте программу, чтобы чертить демонстрационные значения от объединенного логарифмически нормального распределения для роста опухоли чувствительные параметры: L0, L1, w0, k1, k2 и Cl_Central.
На вкладке Home выберите Program> Generate Samples. Новая программа открывается.
В разделе Variants на шаге Model программы кликните по опции, чтобы просмотреть варианты, которые будут применены. Затем выберите parameterEstimates, который содержит предполагаемые значения параметров.
На шаге Generate Samples нажмите кнопку графика, чтобы отключить генерацию графика по умолчанию. Вы построите выборки позже.
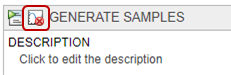
В разделе Parameter Set, набор Type к values from a distribution.
Установите Number Of Samples на 25.
Дважды кликните пустую ячейку в столбце Component Name и введите L0.
Измените Distribution для L0 к Lognormal. По определению mu является средним значением логарифмических значений. Так, изменение mu к-0.5644, который является log(Current Value) или log(0.5687). Измените sigma в 0,01. Для маленького значения sigma среднее значение логарифмически нормального распределения приблизительно равно log(Current Value). Для получения дополнительной информации смотрите Логарифмически нормальное Распределение (Statistics and Machine Learning Toolbox).
Дважды кликните пустую ячейку в столбце Component Name и добавьте L1. Повторите тот же процесс, чтобы изменить распределение в логарифмически нормальный и установить значения sigma и mu. Точно так же добавьте w0, k1, k2 и ke. Эта таблица приводит соответствующие значения mu этих параметров, которые можно скопировать и вставить в программное обеспечение. Измените sigma в 0,01 для каждого параметра.
Параметр | Текущее значение | mu или журнал (текущее значение) |
|---|---|---|
L0 | 0.5687 | -0.5644 |
L1 | 0.2690 | -1.3130 |
w0 | 1 | 0 |
k1 | 0.4643 | -0.7672 |
k2 | 8.4200e-4 | -7.0797 |
Cl_Central | 0.6672 | -0.4047 |
Для Sampling используйте опцию по умолчанию: random sampling with rank correlation matrix, где матрица является единичной матрицей.
Следующий рисунок показывает раздел Parameter Set всеми сконфигурированными параметрами. Красные числа соответствуют предыдущим шагам.
(Необязательно) Сохраните проект под новым именем путем выбора Save > Save Project As на вкладке Home.
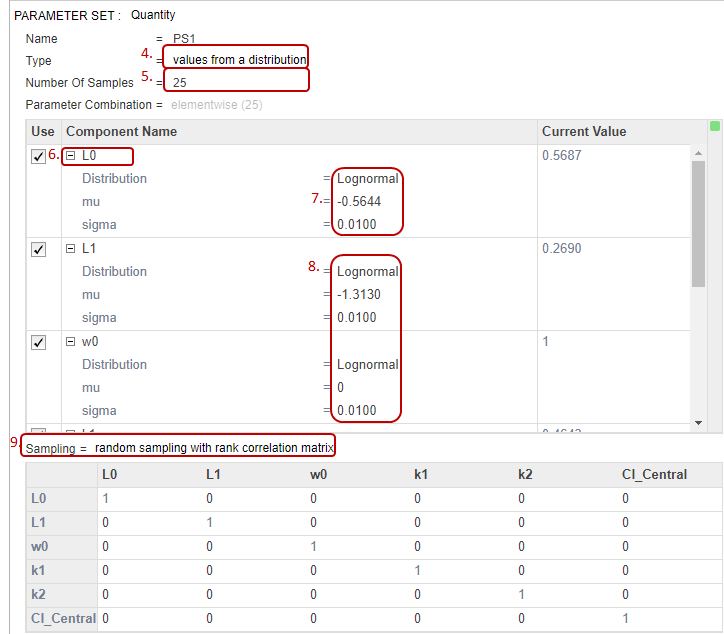
Добавьте информацию о дозах как другой набор параметров путем определения различной суммарной дозы — а именно, шесть суммарных доз, которые равномерно распределены от 5 до 35 мг. Затем объедините дозы с первым набором параметров (виртуальные пациенты) использование Декартова метода комбинации. Этот метод комбинирует каждого виртуального пациента с каждой суммарной дозой, чтобы сгенерировать в общей сложности 150 итераций (или сценарии симуляции). Для получения дополнительной информации см. Сценарии Симуляции Объединения в SimBiology.
В нижней части шага Generate Samples нажмите Add parameter set to scan. Другой набор параметров (PS2) появляется.
Дважды кликните пустую ячейку в столбце Component Name и введите interval. Список выбора появляется. Выберите interval_dose.Amount.
Установите Type на Individual Values и набор Values к [5:6:35].
Около верхней части шага Generate Samples, под Parameter Set Combinations, гарантируют, что тип Combination установлен в cartesian объединить PS1 и PS2.
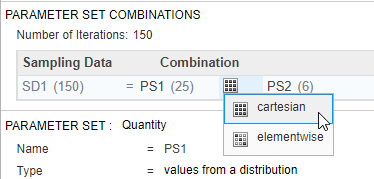
Если вы задали объединенное логарифмически нормальное распределение для параметров модели, области значений суммарных доз и метода комбинации, можно запустить шаг, чтобы сгенерировать различные параметры и комбинации дозы.
Нажмите кнопку Run this program step, чтобы сгенерировать выборки.
Совет
Можно запустить каждый шаг выполнения программы отдельно. Шаг выполнения включает кнопку Run рядом с именем шаг. Выполнение отдельного шага особенно полезно, если программа содержит несколько шагов, и вы хотите видеть, что промежуточное звено следует из конкретного шага. Путем выполнения так, можно внести изменения по мере необходимости прежде, чем запустить следующий шаг или целую программу. Чтобы запустить целую программу, нажмите кнопку Run на вкладке Home.
По умолчанию приложение хранит сгенерированный samples в папке LastRun программы.
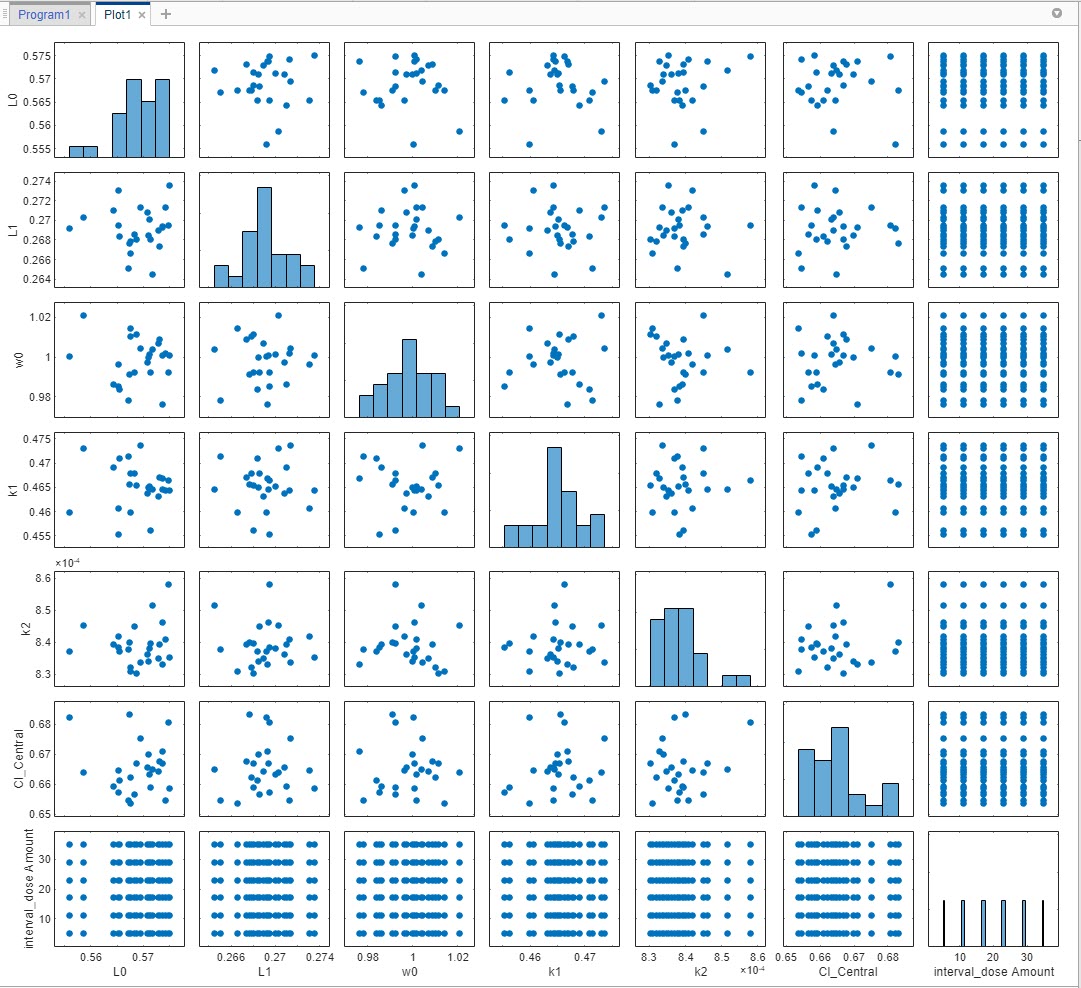
Визуализируйте сгенерированные выборки. В панели Browser слева, расширьте папку Program1. Расширьте папку LastRun, и затем нажмите samples. В разделе Plot по вкладке Home нажмите Plot Matrix. График показывает, что распределение каждого параметра варьировалось вокруг его значения модели, кроме суммарных доз, которые появляются в универсальной области значений. Обратите внимание на то, что ваш график может варьироваться из графика, показанного здесь из-за случайности.
Примечание
Можно позволить генерации графика по умолчанию отобразить график автоматически каждый раз, когда вы запускаете шаг. Для этого нажмите кнопку графика наверху шага Generate Samples. Следует иметь в виду, что то, когда шаг программы имеет много выборок, строя все выборки, может быть трудоемким.
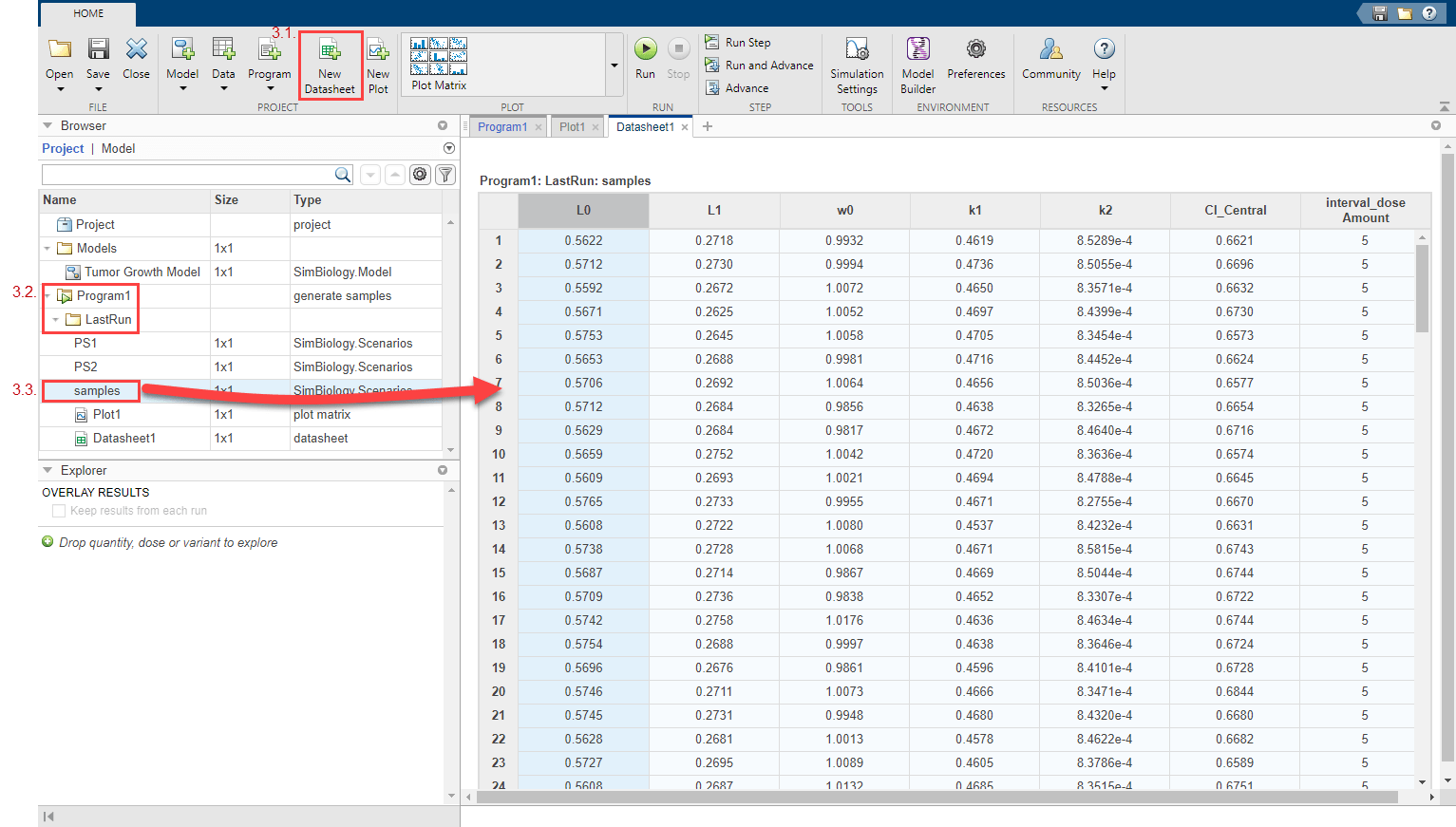
Отобразите сгенерированных виртуальных пациентов и комбинации дозы численно в табличном формате. На вкладке Home нажмите New Datasheet. От папки LastRun программы перетащите samples в новую таблицу данных. Каждая строка таблицы представляет сценарий симуляции различными значениями параметра модели и суммарными дозами.
Если у вас есть готовые выборки, можно симулировать модель, чтобы исследовать рост опухоли виртуальных пациентов, получающих различные суммарные дозы.
Вернитесь к программе путем нажатия на вкладку Program1.
Щелкните (+) значок в верхнем левом углу и нажмите Simulation.
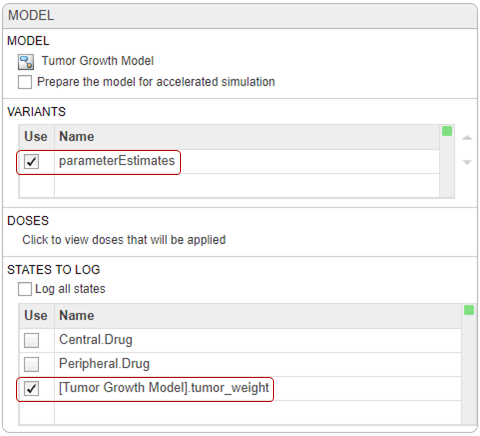
На шаге настройки Model, на разделе Variants, убеждаются, что parameterEstimates выбран. В разделе States To Log кликните по опции для просмотра состояний, и затем снимите все флажки кроме [Tumor Growth Model].tumor_weight.
Наверху шага Simulation (прокручивают вниз, чтобы видеть этот шаг), нажмите кнопку графика, чтобы отключить генерацию графика по умолчанию.
Нажмите кнопку Run this program step, чтобы симулировать модель.
Приложение симулирует сгенерированные сценарии от шага Generate Samples, который вы запустили ранее. Вы не должны повторно выполнять шаг.
Если симуляция заканчивается, приложение сохраняет симуляцию results в папке LastRun.
В папке LastRun в панели Browser нажмите results. В разделе Plot по вкладке Home нажмите time. Каждая линия в графике представляет профиль веса опухоли каждого сценария симуляции.
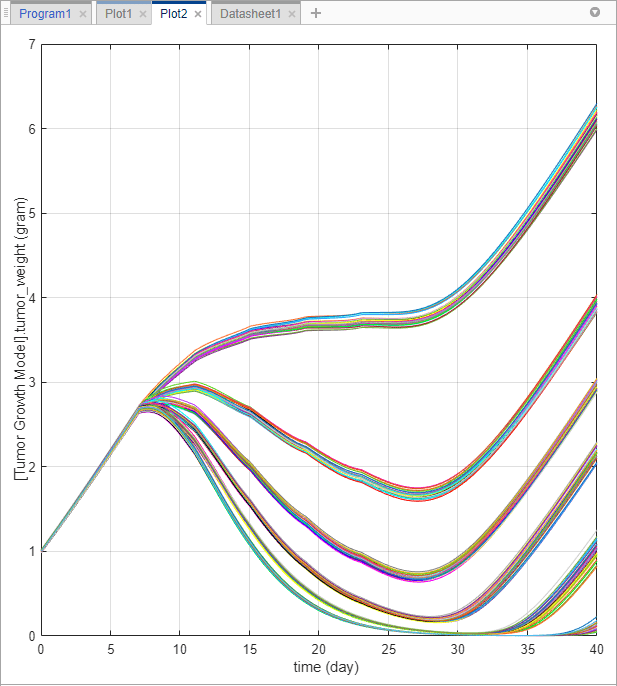
График показывает, что профили веса опухоли появляются в группах, соответствуя различным суммарным дозам. Чтобы лучше визуализировать это наблюдение, можно нарезать данные суммарными дозами. Для этого можно использовать таблицу Slice Data, которая содержит сводные данные режущих переменных, которые в настоящее время используются в графике и их соответствующих стилях графика.
Совет
Графики поддерживаются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками состояния. Когда данные (или экспериментальные данные или результаты симуляции) удалены или изменены, графики также обновляются согласно изменениям в базовых данных.
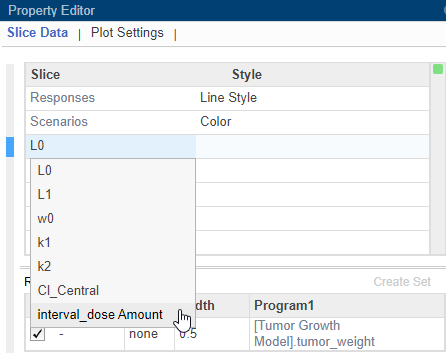
В таблице Slice Data дважды кликните L0 и выберите interval_dose Amount.
Совет
Можно нарезать данные с помощью различных переменных разрезания. Каждая переменная разрезания появляется в графике с различным визуальным стилем (или канал), такой как цвет, стиль линии и положение осей. Разрезание переменных может представлять атрибуты данных, такие как ответы или сценарии (то есть, группы или запуски симуляции). Разрезание переменных может также быть ковариантами или значениями параметров, сопоставленными со сценарием или группой. По умолчанию приложение обеспечивает режущие переменные для различных переменных отклика и различных сценариев в отображенных на графике данных. Можно добавить другие визуальные стили (или каналы) для наборов ответов и сопоставленного параметра или ковариационных переменных.
Дважды кликните пустую ячейку в столбце Style и выберите Grid.

Обновления приложения Plot2, чтобы показать каждую суммарную дозу на ее собственных осях.
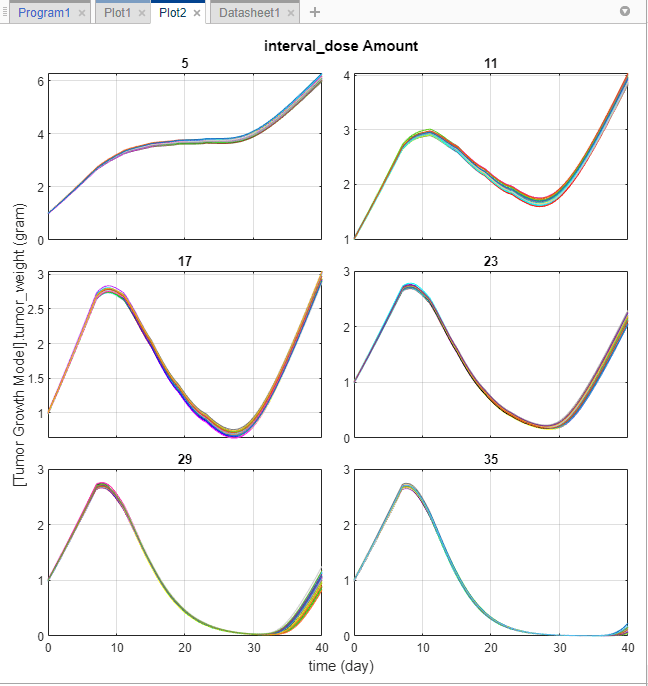
График показывает, что различные суммарные дозы проигрывают решающие роли в росте опухоли виртуальных пациентов. Из этого графика можно получить некоторое начальное понимание оптимальных суммарных доз и планирования дозы. Например, предположите, что целевой вес опухоли, чтобы достигнуть составляет 0,5 грамма. Результаты симуляции указывают, что суммарная доза 23 миллиграммов или больше может достигнуть той цели (можно подстроить область значений суммарных доз далее путем тонкой настройки его на шаге Generate Samples программы). Можно использовать эту информацию в сочетании с существующей информацией о лекарственной токсичности (не обсужденный в этом примере), чтобы получить режим дозирования, который удовлетворяет обоим требованиям эффективности и безопасности, например.
Можно также постобработать результаты симуляции, чтобы посмотреть на корреляцию между суммарными дозами и весом опухоли в другом отношении.
Вернитесь к программе путем нажатия на вкладку Program1. Кликните (+) значок около верхней части программы и выберите Calculate Observables под Postprocessing.
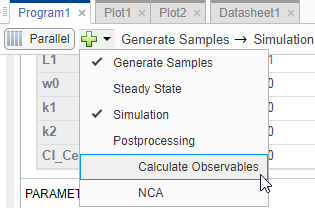
Шаг Postprocessing: Calculate Observables появляется ниже шага Simulation.
Дважды кликните первую ячейку в столбце Name. Введите stat1.
В столбце Expression введите следующее выражение: min(vertcat(nan,tumor_weight(time>=7))). Выражение возвращает минимальный вес опухоли после первой дозы, примененной в день 7 от каждой симуляции.
Примечание
Каждый раз, когда вы добавляете выражение в таблицу Observables на шаге, выражение автоматически добавляется как observable возразите против соответствующей модели.
В столбце Units введите gram.
Повторно выполните шаг симуляции путем щелкания по кнопке Run наверху шага. Приложение симулирует и оценивает stat1 выражение для каждого сценария симуляции или итерации, и генерирует следующий график. x - ось представляет параметры и y - ось представляет минимальный вес опухоли. Каждая точка представляет сценарий симуляции. График также показывает, что нет никакой корреляции между размером опухоли и значениями различных параметров модели, за исключением суммарных доз (самый правый подграфик).
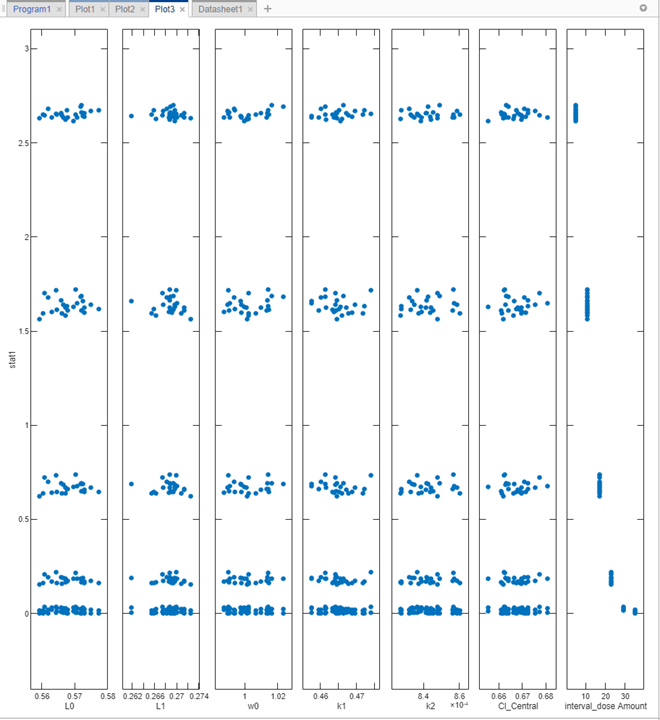
Покажите только подграфик суммарной дозы. В Plot Settings снимите все флажки в таблице Parameters, кроме interval_dose Amount.
В разделе Grid выберите both, чтобы показать сетки и для x - и для y - ось.
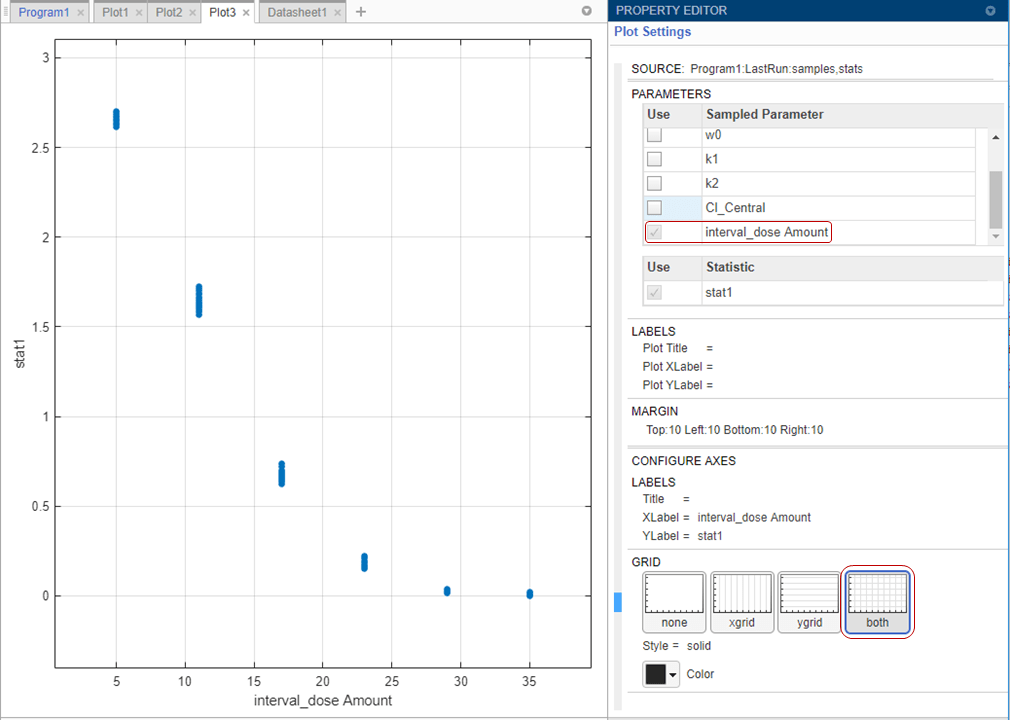
График подтверждает, что более высокие суммарные дозы управляют ростом опухоли лучше. Эти начальные результаты симуляции указывают, что суммарные дозы 23 мг или больше могли достигнуть гипотетического порога веса опухоли 0,5 граммов или ниже. Можно далее настроить область значений суммарных доз на шаге Generate Samples.
Совет
Чтобы в интерактивном режиме исследовать отображенные на графике данные, экспорт график к отдельному окну рисунка путем выбора Export Plot из контекста (щелкают правой кнопкой) по меню графика.
Этот пример показывает вам, как сгенерировать выборки, чтобы представлять виртуальных пациентов и выполнить симуляции Монте-Карло, чтобы исследовать ответ модели на росте опухоли под различными суммарными дозами. Результаты симуляции указывают на область значений для суммарных доз и расписаний дозы, который управляет ростом опухоли для различных виртуальных пациентов. Вы могли далее настроить режимы дозирования так, чтобы дозы остались в некоторых известных порогах эффективности и токсичности. Для подобного анализа смотрите, что Режимы дозирования Скана в качестве примера Используют Приложение SimBiology Model Analyzer.
[1] Simeoni, M., П. Магни, К. Кэммия, Г. Де Николао, V. Шафраны, Э. Пезенти, М. Джермани, я. Poggesi и М. Роккетти. 2004. Прогнозирующее фармакокинетическо-фармакодинамическое моделирование кинетики роста опухоли в моделях ксенотрансплантата после администрирования противораковых агентов. Исследования рака. 64:1094-1101.
Observable | SimBiology Model Analyzer