Браузер NGS позволяет визуально проверять и исследовать выравнивание считываемых последовательностей с эталонной последовательностью в поддержку анализа, который измеряет генетические вариации и экспрессию генов. Браузер NGS позволяет:
Визуализация считывания последовательности в соответствии с нуклеотидной эталонной последовательностью.
Сравнение нескольких наборов данных, выровненных по общей ссылочной последовательности.
Просмотр покрытия различных баз и областей ссылочной последовательности.
Исследовать качество и другие детали выровненных чтений.
Выявление несоответствий из-за ошибок или полиморфизмов, вызывающих базовые вызовы.
Визуализация вставок и удалений.
Извлеките аннотации элементов относительно определенной области последовательности привязок.
Исследовать области, представляющие интерес для выравнивания, определяемые различными анализами.
Можно визуализировать и исследовать выровненные данные до, во время или после любой предварительной обработки (фильтрация, повторная калибровка качества) или шагов анализа, выполняемых для выровненных данных.
Чтобы открыть браузер NGS, введите в окне команд MATLAB следующее:
ngsbrowser
Либо щелкните Обозреватель NGS (NGS Browser) на вкладке Приложения (Apps).
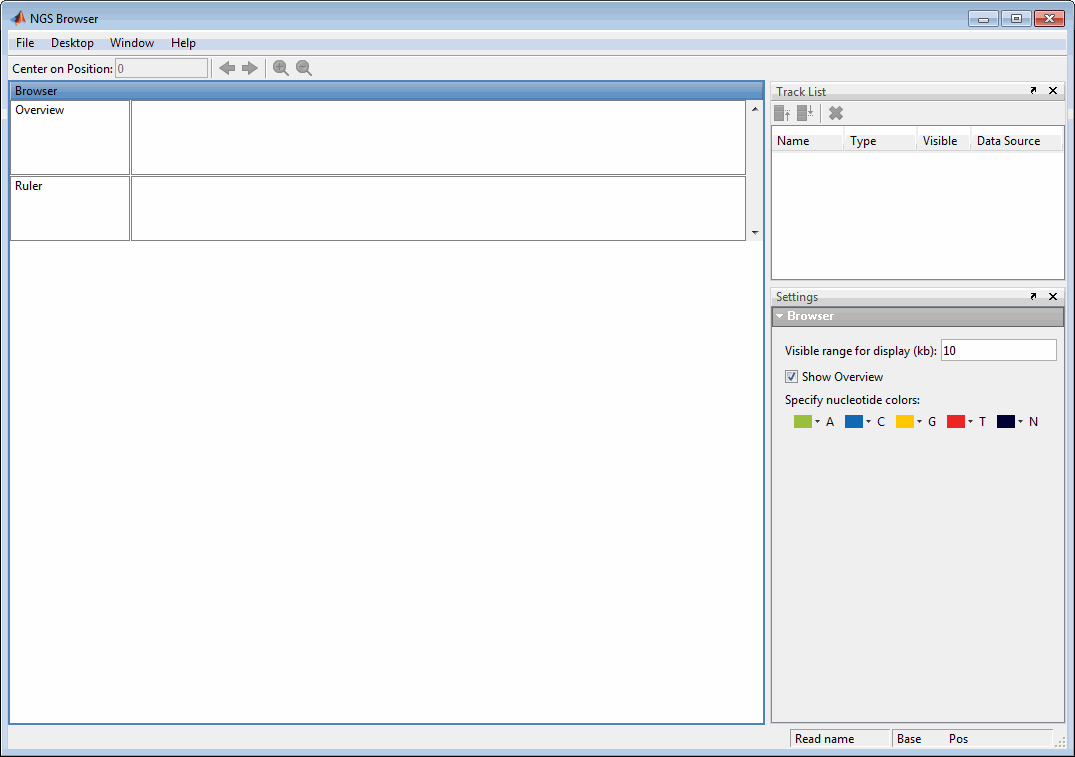
Отображение в браузере опорной дорожки, одной дорожки трассы и одной дорожки аннотации
В обозреватель NGS можно импортировать одну ссылочную последовательность. Ссылочная последовательность должна находиться в файле FASTA.
Выберите меню «Файл» > «Добавить данные из файла».
В диалоговом окне Открыть (Open) выберите файл FASTA и нажмите кнопку Открыть (Open).
Совет
Вы можете использовать getgenbank функции с помощью ToFile и SequenceOnly аргументы пары «имя-значение» для получения ссылочной последовательности из базы данных GenBank и сохранения ее в файле в формате FASTA.
Можно импортировать несколько наборов данных последовательности считывания данных трассы. Данные выравнивания должны находиться в одном из следующих элементов:
BioMap объект
Совет
Создайте объект BioMap из файла в формате SAM- или BAM для исследования, подмножества и фильтрации данных перед их импортом в браузер NGS.
Файл в формате SAM или BAM
Примечание
Файл в формате SAM или BAM должен:
Иметь записи, упорядоченные по начальной позиции в ссылочной последовательности.
Файл индекса IDX (для файла в формате SAM) или файлы индекса BAI и LINEARINDEX (для файла в формате BAM) хранятся в том же расположении, что и исходный файл. В противном случае исходный файл должен храниться в папке, к которой имеется доступ для записи, поскольку MATLAB ® должен создавать и хранить индексные файлы в этой папке.
Совет
Попробуйте использовать SAMtools, чтобы проверить, упорядочены ли чтения в файле в формате SAM- или BAM по позициям в последовательности ссылок, а также, при необходимости, переупорядочить их.
Совет
Если индексные файлы (IDX или BAI и LINEARINDEX) не хранятся в том же месте, что и исходный файл, а исходный файл хранится в месте, к которому отсутствует доступ для записи, импорт данных из исходного файла непосредственно в браузер невозможен. Вместо этого создайте BioMap из исходного файла с помощью IndexDir аргумент пары имя-значение, а затем импортируйте объект BioMap в браузер.
Чтобы импортировать последовательность, прочтите данные трассы:
Выберите меню «Файл» > «Добавить данные из файла» или «Файл» > «Импортировать данные трассы из рабочего пространства MATLAB».
Выберите файл в формате SAM, файл в формате BAM или объект BioMap.
Если выбран файл, содержащий несколько последовательностей ссылок, в диалоговом окне Выбрать ссылку (Select Reference) выберите ссылку или отсканируйте файл на наличие доступных ссылок и их сопоставленные счетчики чтения. Нажмите кнопку ОК.
Повторите предыдущие шаги для импорта дополнительных наборов данных.
Можно импортировать несколько наборов аннотаций элементов из файлов в формате GFF- или GTF, содержащих данные для одной ссылочной последовательности.
Выберите меню «Файл» > «Добавить данные из файла».
В диалоговом окне Открыть (Open) выберите файл в формате GFF- или GTF и нажмите кнопку Открыть (Open).
Повторите предыдущие шаги для импорта дополнительных аннотаций.
Для увеличения и уменьшения масштаба:
| Используйте |
| или перетащите кромку резиновой области в области Обзор (Overview), удерживая нажатой кнопку мыши. |
|
|
Для панорамирования по трассе:
| Используйте |
| или перетащите rubberband в области Обзор (Overview). |
|
|
Совет
Используйте клавиши со стрелками влево и вправо для панорамирования с шагом в одну базовую пару (bp).
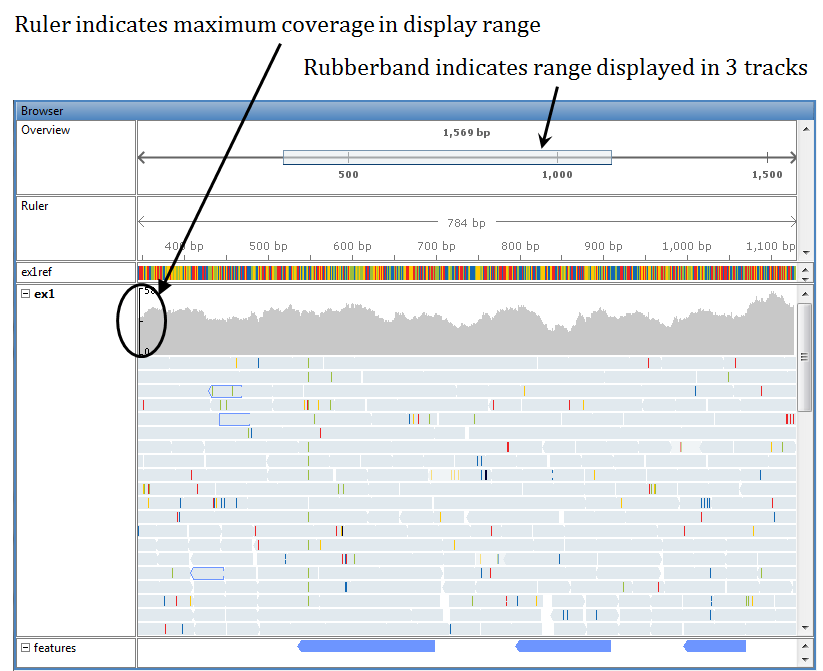
В верхней части каждой дорожки трассы на виде покрытия отображается покрытие каждой базы в ссылочной последовательности. Вертикальная линейка на левом краю вида покрытия указывает максимальное покрытие в диапазоне отображения. Наведите указатель мыши на позицию в представлении покрытия для отображения местоположения и счетчиков.
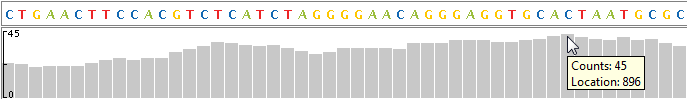
Примечание
Браузер вычисляет зону покрытия при разрешении базовой пары, а не при биннинге, даже при увеличении.
Чтобы изменить отображаемый процент покрытия, щелкните в любом месте трассы, а затем измените параметры покрытия трассы.
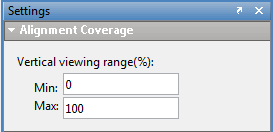
Совет
Установите значение Max больше 100, если необходимо, при сравнении охвата нескольких дорожек чтения.
Каждая дорожка выравнивания включает в себя вид набора коротких считываний, выровненных по ссылочной последовательности.
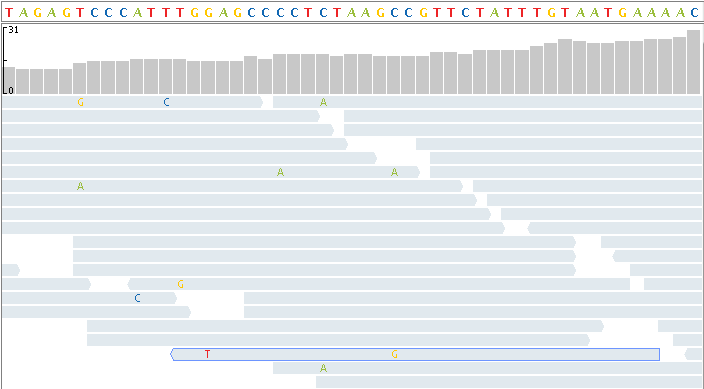
Ограничьте глубину считываний, отображаемых на виде сваи, установив максимальную глубину считывания отображения в настройках «Трасса свай».
Совет
Ограничение глубины коротких считываний в ракурсе pileup не изменяет счетчиков, отображаемых в ракурсе покрытия.
Сравнение нескольких наборов данных, с каждым набором данных в своей собственной дорожке, с общей ссылочной последовательностью. Список дорожек используется для отображения/скрытия, упорядочивания и удаления дорожек данных.
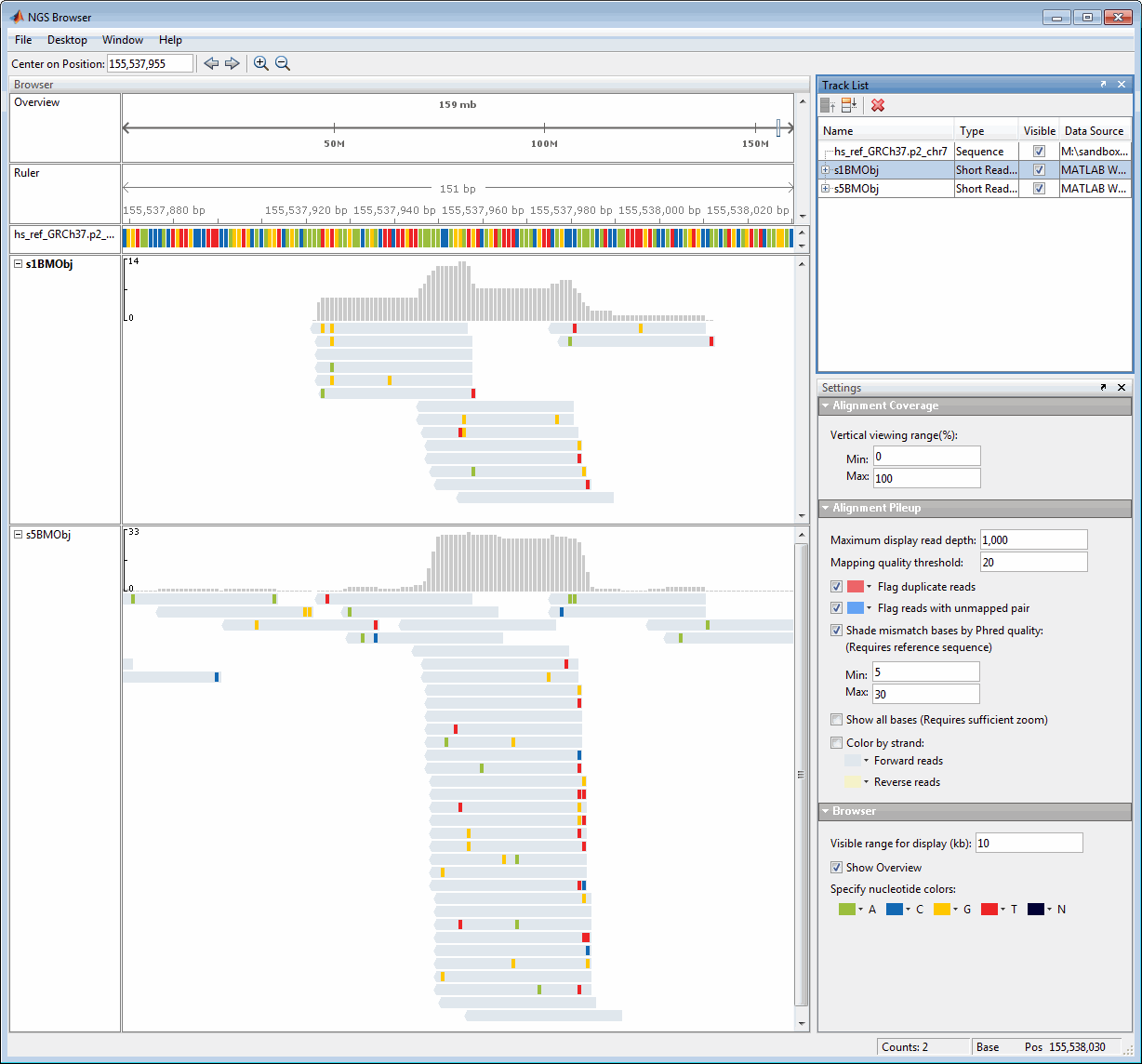
Наведите указатель мыши на позицию в режиме чтения, чтобы отобразить информацию о направлении, местоположении, качестве и сопоставлении пряди для базы, считанного объекта и его спаренной пары.

Щелкните в любом месте трассы, чтобы отобразить параметры «Свайная трасса трассы».
Установите пороговое значение качества отображения в разделе «Трасса пиловок», чтобы отметить низкокачественные чтения. Чтения с качеством отображения ниже этого уровня отображаются более светлым оттенком серого.
Выберите Флажок повторяющиеся чтения и выберите цвет контура.
Выберите пункт Flag reads with unmapped pair и выберите цвет контура.
Несовпадения отображаются в виде цветных блоков или букв в зависимости от уровня масштабирования.
Увеличенный вид чтения - несовпадения отображаются в виде полос
![]()
Увеличено в виду чтения - несовпадения отображаются в виде букв
![]()
В дополнение к базовой информации о качестве Phred, которая отображается во всплывающей подсказке, можно визуализировать различия в качестве с помощью баз несоответствия Shade по параметрам качества Phred.
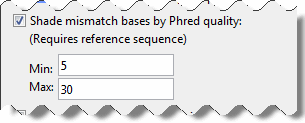
Блоки несоответствия или буквы отображаются в:
Светлый оттенок - несовпадение баз с показателями Phred ниже минимального
Градуировка средних оттенков - несовпадение баз с показателями Phred в диапазоне от минимального до максимального
Темный оттенок - несовпадение баз с показателями Phred выше максимума
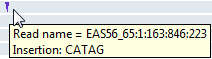
Обозреватель NGS обозначает вставки![]() символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию о нем.
символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию о нем.
Обозреватель NGS обозначает удаления тире.

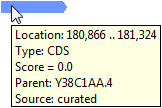
После импорта файла аннотаций элементов можно выполнять масштабирование и панорамирование для просмотра аннотаций элементов, связанных с интересующей областью трассы. Наведите указатель мыши на аннотацию элемента.
Распечатайте или экспортируйте изображение браузера, выбрав меню «Файл» > «Печать изображения» или «Файл» > «Экспорт изображения».