Этот NGS Browser позволяет вам визуально проверить и исследовать согласование показаний последовательности с ссылкой последовательностью в поддержку анализов, которые измеряют генетические изменения и экспрессию генов. Этот NGS Browser позволяет вам:
Визуализируйте показания последовательности, выровненные по нуклеотидной эталонной последовательности.
Сравните несколько наборов данных, выровненных по общей ссылочной последовательности.
Просмотрите покрытие различных основ и областей ссылки последовательности.
Исследуйте качество и другие детали выровненных показаний.
Идентифицируйте несоответствия из-за ошибок вызова базы или полиморфизмов.
Визуализация вставок и удалений.
Извлеките функцию аннотации относительно определенной области ссылки последовательности.
Исследуйте необходимые области в выравнивании, определяемые различными анализами.
Можно визуализировать и исследовать выровненные данные до, во время или после любой предварительной обработки (фильтрация, перекалибровка качества) или шагов анализа, которые вы выполняете с выровненными данными.
Чтобы открыть NGS Browser, введите следующее в Командном окне MATLAB:
ngsbrowser
Также щелкните NGS Browser на вкладке Apps.
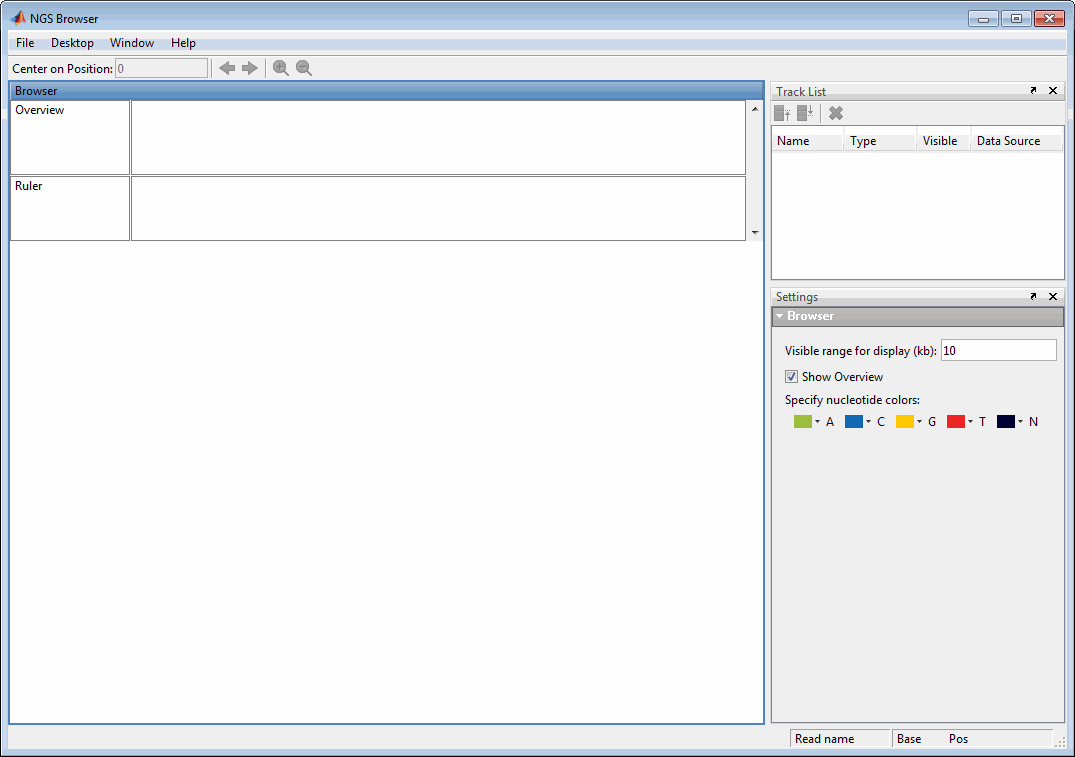
Браузер, отображающий ссылочную дорожку, одну трассу выравнивания и одну трассу аннотации
Можно импортировать одну последовательность ссылок в NGS Browser. Ссылочная последовательность должна быть в файле FASTA.
Выберите File > Add Data from File.
В диалоговом окне «Открыть» выберите файл FASTA и нажмите кнопку Open.
Совет
Вы можете использовать getgenbank функция со ToFile и SequenceOnly аргументы пары "имя-значение" для извлечения ссылочной последовательности из базы данных GenBank и сохранения ее в файл в формате FASTA.
Можно импортировать несколько наборов данных последовательности считанных данных выравнивания. Данные выравнивания должны быть следующими:
BioMap объект
Совет
Создайте объект BioMap из файла в формате SAMSUNG или BAM, чтобы исследовать, подмножествовать и фильтровать данные перед импортом в NGS Browser.
САМ- или BAM-форматированный файл
Примечание
Ваш сам- или BAM-форматированный файл должен:
Иметь чтения, упорядоченные по начальной позиции в ссылочной последовательности.
Иметь файл индекса IDX (для файла в формате SAM) или файлы индекса BAI и LINEARINDEX (для файла в формате BAM), хранящиеся в том же месте, что и ваш исходный файл. В противном случае исходный файл должен храниться в том месте, в которое вы имеете доступ для записи, потому что MATLAB® необходимо создать и сохранить файлы индекса в этом расположении.
Совет
Попробуйте с помощью SAMtools проверить, упорядочены ли чтения в файле с форматированием САМ- или БАМ по позициям в ссылочной последовательности, а также при необходимости переупорядочить их.
Совет
Если у вас нет индексы файлов (IDX или BAI и LINEARINDEX), хранящихся в том же месте, что и ваш исходный файл, и ваш исходный файл хранится в том месте, в которое вы не имеете доступа для записи, вы не можете импортировать данные из исходного файла непосредственно в браузер. Вместо этого создайте BioMap объект из исходного файла с помощью IndexDir аргумент пары "имя-значение", а затем импортируйте объект BioMap в браузер.
Для импорта последовательности считайте данные выравнивания:
Выберите File > Add Data from File или File > Import Alignment Data from MATLAB Workspace.
Выберите файл в формате SAM, файл в формате BAM или объект BioMap.
Если вы выбираете файл, содержащий несколько ссылочных последовательностей, в диалоговом окне Выбрать ссылку (Select Reference) выберите ссылку или просканируйте файл на наличие доступных ссылок и их сопоставленных отсчетов чтения. Нажмите OK.
Повторите предыдущие шаги, чтобы импортировать дополнительные наборы данных.
Можно импортировать несколько наборов функции аннотаций из файлов в формате GFF- или GTF, которые содержат данные для одной последовательности ссылки.
Выберите File > Add Data from File.
В диалоговом окне «Открыть» выберите файл в формате GFF- или GTF и нажмите Open.
Повторите предыдущие шаги, чтобы импортировать дополнительные аннотации.
Для масштабирования и уменьшения:
| Используйте |
| или перетащите ребро резинки в области «Обзор». |
|
|
Для панорамирования вдоль выравнивания:
| Используйте |
| или перетащите резинку в область Обзор (Overview). |
|
|
Совет
Используйте клавиши со стрелами влево и вправо для панорамирования с шагом в одну базовую пару (bp).
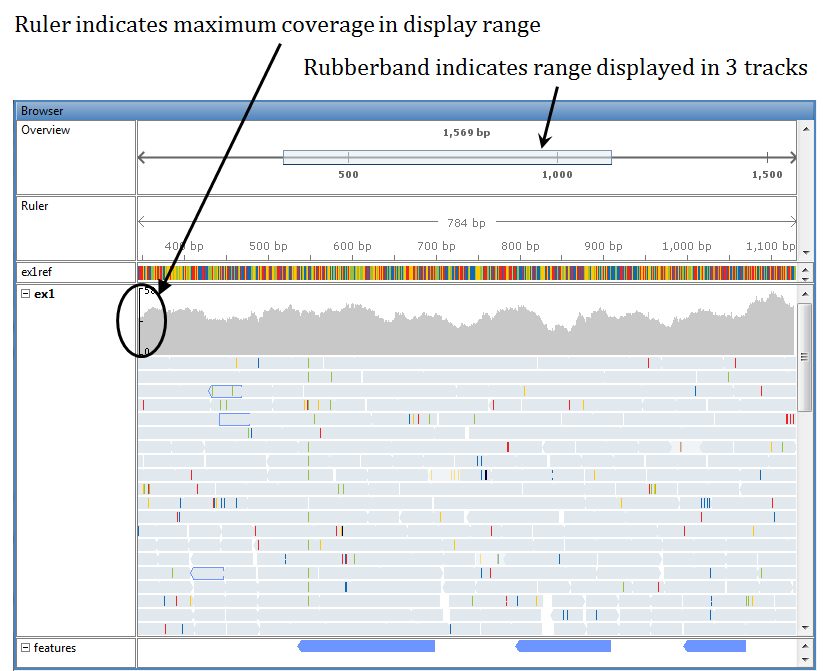
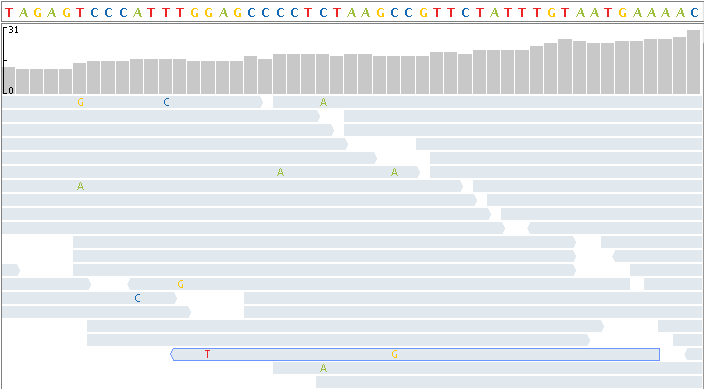
В верхней части каждой дорожки выравнивания в виде покрытия отображается покрытие каждой основы в ссылочной последовательности. Вертикальная линейка на левом крае вида покрытия указывает максимальное покрытие в области значений отображения. Наведите указатель мыши на положение в виде переадресации, чтобы отобразить местоположение и счетчики.
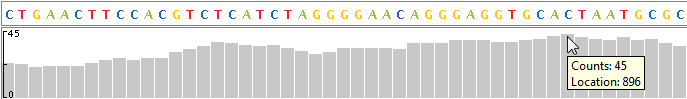
Примечание
Браузер вычисляет покрытие при разрешении базовой пары, вместо раскладывания, даже при масштабировании.
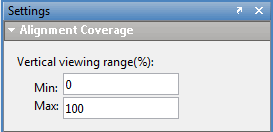
Чтобы изменить процент покрытия отображаемого, щелкните в любом месте выравнивания дорожки, а затем измените параметры Выравнивания покрытия.
Совет
Установите Max значение, больше 100, если необходимо, при сравнении покрытия нескольких дорожек показаний.
Каждая трасса выравнивания включает вид сваи коротких показаний, выровненных по ссылочной последовательности.
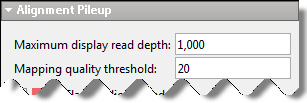
Ограничьте глубину показаний, отображаемых в виде сваи, путем установки Maximum display read depth в настройках Выравнивания Pileup.
Совет
Ограничение глубины коротких показаний в представлении сваи не изменяет счетчики, отображаемые в представлении покрытия.
Сравните несколько наборов данных с каждым набором данных в своей собственной дорожке с общей эталонной последовательностью. Используйте Track List, чтобы показать/скрыть, упорядочить и удалить треки данных.
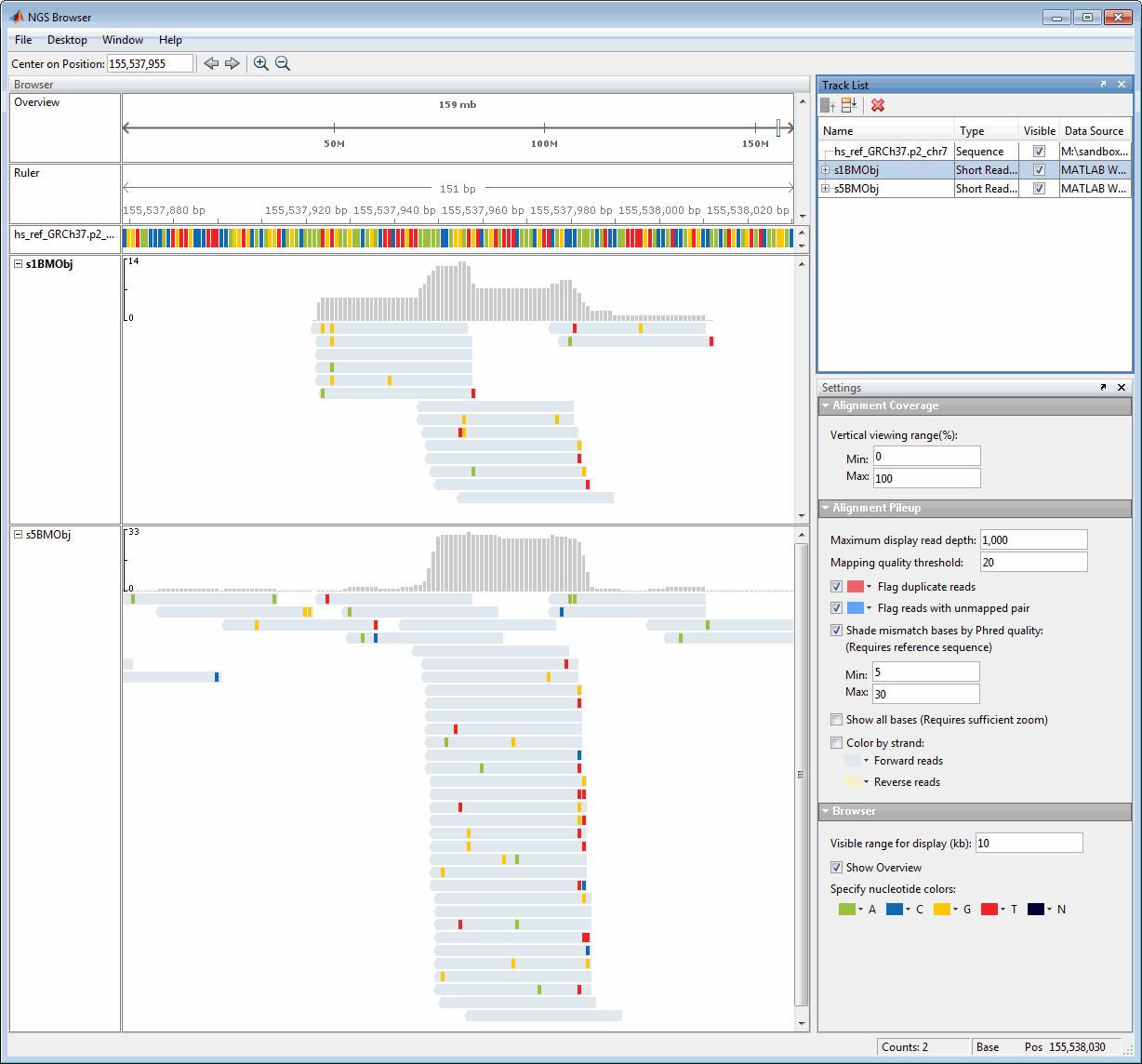
Наведите указатель мыши на положение в считанном направлении, чтобы отобразить направление цепи, местоположение, качество и отображение информацию для основы, чтения и его парного соединения.

Щелкните в любом месте выравнивания дорожки, чтобы отобразить Выравнивание настройки Pileup.
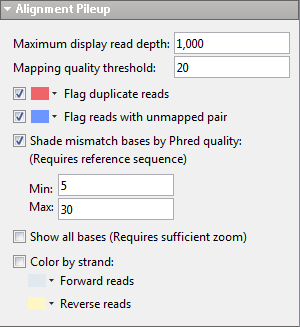
Установите Mapping quality threshold в разделе Alignment Pileup, чтобы отметить низкокачественные чтения. Чтения с качеством отображения ниже этого уровня появляются в более легком оттенке серого.
Выберите Flag duplicate reads и цвет контура.
Выберите Flag reads with unmapped pair и цвет контура.
Несоответствия отображаются как цветные блоки или буквы, в зависимости от уровня масштаба.
Изменение масштаба представления read - Несоответствия отображаются как полосы
![]()
Масштабирование с учетом чтения - Несоответствия отображаются как буквы
![]()
В дополнение к базовой информации о качестве Phred, которая отображается в подсказке, можно визуализировать различия в качестве с помощью настроек Shade mismatch bases by Phred quality.
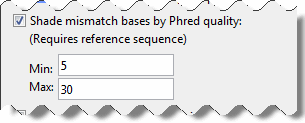
Блоки или буквы несоответствия отображаются в:
Светлый оттенок - Несоответствие основ с Phred счетов ниже минимума
Градация средних оттенков - Несоответствие основ с Фредом счета в пределах минимального к максимальной области значений
Темный оттенок - Несоответствие основ с Phred счетов выше максимума
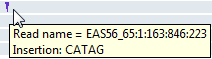
Этот NGS Browser обозначает вставки![]() символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию о нем.
символом. Наведите указатель мыши на символ вставки, чтобы отобразить информацию о нем.
Удаление NGS Browser обозначается штрихами.

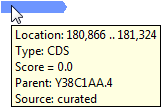
После импорта файла аннотации функции можно масштабировать и панорамирование, чтобы просмотреть функции аннотации, связанные с необходимой областью в выравнивание. Наведите указатель мыши на аннотацию функции.
Распечатайте или экспортируйте изображение браузера, выбрав File > Print Image или File > Export Image.