Создайте агломеративные кластеры из данных
T = clusterdata(X,cutoff)X, учитывая пороговое значение cutoff для вырезания агломеративного иерархического дерева, которое linkage функция генерирует из X.
clusterdata поддерживает агломеративную кластеризацию и включает в себя pdist, linkage, и cluster функций, которые можно использовать отдельно для более детального анализа. Для получения дополнительной информации смотрите Описание алгоритма.
Если 'Linkage' является 'centroid' или 'median', затем linkage может создать дерево кластеров, которое не является монотонным. Этот результат происходит, когда расстояние от объединения двух кластеров, r и s, до третьего кластера меньше, чем расстояние между r и s. В этом случае в дендрограмме, нарисованной с ориентацией по умолчанию, путь от листа к корневому узлу делает несколько шагов вниз. Чтобы избежать этого результата, задайте другое значение для 'Linkage'. На следующем изображении показано немонотонное дерево кластеров.
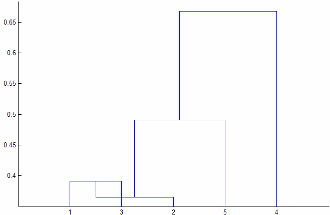
В этом случае кластер 1 и кластер 3 соединяются в новый кластер, в то время как расстояние между этим новым кластером и кластером 2 меньше, чем расстояние между кластером 1 и кластером 3.
Если вы задаете значение c для cutoff входной параметр, затем T = clusterdata(X, c)
Создайте вектор Евклидова расстояния между парами наблюдений в X при помощи pdist.
Y = pdist(X, 'euclidean')
Создайте агломеративное иерархическое дерево кластеров из Y при помощи linkage с 'single' способ вычисления кратчайшего расстояния между кластерами.
Z = linkage(Y, 'single')
Если 0 < c < 2, использование cluster для определения кластеров из Z когда несогласованные значения меньше c.
T = cluster(Z, 'Cutoff', c)
Если c является целым числом значения ≥ 2, использование cluster чтобы найти максимум c кластеры из Z.
T = кластер (Z, 'MaxClust', c)
Если у вас есть иерархическое дерево кластеров Z (выходные выходы linkage функция для матрицы входных данных X), вы можете использовать cluster для выполнения агломеративной кластеризации на Z и возвращает назначение кластера для каждого наблюдения (строки) в X.
cluster | dendrogram | inconsistent | kmeans | linkage | pdist