Постройте график Ramachandran для данных о Банке данных белка (PDB)
ramachandran(PDBid)
ramachandran(File)
ramachandran(PDBStruct)
RamaStruct = ramachandran(...)
ramachandran(..., 'Chain', ChainValue,
...)
ramachandran(..., 'Plot', PlotValue,
...)
ramachandran(..., 'Model', ModelValue,
...)
ramachandran(..., 'Glycine', GlycineValue,
...)
ramachandran(..., 'Regions', RegionsValue,
...)
ramachandran(..., 'RegionDef', RegionDefValue,
...)
PDBid | Вектор символов или строка, задающая уникальный идентификатор для структуры белка, записывают в базе данных PDB. Примечание Каждая структура в базе данных PDB представлена алфавитно-цифровым идентификатором с четырьмя символами. Например, |
File | Вектор символов или строка, задающая имя файла или путь и имя файла. Файл, на который ссылаются, является Банком данных белка (PDB) - отформатированный файл. Если вы задаете только имя файла, тот файл должен быть на MATLAB® путь поиска файлов или в Текущем каталоге MATLAB. |
PDBStruct | Структура MATLAB, содержащая PDB-отформатированные-данные, такой, как возвращено getpdb или pdbread. |
ChainValue | Вектор символов, строка, представляет в виде строки вектор или массив ячеек из символьных векторов, который задает цепь (цепи), чтобы вычислить углы скрученности для и график. Выбор:
|
PlotValue | Вектор символов или строка, задающая, как построить цепи. Выбор:
|
ModelValue | Целое число, которое задает модель структуры, чтобы рассмотреть. Значением по умолчанию является 1. |
GlycineValue | Управляет выделением глициновых остатков с кругом в графике. Выбором является true или false (значение по умолчанию). |
RegionsValue | Управляет рисунком областей ссылки Ramachandran в графике. Выбором является Области по умолчанию являются базовой предназначенной для правой руки альфой, базовой бетой, базовой предназначенной для левой руки альфой, и позволенный, с базовыми областями, соответствующими точкам данных предпочтительных значений psi/phi угловых пар и позволенным областям, соответствующим возможным, но порицаемым значениям psi/phi угловых пар, на основе простых энергетических факторов. Контуры этих областей по умолчанию основаны на вычислениях Моррисом и др., 1992. Примечание При использовании палитры по умолчанию, красной = предназначенная для правой руки базовая альфа, базовая бета и базовая предназначенная для левой руки альфа, в то время как желтый = позволенный. |
RegionDefValue | Структура MATLAB или массив структур (при определении нескольких областей) содержащий информацию (имя, цвет и контуры) для пользовательских ссылочных областей в графике Ramachandran. Каждая структура должна содержать следующие поля:
Совет Если вы задаете пользовательские ссылочные области, в которых меньшая область содержится или покрывается более крупной областью, перечислите структуру для меньшей области сначала в массиве так, чтобы это было построено последнее и видимое в графике. |
RamaStruct | Структура MATLAB или массив структур (если белок содержит кратные цепи). Каждая структура содержит следующие поля:
Для описаний полей см. следующую таблицу. |
График Ramachandran является графиком угла скрученности phi, Φ, (угол скрученности между C-N-CA-C атомы) по сравнению с углом скрученности psi, Ψ, (угол скрученности между N-CA-C-N атомы) для каждого остатка последовательности белка.
ramachandran( генерирует график Ramachandran для белка, заданного идентификатором базы данных PDB PDBid)PDBid.
ramachandran( генерирует график Ramachandran для белка, заданного File)File, PDB-отформатированный файл.
ramachandran( генерирует график Ramachandran для белка, сохраненного в PDBStruct)PDBStruct, структура MATLAB, содержащая PDB-отформатированные-данные, такой, как возвращено getpdb или pdbread.
RamaStruct = ramachandran(...)
| Поле | Описание |
|---|---|
Angles | Матрица с тремя столбцами, содержащая углы скрученности phi (Φ), psi (Ψ), и омега (ω) для каждого остатка в последовательности, упорядоченной порядковым номером остатка. Количество строк в матрице равно количеству строк в Примечание
|
ResidueNum | Вектор-столбец, содержащий порядковые номера остатка из файла PDB. Примечание
Углы перечислены в |
ResidueName | Вектор-столбец, содержащий остаток, называет для белка. |
Chain | Вектор символов или строка, задающая цепи в белке. |
HPoints | Обработайте к точкам данных в графике. |
ramachandran (..., ' вызовы PropertyName', PropertyValue, ...)ramachandran с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
ramachandran(..., 'Chain', задает цепь (цепи), чтобы вычислить углы скрученности для и график. Выбор:ChainValue,
...)
'All' (значение по умолчанию) — Углы скрученности для всех цепей вычислены и построены.
Вектор символов или строка, задающая цепочечный ID, который является чувствительным к регистру.
Массив ячеек из символьных векторов или вектор строки, задающий цепочечные идентификаторы, которые являются чувствительными к регистру.
ramachandran(..., 'Plot', задает, как построить цепи. Выбор:PlotValue,
...)
'None' — Графики ничто.
'Separate' — Углы скрученности графиков для всех заданных цепей в отдельных графиках.
'Combined' (значение по умолчанию) — Углы скрученности графиков для всех заданных цепей в одном построенном графике.
ramachandran(..., 'Model', задает модель структуры, чтобы рассмотреть. Значением по умолчанию является ModelValue,
...)1.
ramachandran(..., 'Glycine', управляет выделением глициновых остатков с кругом в графике. Выбором является GlycineValue,
...)true или false (значение по умолчанию).
ramachandran(..., 'Regions', управляет рисунком областей ссылки Ramachandran в графике. Выбором является RegionsValue,
...)true или false (значение по умолчанию).
Области по умолчанию являются базовой предназначенной для правой руки альфой, базовой бетой, базовой предназначенной для левой руки альфой, и позволенный, с базовыми областями, соответствующими точкам данных предпочтительных значений psi/phi угловых пар и позволенным областям, соответствующим возможным, но порицаемым значениям psi/phi угловых пар, на основе простых энергетических факторов. Контуры этих областей по умолчанию основаны на вычислениях Моррисом и др., 1992.
Примечание
При использовании палитры по умолчанию, затем красной = базовая предназначенная для правой руки альфа, базовая бета и базовая предназначенная для левой руки альфа, в то время как желтый = позволенный.
ramachandran(..., 'RegionDef', указывает информацию (имя, цвет и контур) для пользовательских ссылочных областей в графике Ramachandran. RegionDefValue,
...)RegionDefValue структура MATLAB или массив структур, содержащих следующие поля:
Name — Вектор символов или строка, задающая имя для области.
Color — Вектор символов или строка или трехэлементный числовой вектор из значений RGB, задающих цвет для области в графике.
Patch — 2 N матрицей значений, первая строка, содержащая угол скрученности phi (Φ) значения и вторая строка, содержащая угол скрученности psi (Ψ) значения. Когда psi/phi угловые пары построены, точки данных задают контур для области. N является количеством точек данных, должен был задать область.
Совет
Если вы задаете пользовательские ссылочные области, в которых меньшая область содержится или покрывается более крупной областью, перечислите структуру для меньшей области сначала в массиве так, чтобы это было построено последнее и видимое в графике.
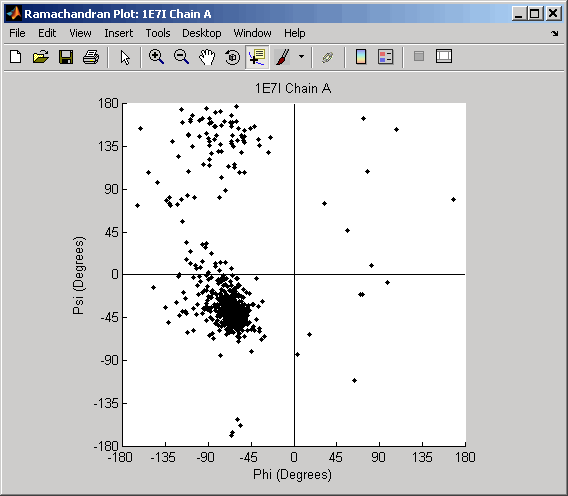
Постройте график Ramachandran для человеческого альбумина сыворотки, объединенного с octadecanoic кислотой, которая имеет идентификатор базы данных PDB 1E7I.
ramachandran('1E7I')
Используйте getpdb функция, чтобы получить данные о структуре белка для человеческого соматотропина от базы данных PDB и сохранить информацию к файлу.
getpdb('1a22','ToFile','1a22.pdb');Вычислите углы скрученности и постройте график Ramachandran для цепи человеческого соматотропина, представленного в pdb файле, 1a22.pdb.
ChainA1a22Struct = ramachandran('1a22.pdb','chain','A')
ChainA1a22Struct =
Angles: [191x3 double]
ResidueNum: [191x1 double]
ResidueName: {191x1 cell}
Chain: 'A'
HPoints: 370.0012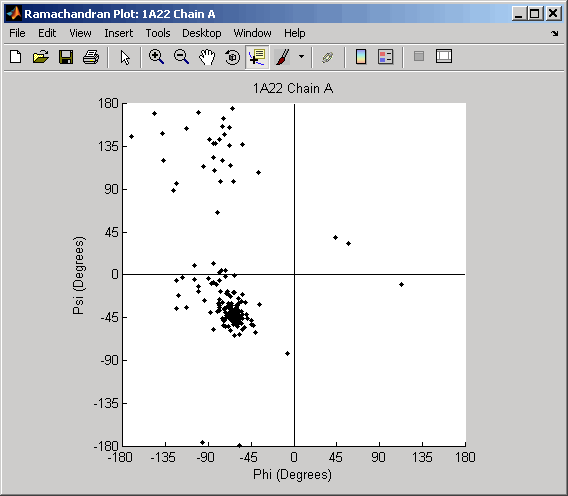
Используйте getpdb функция, чтобы получить данные о структуре белка для человеческого соматотропина от базы данных PDB и хранить информацию в структуре.
Struct1a22 = getpdb('1a22');Постройте построенный Ramachandran для всех цепей человеческого соматотропина, представленного в pdb структуре, 1a22Struct. Подсветите глициновые остатки (с кругом) и чертите ссылочные области Ramachandran в графике.
ramachandran(Struct1a22,'glycine',true,'regions',true);
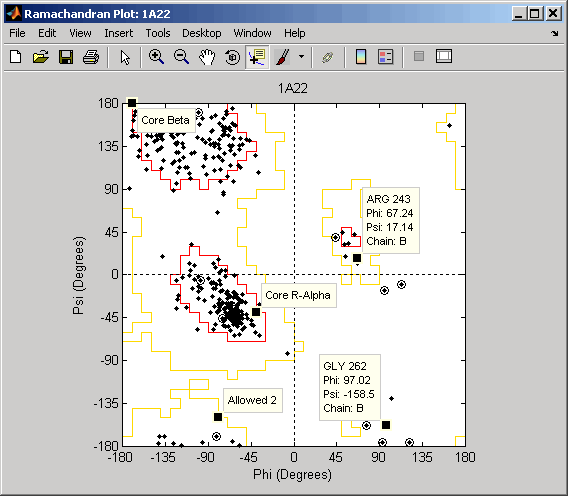
Совет
Кликните по точке данных, чтобы отобразить всплывающую подсказку с информацией об остатке. Кликните по области, чтобы отобразить всплывающую подсказку, задающую область. Нажмите и удержите клавишу Alt, чтобы отобразить несколько всплывающих подсказок.
Постройте отдельный график Ramachandran для каждой цепи человеческого соматотропина, представленного в pdb структуре, 1a22Struct. Подсветите глициновые остатки (с кругом) и чертите ссылочные области Ramachandran в графике.
ramachandran(Struct1a22,'plot','separate','chain','all',...
'glycine',true,'regions',true)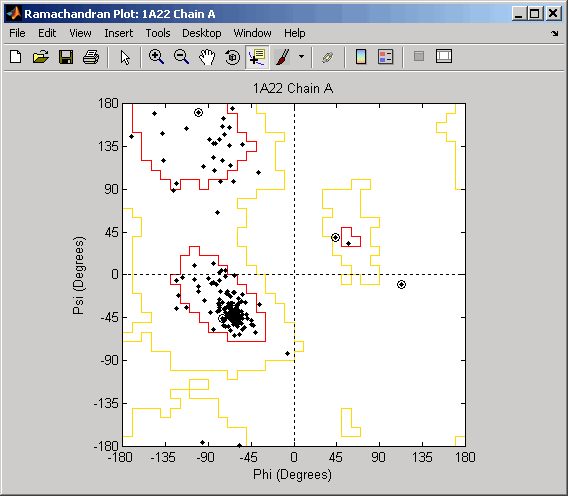
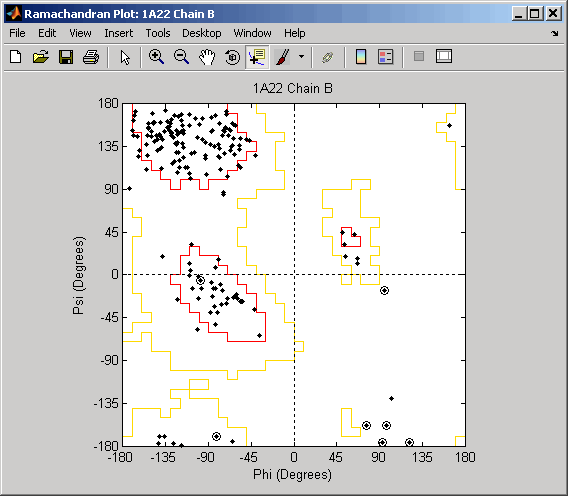
Создайте массив двух структур, содержащих углы скрученности для цепей A и D в киназе белка Calcium/Calmodulin-dependent, которая имеет идентификатор базы данных PDB 1hkx.
a = ramachandran('1hkx', 'chain', {'A', 'D'})
a =
1x2 struct array with fields:
Angles
ResidueNum
ResidueName
Chain
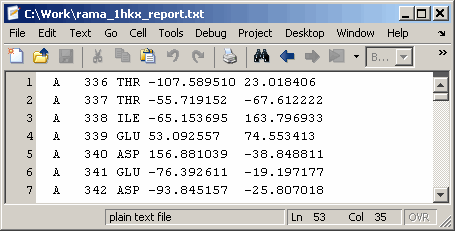
HPointsЗапишите разграниченный вкладкой файл отчета, содержащий углы скрученности phi (Φ) и psi (Ψ) для цепей A и D в киназе белка Calcium/Calmodulin-dependent.
fid = fopen('rama_1hkx_report.txt', 'wt');
for c = 1:numel(a)
for i = 1:length(a(c).Angles)
if ~all(isnan(a(c).Angles(i,:)))
fprintf(fid,'%s\t%d\t%s\t%f\t%f\n', a(c).Chain, ...
a(c).ResidueNum(i), a(c).ResidueName{i}, ...
a(c).Angles(i,1:2));
end
end
end
fclose(fid);
Просмотрите файл, который вы создали в редакторе MATLAB.
edit rama_1hkx_report.txt
[1] Моррис, A.L., Макартур, M.W., Хатчинсон, НАПРИМЕР, и Торнтон, J.M. (1992). Стереохимическое качество координат структуры белка. БЕЛКИ: структура, функция и генетика 12, 345–364.
getpdb | molviewer | pdbdistplot | pdbread | proteinpropplot