Симулируйте модель SimBiology, добавив изменения путем выборки ошибочной модели
[ возвращает результаты симуляции ynew,parameterEstimates]
= random(resultsObj)ynew с добавленным шумом с помощью ошибочной информации о модели задан resultsObj.ErrorModelInfo свойство и оцененные значения параметров parameterEstimates.
[ использует заданный ynew,parameterEstimates]
= random(resultsObj,data,dosing)data и dosing информация.
[ также применяет заданные варианты к каждой симуляции.ynew,parameterEstimates]=
random(resultsObj,data,dosing,'Variants',v)
Примечание
Шум только добавляется к состояниям, которые являются ответами, которые являются состояниями, включенными в responseMap входной параметр, когда вы вызвали sbiofit или свойство ResponseMap fitproblem. Если существует отдельная ошибочная модель для каждого ответа, шум добавляется к каждому ответу отдельно с помощью соответствующей ошибочной модели.
Этот пример использует дрожжи гетеротримерные данные модели белка G и экспериментальные данные, о которых сообщают [1]. Для получения дополнительной информации о модели, смотрите раздел Background в Сканировании Параметра, Оценке Параметра и Анализе чувствительности в Дрожжах Гетеротримерный Цикл Белка G.
Загрузите модель белка G.
sbioloadproject gproteinВведите экспериментальные данные, содержащие курс времени для части активного белка G, как сообщается в ссылочной газете [1].
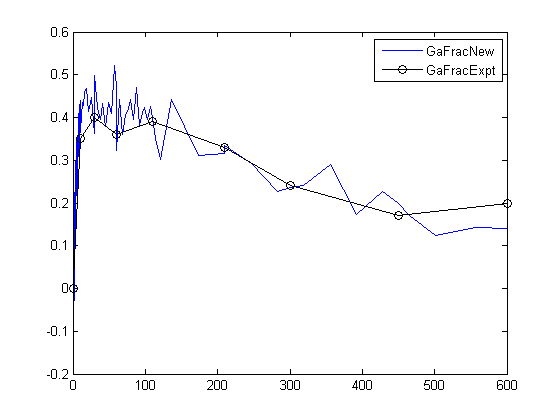
time = [0 10 30 60 110 210 300 450 600]'; GaFracExpt = [0 0.35 0.4 0.36 0.39 0.33 0.24 0.17 0.2]';
Создайте groupedData основанный на объектах на экспериментальных данных.
tbl = table(time,GaFracExpt); grpData = groupedData(tbl);
Сопоставьте соответствующий компонент модели с экспериментальными данными. Другими словами, укажите, которому разновидность в модели соответствует который переменная отклика в данных. В этом примере сопоставьте параметр модели GaFrac к переменной GaFracExpt экспериментальных данных от grpData.
responseMap = 'GaFrac = GaFracExpt'; Используйте estimatedInfo объект задать параметр модели kGd в качестве параметра быть оцененным.
estimatedParam = estimatedInfo('kGd');Выполните оценку параметра. Используйте аргумент пары "имя-значение" 'ErrorModel' задавать ошибочную модель, которая добавляет ошибку в данные моделирования.
fitResult = sbiofit(m1,grpData,responseMap,estimatedParam,'ErrorModel','proportional');
Просмотрите предполагаемое значение параметров kGd.
fitResult.ParameterEstimates
ans =
Name Estimate StandardError
_____ ________ _____________
'kGd' 0.11 0.00064116 Используйте random метод, чтобы получить данные моделирования с добавленным шумом с помощью пропорциональной ошибочной модели, которая была задана sbiofit. Обратите внимание на то, что шум добавляется только к состоянию ответа, которое является GaFrac параметр.
[ynew,paramEstim] = random(fitResult);
Выберите данные моделирования для GaFrac параметр.
GaFracNew = select(ynew,{'Name','GaFrac'});Постройте результаты симуляции.
plot(GaFracNew.Time,GaFracNew.Data)
hold on
Отобразите экспериментальные данные на графике, чтобы сравнить его с симулированными данными.
plot(time,GaFracExpt,'Color','k','Marker','o') legend('GaFracNew','GaFracExpt')
resultsObj — Результаты оценкиOptimResults возразите | NLINResults объектОценка заканчивается в виде OptimResults object или NLINResults object, который содержит результаты оценки, возвращенные sbiofit. Это должен быть скалярный объект.
data — Сгруппированные данные или выходные временаgroupedData возразите | вектор | массив ячеек векторовСгруппированные данные или выходные времена в виде a groupedData
object, вектор или массив ячеек векторов выходных времен.
Если это - вектор из моментов времени, random симулирует модель с новыми моментами времени с помощью оценок параметра от объекта resultsObj результатов.
Если это - массив ячеек векторов из моментов времени, random симулирует времена модели n с помощью выходных времен от каждого временного вектора, где n является длиной data.
Если это - groupedData объект, это должно иметь независимую переменную, такую как Время. Это должно также иметь переменную группы, если обучающие данные, используемые для подбора кривой, имеют такую переменную. Можно использовать groupedData возразите, чтобы запросить различные комбинации категорий если resultsObj содержит оценки параметра для каждой категории. random симулирует модель для каждой группы с заданными категориями. Например, предположите, что у вас есть набор оценок параметра для сексуальной категории (штекеры по сравнению с розетками) и категории возраста (молодой по сравнению со старым) в ваших обучающих данных. Можно использовать random симулировать ответы старого штекера (или любая другая комбинация) несмотря на то, что такой пациент не может существовать в обучающих данных.
Если resultsObj от оценки специфичных для категории параметров, data должен быть groupedData объект.
Примечание
Если UnitConversion включен для базовой модели SimBiology, которая использовалась для подбора кривой и data groupedData объект, data должен задать модули действительной переменной через data.Properties.VariableUnits свойство. Если это - числовой векторный массив или массив ячеек векторов из моментов времени, random использует TimeUnits модели.
dosing — Информация о дозах[] | {} | 2D матрица дозы возражает | вектор ячейки из объектов дозыИнформация о дозах в виде пустого [] или {}, 2D матрица или вектор ячейки из объектов дозы SimBiology (ScheduleDose object или RepeatDose object).
Если dosing пусто, никакие дозы не применяются в процессе моделирования, даже если модель имеет активные дозы.
Для матрицы объектов дозы это должно иметь одну строку или одну строку на группу во входных данных. Если это имеет одну строку, те же дозы применяются ко всем группам в процессе моделирования. Если это имеет несколько строк, каждая строка применяется к отдельной группе в том же порядке, как группы появляются во входных данных. Несколько столбцов позволены так, чтобы можно было применить несколько объектов дозы к каждой группе.
Для вектора ячейки из доз это должно иметь один элемент или один элемент на группу во входных данных. Каждым элементом должен быть [] или вектор из доз. Каждый элемент ячейки применяется к отдельной группе в том же порядке, как группы появляются во входных данных.
В дополнение к ручному построению использования объектов дозы sbiodose, если вход groupedData объект имеет информацию о дозах, можно использовать createDoses метод, чтобы создать дозы.
Объекты дозы dosing введите должно быть сопоставимо с исходными данными о дозировании, используемыми с sbiofit. Объекты должны иметь те же значения для свойств дозы (таких как TargetName) или должен быть параметрирован таким же образом как исходные данные о дозировании. Например, предположите, что исходная матрица дозирования имеет два столбца доз, где дозы в первом столбце предназначаются для разновидностей x и тех во вторых целевых разновидностях y столбца. Затем dosing должен иметь дозы в первом столбце, предназначающемся для разновидностей x и тех во втором столбце, предназначающемся для разновидностей y. Параметрированный пример дозы следующие. Предположим что Amount свойство дозы используется в оригинале sbiofit вызов параметрируется к ограниченному по объему моделью параметру 'A'. Все дозы для соответствующей группы (столбец) в dosing матричный вход должен иметь Amount свойство параметрируется к 'A'.
Количество строк в dosing элементы матрицы или число элементов в dosing вектор ячейки и количество групп или выходных временных векторов в data определите общее количество результатов симуляции в выходе ynew. Для получения дополнительной информации см. таблицу в ynew описание аргумента.
Примечание
Если UnitConversion включен для базового SimBiology® модель, которая использовалась для подбора кривой, dosing должен задать допустимую сумму и единицы измерения времени.
v — Варианты, чтобы применяться[] | {} | 2D матрица вариантов | вектор ячейки из вариантовВарианты, чтобы применяться в виде пустого массива ([], {}), 2D матрица или вектор ячейки из различных объектов.
Если вы не задаете этот аргумент, функция имеет следующее поведение в зависимости от ли второй входной параметр (data) задан также или нет.
Если data не задан, функция применяет специфичные для группы варианты от исходного вызова до sbiofit.
Если data векторный массив или массив ячеек выходных времен, функция не применяет специфичные для группы варианты.
Если data groupedData объект, функция применяет варианты только к группам, идентификатор группы которых совпадает с идентификатором группы в исходных обучающих данных, который использовался в вызове sbiofit.
Примечание
Базовые варианты, которые были заданы вариантами позиционный входной параметр в исходном вызове sbiofit всегда применяются к модели, и они применяются перед любыми специфичными для группы вариантами.
Если нет никаких базовых вариантов, то есть, вы не задавали variants введите при вызове sbiofit, random функция все еще применяет активные варианты модели, если существует кто-либо.
Если значением аргумента является [] или {}, функция не применяет специфичных для группы вариантов.
Если это - матрица вариантов, это должно иметь или одну строку или одну строку на группу. Каждая строка применяется к отдельной группе в том же порядке, как группы появляются в data или dosing. Если это имеет одну строку, те же варианты применяются ко всем группам в процессе моделирования. Если существует несколько столбцов, варианты применяются в порядке от первого столбца до последнего.
Если это - вектор ячейки из различных объектов, количество ячеек должно быть один или должно совпадать с количеством групп во входных данных. Каждым элементом должен быть [] или вектор из вариантов. Если существует отдельная ячейка, содержащая вектор из вариантов, они применяются ко всем симуляциям. Если существует несколько ячеек, варианты в i th ячейка применяются к симуляции i th группа.
Функция задает количество групп путем исследования data, и dosing входные параметры.
data может иметь 1 или группы N.
Если data и dosing аргументы не заданы, затем данные по умолчанию и дозирование определяются можно следующим образом:
Для необъединенных подгонок они - данные и дозирующий для одной группы, сопоставленной с этим результаты подгонки.
Для всех других подгонок они - целый набор данных и дозирования связанного с вызовом sbiofit.
ynew — Результаты симуляции с шумомSimData объектыРезультаты симуляции, возвращенные как вектор из SimData объекты. О состояниях сообщают в ynew состояния, которые были включены в responseMap входной параметр sbiofit а также любые другие состояния, перечисленные в StatesToLog свойство опций во время выполнения (RuntimeOptions) из модели SimBiology.
Общее количество результатов симуляции в ynew зависит от количества групп или выходных временных векторов в data и количество строк в dosing матрица.
Количество групп или выходных временных векторов в data | Количество строк в dosing матрица | Результаты симуляции |
|---|---|---|
1 | 0 | Общее количество Никакие дозы не применяются в процессе моделирования. |
1 | 1 | Общее количество Данная строка доз применяется во время симуляции. |
1 | N | Общее количество Каждая строка |
N | 0 | Общее количество Никакие дозы не применяются в процессе моделирования. |
N | 1 | Общее количество Та же строка доз применяется к каждой симуляции. |
| N | N | Общее количество Каждая строка |
| M | N | Функция выдает ошибку когда M ≠ N. |
parameterEstimates — Предполагаемые значения параметровПредполагаемые значения параметров, возвращенные как таблица. Это идентично resultsObj.ParameterEstimates свойство.
[1] И, T-M., Kitano, H. и Саймон, M. (2003). Количественная характеристика дрожжей гетеротримерный цикл белка G. PNAS. 100, 10764–10769.
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.