Объект, содержащий результаты вычисления элементарных эффектов для глобального анализа чувствительности (GSA)
SimBiology.gsa.ElementaryEffects объект содержит результаты GSA, возвращенные sbioelementaryeffects. Объект содержит вычисленные элементарные эффекты относительно входных параметров параметра.
Создайте SimBiology.gsa.ElementaryEffects объектное использование sbioelementaryeffects.
AbsoluteEffects — Отметьте, чтобы использовать абсолютные значения элементарных эффектовtrue (значение по умолчанию) | falseОтметьте, чтобы использовать абсолютные значения элементарных эффектов в виде true или false. По умолчанию функция использует абсолютные значения элементарных эффектов. Используя неабсолютные значения может составить в среднем при вычислении среднего значения. Для получения дополнительной информации смотрите Элементарные Эффекты для Глобального Анализа чувствительности.
Типы данных: логический
Time — Моменты времениЭто свойство доступно только для чтения.
Моменты времени, в которых элементарные эффекты вычисляются в виде столбца числовой вектор. Свойством является [] если все observables являются скалярами.
Типы данных: double
Results — GSA заканчивается с элементарными эффектамиЭто свойство доступно только для чтения.
GSA заканчивается с элементарными эффектами в виде массива структур. Размером массива является params-by-observables, где params является количеством входных параметров (входные параметры чувствительности), и observables является количеством observables (чувствительность выходные параметры).
Каждая структура содержит следующие поля.
Parameter — Имя входного параметра в виде вектора символов
Observable — Имя заметного в виде вектора символов
Mean — Среднее значение абсолютных значений элементарных эффектов в виде скалярного или числового вектора
StandardDeviation — Стандартное отклонение абсолютных элементарных эффектов в виде скалярного или числового вектора
Если все observables являются скаляром, то Mean и StandardDeviation поля заданы как скаляры. Если какие-либо observables являются векторами, Mean и StandardDeviation числовые векторы из длины Time. Если некоторые observables являются скалярами, и некоторые - векторы, скаляр observables расширены до скаляра, где каждый момент времени имеет то же значение.
Типы данных: struct
GridLevel — Уровень дискретизации области параметраЭто свойство доступно только для чтения.
Уровень дискретизации области параметра в виде положительного даже целое число. Этот параметр задает сетку равноотстоящих точек в области параметра, где каждая размерность дискретизируется с помощью Gridlevel+1
Для получения дополнительной информации смотрите Элементарные Эффекты для Глобального Анализа чувствительности.
Типы данных: double
GridDelta — Размер шага для вычисления элементарных эффектовGridLevel/2GridLevelЭто свойство доступно только для чтения.
Размер шага для вычисления элементарных эффектов в виде положительного целого числа между 1 и GridLevel. Размер шага измеряется в терминах узлов решетки между соседними точками. Следующий рисунок показывает примеры различных значений дельты сетки.
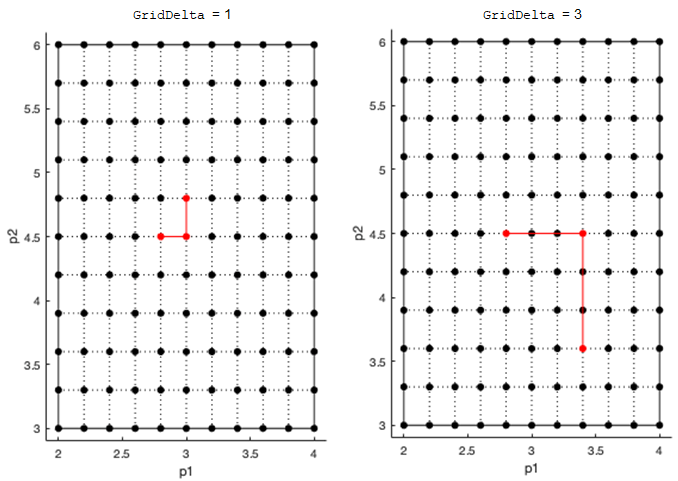
Для получения дополнительной информации смотрите Элементарные Эффекты для Глобального Анализа чувствительности.
Типы данных: double
PointSelection — Метод, чтобы выбрать точки выборки, чтобы вычислить элементарные эффекты"chain" (значение по умолчанию) | "radial"Это свойство доступно только для чтения.
Метод, чтобы выбрать точки выборки, чтобы вычислить элементарные эффекты в виде "chain" или "radial". "chain" укажите, что выбор использует метод Морриса [1]. "radial" укажите, что выбор использует метод Sohier [2]. Для получения дополнительной информации смотрите Элементарные Эффекты для Глобального Анализа чувствительности.
Типы данных: double
ParameterSamples — Выбранные значения параметровЭто свойство доступно только для чтения.
Выбранные значения параметров в виде таблицы. Таблица содержит (1+numel(params))*NumberSamples строки и numel(params) столбцы.
sbioelementaryeffects блоки использования k+1 строки, где k является количеством входных параметрических усилителей, чтобы вычислить элементарный эффект для каждого входного параметра. Общее количество этих блоков равно общему количеству выборок. Можно получить индексы блока путем выполнения kron((1:NumberSamples)',ones(numel(params)+1,1)). Для получения дополнительной информации смотрите Элементарные Эффекты для Глобального Анализа чувствительности.
Типы данных: table
Observables — Имена ответов модели или observablesЭто свойство доступно только для чтения.
Имена ответов модели или observables в виде массива ячеек из символьных векторов.
Типы данных: char
SimulationInfo — Информация о симуляции используемые в вычислениях элементарные эффектыЭто свойство доступно только для чтения.
Информация о симуляции, такая как данные моделирования и выборки параметра, используемые в вычислениях элементарные эффекты в виде структуры. Структура содержит следующие поля.
SimFunction — SimFunction объект используется для ответов имитационной модели или observables.
SimData — SimData массив размера [NumberSamples,1], где NumberSamples является количеством отсчетов. Массив содержит результаты симуляции от ParameterSamples.
OutputTimes — Числовой вектор-столбец, содержащий общий временной вектор всего SimData объекты.
Bounds — Числовая матрица размера [params,2]. params является количеством входных параметров. Первый столбец содержит нижние границы, и второй столбец содержит верхние границы для входных параметров чувствительности.
DoseTables — Массив ячеек таблиц дозы используется для SimFunction оценка. DoseTables выход getTable(doseInput), где doseInput является значением, заданным для 'Doses' аргумент пары "имя-значение" в вызове sbiosobol, sbiompgsa, или sbioelementaryeffects. Если никакие дозы не применяются, это поле установлено в [].
ValidSample — Логический вектор из размера [NumberSamples,1] указание, перестала ли симуляция для конкретной выборки работать. Передискретизация данных моделирования (SimData) может привести к NaN значения, если данные экстраполируются. Такой SimData обозначаются как недопустимые.
InterpolationMethod — Имя метода интерполяции для SimData.
SamplingMethod — Имя метода выборки раньше чертило ParameterSamples.
RandomState — Структура, содержащая состояние rng прежде, чем чертить ParameterSamples.
Типы данных: struct
resample | Передискретизируйте индексы Sobol или элементарные эффекты к новому временному вектору |
addobservable | Вычислите индексы Sobol или элементарные эффекты для нового заметного выражения |
removeobservable | Удалите индексы Sobol или элементарные эффекты observables |
addsamples | Добавьте дополнительные выборки, чтобы увеличить точность индексов Sobol или элементарного анализа эффектов |
plotData | Постройте сводные данные квантиля симуляций модели от глобального анализа чувствительности (требует Statistics and Machine Learning Toolbox), |
plot | Постройте средние значения и стандартные отклонения элементарных эффектов |
bar | Постройте величины средних значений и стандартные отклонения элементарных эффектов |
plotGrid | Постройте сетку параметра, и точки использовались для расчета элементарных эффектов |
Загрузите модель роста опухоли.
sbioloadproject tumor_growth_vpop_sa.sbprojЗаставьте вариант предполагаемыми параметрами и дозой применяться к модели.
v = getvariant(m1);
d = getdose(m1,'interval_dose');Получите активный configset и установите вес опухоли как ответ.
cs = getconfigset(m1);
cs.RuntimeOptions.StatesToLog = 'tumor_weight';Симулируйте модель и постройте профиль роста опухоли.
sbioplot(sbiosimulate(m1,cs,v,d));

Выполните глобальный анализ чувствительности (GSA) на модели, чтобы найти параметры модели, к которым рост опухоли чувствителен.
Во-первых, задайте параметры модели интереса, которые вовлечены в фармакодинамику роста опухоли. Задайте ответ модели как вес опухоли.
modelParamNames = {'L0','L1','w0','k1'};
outputName = 'tumor_weight';Затем выполните GSA путем вычисления элементарных эффектов с помощью sbioelementaryeffects. Используйте 100 как количество отсчетов и набор ShowWaitBar к true показать прогресс симуляции.
rng('default');
eeResults = sbioelementaryeffects(m1,modelParamNames,outputName,Variants=v,Doses=d,NumberSamples=100,ShowWaitbar=true);Покажите средний ответ модели, результаты симуляции и теневую область, покрывающую 90% результатов симуляции.
plotData(eeResults,ShowMedian=true,ShowMean=false);

Можно настроить область квантиля к различному проценту путем определения Alphas для более низких и верхних квантилей всех ответов модели. Например, alpha значение 0,1 графиков теневая область между 100*alpha и 100*(1-alpha) квантили всех симулированных ответов модели.
plotData(eeResults,Alphas=0.1,ShowMedian=true,ShowMean=false);

Постройте курс времени средних значений и стандартные отклонения элементарных эффектов.
h = plot(eeResults);
% Resize the figure.
h.Position(:) = [100 100 1280 800];![Figure contains 8 axes objects. Axes object 1 with title sensitivity output [Tumor Growth Model].tumor_weight contains an object of type line. Axes object 2 with title sensitivity output [Tumor Growth Model].tumor_weight contains an object of type line. Axes object 3 contains an object of type line. Axes object 4 contains an object of type line. Axes object 5 contains an object of type line. Axes object 6 contains an object of type line. Axes object 7 contains an object of type line. Axes object 8 contains an object of type line.](../../examples/simbio/win64/PerformGSAByComputingElementaryEffectsExample_04.png)
Среднее значение эффектов объясняет, оказывают ли изменения входных значений параметров какое-либо влияние на ответ веса опухоли. Стандартное отклонение эффектов объясняет, зависит ли изменение чувствительности от местоположения в области параметра.
От среднего значения графиков эффектов, параметры L1 и w0 кажется, самые чувствительные параметры к весу опухоли, прежде чем доза будет применена в t = 7. Но, после того, как доза применяется, k1 и L0 станьте более чувствительными параметрами и способствуйте больше всего дозируемому после того, как этапу веса опухоли. Графики стандартного отклонения эффектов показывают больше отклонений для больших значений параметров на более позднем этапе (t> 35), чем для этапа прежде-чем-дозы роста опухоли.
Можно также отобразить величины чувствительности в столбиковой диаграмме. Каждая цветная штриховка представляет гистограмму, представляющую значения в разное время. Более темные цвета означают, что те значения происходят чаще по курсу всего времени.
bar(eeResults);
![Figure contains an axes object. The axes object with title sensitivity output [Tumor Growth Model].tumor_weight contains 18 objects of type patch, line. These objects represent mean, standard deviation.](../../examples/simbio/win64/PerformGSAByComputingElementaryEffectsExample_05.png)
Можно также построить сетки параметра, и выборки использовались для расчета элементарных эффектов.
plotGrid(eeResults)

Можно задать больше выборок, чтобы увеличить точность элементарных эффектов, но симуляция может занять больше времени, чтобы закончиться. Используйте addsamples добавить больше выборок.
eeResults2 = addsamples(eeResults,200);
SimulationInfo свойство объекта результата содержит различную информацию для вычисления элементарных эффектов. Например, данные о симуляции модели (SimData) для каждой симуляции с помощью набора выборок параметра хранятся в SimData поле свойства. Это поле является массивом SimData объекты.
eeResults2.SimulationInfo.SimData
SimBiology SimData Array : 1500-by-1 Index: Name: ModelName: DataCount: 1 - Tumor Growth Model 1 2 - Tumor Growth Model 1 3 - Tumor Growth Model 1 ... 1500 - Tumor Growth Model 1
Можно узнать, перестала ли какая-либо симуляция модели работать во время расчета путем проверки ValidSample поле SimulationInfo. В этом примере поле не показывает не пройдено запусков симуляции.
all(eeResults2.SimulationInfo.ValidSample)
ans = logical
1
Можно добавить пользовательские выражения как observables и вычислить элементарные эффекты добавленного observables. Например, можно вычислить эффекты для максимального веса опухоли путем определения пользовательского выражения можно следующим образом.
% Suppress an information warning that is issued. warnSettings = warning('off', 'SimBiology:sbservices:SB_DIMANALYSISNOTDONE_MATLABFCN_UCON'); % Add the observable expression. eeObs = addobservable(eeResults2,'Maximum tumor_weight','max(tumor_weight)','Units','gram');
Постройте вычисленные результаты симуляции, показывающие 90%-ю область квантиля.
h2 = plotData(eeObs); h2.Position(:) = [100 100 1500 800];

Можно также удалить заметное путем определения его имени.
eeNoObs = removeobservable(eeObs,'Maximum tumor_weight');Восстановите настройки предупреждения.
warning(warnSettings);
[1] Моррис, Макс Д. “Планы Выборки факториала относительно Предварительных Вычислительных Экспериментов”. Технометрики 33, № 2 (май 1991): 161–74.
[2] Sohier, Анри, Жан-Лу Фарг и Хелене Пит-Ланир. “Улучшение Представительности Метода Морриса для Воздушного Запуска, чтобы вращаться вокруг Разделения”. Объемы Продолжений IFAC 47, № 3 (2014): 7954–59.
У вас есть модифицированная версия этого примера. Вы хотите открыть этот пример со своими редактированиями?
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.