В этом примере показано, как выполнить неразделенный на отсеки анализ, чтобы вычислить параметры NCA и оценить параметры модели [1] роста опухоли от экспериментальных данных с помощью нелинейной регрессии в приложении SimBiology Model Analyzer.
Моделью, используемой в этом примере, является SimBiology® реализация фармакокинетической/фармакодинамической модели (PK/PD) Simeoni и др. Это определяет количество эффекта лекарств от рака на кинетике роста опухоли от в естественных условиях исследований на животных. Фармакокинетика препарата описана моделью 2D отсека с дозированием болюсного внутривенного введения и линейным устранением (ke) из отсека Central. Рост опухоли является двухфазным процессом с начальным экспоненциальным ростом, сопровождаемым линейным ростом. Темп роста распространяющихся опухолевых клеток описан
L0, L1 и Ψ являются параметрами роста опухоли, x1 является весом распространяющихся опухолевых клеток, и w является общим весом опухоли. В отсутствие любых наркотиков опухоль состоит из распространяющихся ячеек только, то есть, w = x1. В присутствии противоракового агента часть распространяющихся ячеек преобразовывается в нераспространяющиеся ячейки. Уровень этого преобразования принят, чтобы быть функцией концентрации препарата в плазме и факторе эффективности k2. Нераспространяющиеся ячейки x2 проходят серию транзитных этапов (x3 и x4) и в конечном счете очищены от системы. Поток - через транзитных отсеков моделируется как процесс первого порядка с константой скорости k1.
Модель SimBiology вносит эти корректировки в фармакодинамику роста опухоли:
Вместо того, чтобы задать вес опухоли как сумму x1, x2, x3 и x4, модель задает вес опухоли реакцией под названием Increase, null → tumor_weight, со скоростью реакции .
tumor_weight является общим весом опухоли, x1 является весом распространяющихся опухолевых клеток и L0, и L1 является параметрами роста опухоли.
Точно так же модель задает уменьшение в весе опухоли реакцией под названием Decay, tumor_weight → null, со скоростью реакции k1*x4. Постоянный k1 является параметром форвардного курса, и x4 является последними разновидностями в ряду транзитных сокращений веса опухоли.
ke является функцией разрешения и объемом центрального отсека: ke = Cl_Central/Central.
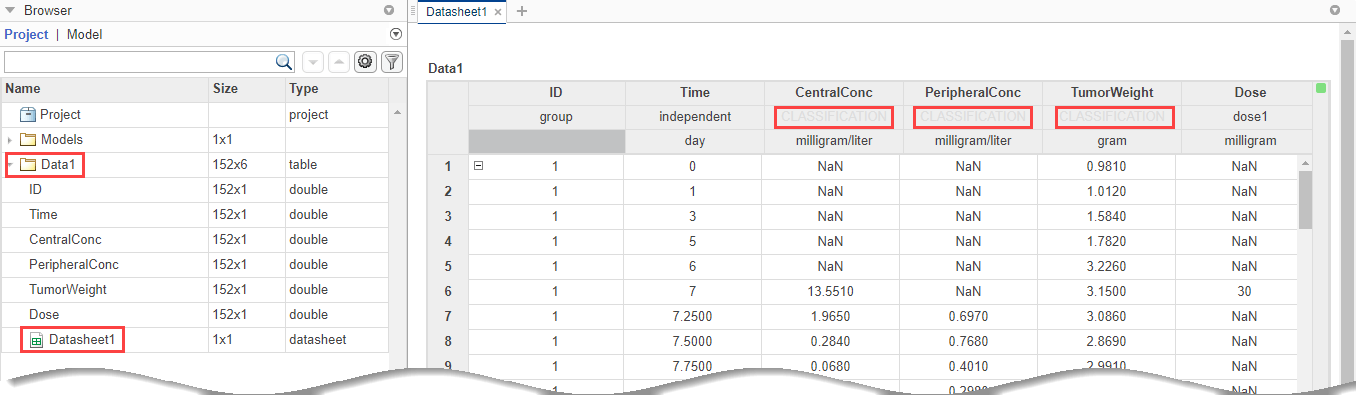
Экспериментальные (синтетические) данные содержат измерения от восьми пациентов для трех ответов: измеренные концентрации препарата в центральном отсеке, в периферийном отсеке и измеренном весе опухоли. Данные также содержат информацию о дозах, и каждый пациент получает дозу IV в день 7.
Набор данных содержит следующие столбцы.
ID — Идентификаторы пациентов
Время Времена, когда измерения проведены
CentralConc — Концентрация препарата в центральном отсеке
PeripheralConc — Концентрация препарата в периферийном отсеке
Dose — Информация о дозах для каждого пациента
NaN значения используются каждый раз, когда нет никакого измерения, или никакая доза не дана.
Откройте приложение SimBiology Model Analyzer путем ввода simBiologyModelAnalyzer в командной строке или путем нажатия на значок приложения на вкладке Apps.
На вкладке Home приложения выберите Open.
Перейдите к папке matlabroot\examples\simbio\data\. matlabroot является папкой, где вы установили MATLAB. Выберите файл с именем проекта tumor_growth_fitPKPD.sbproj. В панели Browser папка Models содержит Tumor Growth Model и папка Data1 содержит экспериментальные данные наряду с информацией о дозах.
Классифицируйте столбцы данных так, чтобы такие переменные классификации могли использоваться программой Fit позже в примере. Приложение выполняет автоматические классификации, столь же соответствующие (такие как ID, Time, столбцы Dose). Но для столбцов данных измеренного отклика, таких как CentralConc, необходимо вручную классифицировать их как зависимые переменные. Для этого откройте сначала таблицу данных можно следующим образом. В панели Browser расширьте папку Data1 и дважды кликните Datasheet1.
В таблице Data1 дважды кликните Classification под CentralConc. Выберите dependent. Повторите тот же процесс для PeripheralConc и TumorWeight. Теперь все столбцы данных имеют соответствующие классификации, и данные готовы к употреблению.
Примечание
Приложение автоматически классифицировало:
Столбец ID как group (сгруппированная переменная).
Столбец Time как independent (независимая переменная).
Столбец Dose как dose1 (переменная дозирования). Если существует больше чем один столбец дозы, они могут быть классифицированы как dose2, dose3, и так далее.
После того, как вы загрузите данные, можно визуализировать измеренные отклики.
В панели Браузера нажмите Data1.
На вкладке Home, в разделе Plot, кликают по графику time. Приложение генерирует график временной зависимости всех трех ответов, а именно: CentralConc, PeripheralConc и TumorWeight.
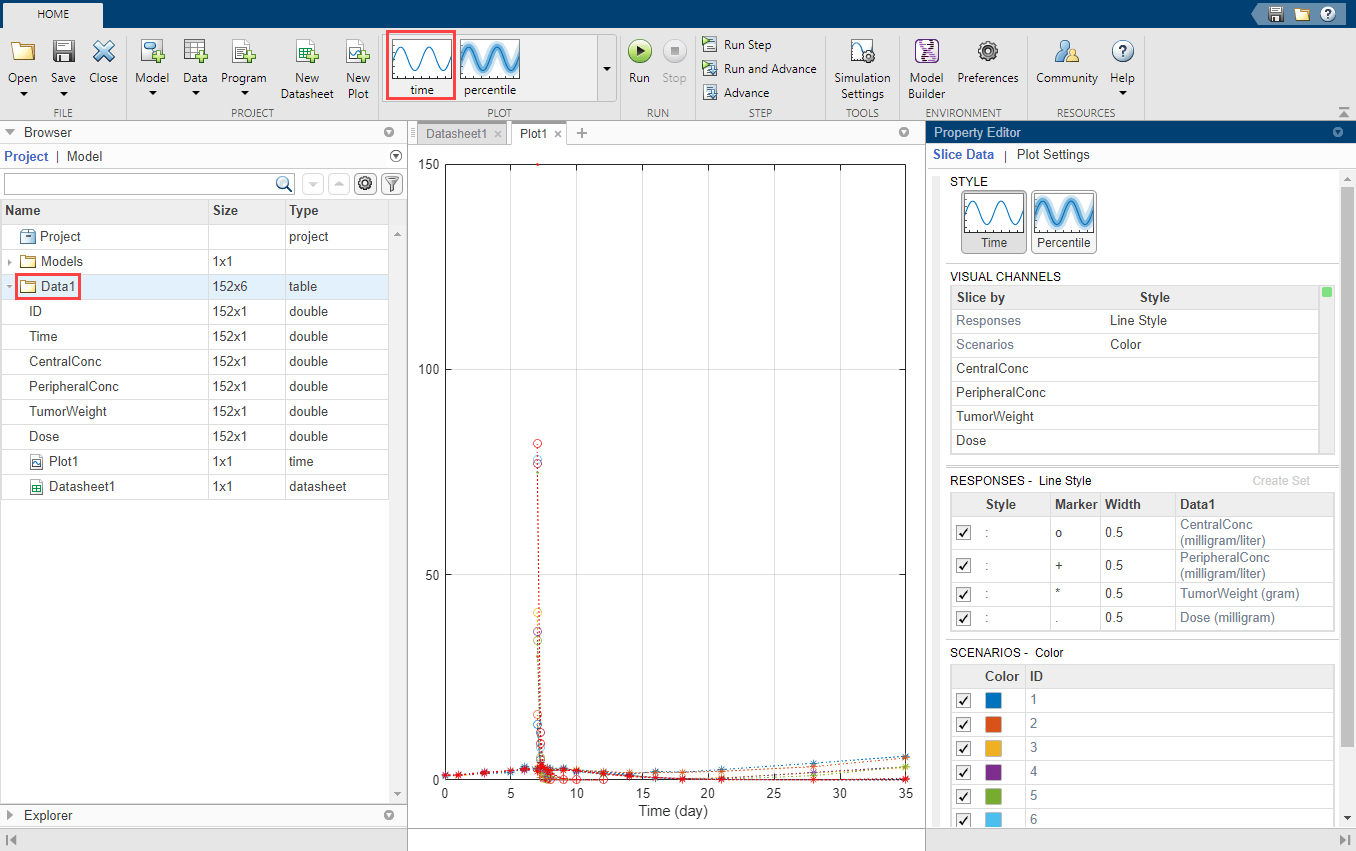
В графике временной зависимости по умолчанию Responses соответствует измеренным откликам и построен с помощью различных стилей линии. Scenarios относится к различным группам (восемь пациентов) в данных и построен с помощью различных цветов.
Совет
Графики поддерживаются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками состояния. Когда данные (или экспериментальные данные или результаты симуляции) удалены или изменены, графики также обновляются согласно изменениям в базовых данных.
Шаги в этом разделе являются дополнительными и не являются необходимыми для подбора кривой. Можно настроить график сделать его более ясным. Например, можно отобразить данные о PD на графике (TumorWeight) на различной оси, чем данные PK (CentralConc и PeripheralConc). Для этого создайте две различных группы (наборы) ответов, где первый набор содержит только TumorWeight, и второй набор содержит CentralConc и PeripheralConc.
Щелкните правой кнопкой по TumorWeight (gram) по таблице Responses и выберите Create New Set. Приложение создает Set 1 и Set 2. Set 1 содержит только TumorWeight, который теперь построен на различной оси, чем Set 2, который содержит CentralConc и PeripheralConc.
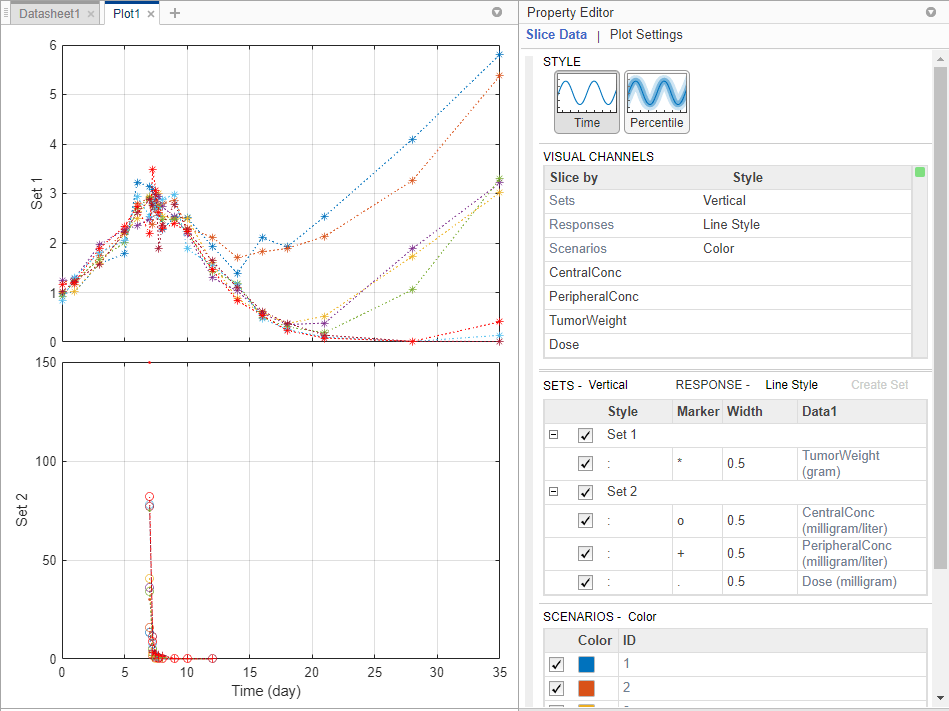
Таблица Visual Channels теперь содержит Sets. Эта таблица является сводной таблицей всех переменных разрезания, которые в настоящее время присутствуют в графике и их соответствующих стилях графика. В этом текущем графике режущими переменными является Sets, Responses и Scenarios.
Совет
Можно нарезать данные с помощью различных переменных разрезания. Каждая переменная разрезания появляется в графике с различным визуальным стилем (или канал), такой как цвет, стиль линии и положение осей. Разрезание переменных может представлять атрибуты данных, такие как ответы или сценарии (то есть, группы или запуски симуляции). Разрезание переменных может также быть ковариантами или значениями параметров, сопоставленными со сценарием или группой. По умолчанию приложение обеспечивает режущие переменные для различных переменных отклика и различных сценариев в отображенных на графике данных. Можно добавить другие визуальные стили (или каналы) для наборов ответов и сопоставленного параметра или ковариационных переменных.
Можно также сгруппировать ответы на основе различных суммарных доз, которые получают пациенты. Существует три различных группы дозы: 30, 75, и 150 мг.
В таблице Visual Channels, в строке Dose, дважды кликают пустую ячейку и выбирают Color. Красный индикатор появляется, потому что другая переменная разрезания (Scenarios) имеет тот же стиль графика. Очистите стиль (визуальный канал) для Scenarios путем выбора пустой.
В таблице Dose приложение имеет автоматически сгруппированный суммарные дозы. Установите Number of Bins на 3. Можно теперь видеть, что суммарная доза оказывает влияние на размер опухоли. Чем выше доза, тем меньшей опухоль становится.
Можно также запросить соответствующую группу дозы от каждой линии путем показа ее всплывающей подсказки. Нажмите Ctrl и кликните по синей линии, чтобы отобразить ее всплывающую подсказку. Чтобы удалить его, Ctrl + Щелкает еще раз где угодно по той же линии.
Используя препарат фармакокинетические данные, можно оценить параметры NCA. NCA является агностиком модели и может дать понимание фармакокинетики препарата без любых базовых предположений. Можно использовать некоторые результаты NCA как первоначальные оценки при калибровке модели к данным, как обсуждено позже в этом примере. Для получения дополнительной информации в списке доступных параметров NCA и их формул, смотрите Неразделенный на отсеки Анализ.
На вкладке Home выберите Program > Non-Compartmental Analysis. Появляется новая программа (Program1).
Шаг настройки Data программы задает набор данных, чтобы использовать для анализа NCA. В этом примере программа автоматически выбирает Data1.
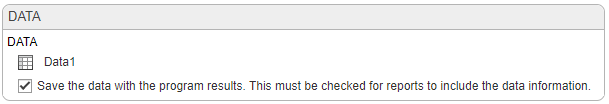
Шаг выполнения NCA задает ассоциации столбца данных и детали алгоритма. В таблице Definition, набор Concentration к CentralConc. Оставьте другие настройки без изменений.
На вкладке Home нажмите Run.
Если анализ NCA завершен, приложение открывает новую таблицу данных, содержащую результаты.
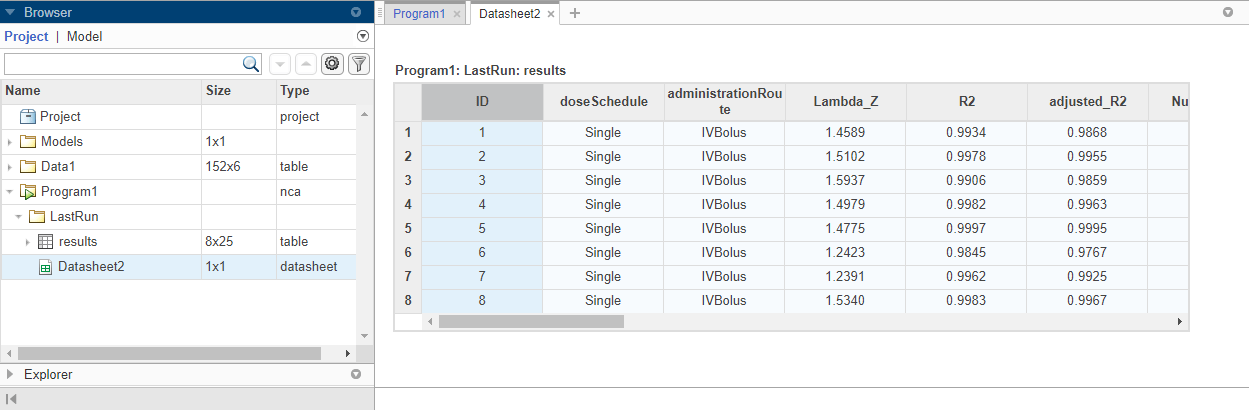
Программа также сохраняет результаты в папке LastRun программы по умолчанию. Чтобы получить доступ к результатам, в панели Browser, расширяют папку Program1. Затем расширьте папку LastRun. Результаты NCA хранятся в таблице под названием results. Для получения дополнительной информации о расчетных параметрах NCA, смотрите Неразделенный на отсеки Анализ.
Можно экспортировать результаты NCA в MATLAB® рабочая область и выполняет дальнейшие анализы данных в командной строке.
Щелкните правой кнопкой по results. Выберите Export Data to MATLAB Workspace.
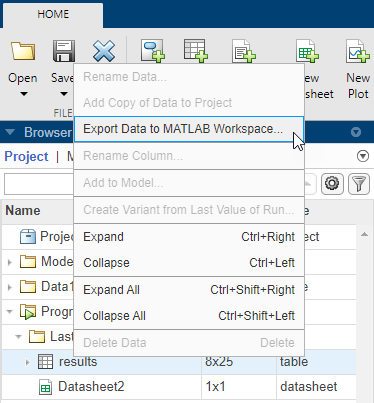
Диалоговое окно SimBiology Data Export открывается. Поменяйте имя переменной к ncatable. Нажмите OK.
После того, как вы экспортируете данные в рабочее пространство MATLAB, можно анализировать данные в командной строке. Например, можно вычислить среднее разрешение препарата из данных NCA и использовать его в качестве значения параметра модели.
SimBiology обеспечивает различные методы регрессии, чтобы оценить параметры модели на основе экспериментальных данных. Этот пример детализирует шаги для использования нелинейного метода регрессии lsqnonlin (требует Optimization Toolbox™) подбирать модель к данным. Если у вас нет Optimization Toolbox, использования приложения fminsearch вместо этого. В целях примера только некоторые параметры модели PK/PD оцениваются, а именно: k1, L0, L1, Cl_Central, k12 и k21.
От вкладки Home выберите Program > Fit Data. Новая программа (Program2) появляется на новой вкладке. Data и шаги Model были предварительно заполнены с Data1 и Tumor Growth Model, соответственно.
По умолчанию шаг Fit автоматически генерирует графики после того, как подбор кривой будет завершен. Отключите генерацию графика путем нажатия на значок графика наверху шага программы Fit на данный момент. Графики будут исследованы позже в примере.
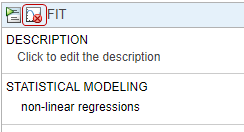
В таблице Data Map задайте отображение между компонентами модели и столбцами данных от входных данных.
Строка group идентифицирует, какой столбец в данных является сгруппированной переменной, такой как идентификаторы пациентов.
Строка independent идентифицирует, какой столбец в данных является независимой переменной, такой как время.
Строка response идентифицирует, которому столбец данных ответа или измерения соответствует который компонент модели. Если существуют данные о множественном ответе, можно добавить больше строк ответа путем нажатия кнопки Response в нижней части таблицы Data Map. Чтобы удалить ответ из таблицы, щелкните правой кнопкой и выберите Delete.
Строка dose from data задает, который сопоставляет столбец в данных, к которому компоненту модели как доза предназначаются. Если существует несколько столбцов дозы, можно добавить больше строк путем нажатия кнопки Dose.
Строка variant from data задает, какой столбец в данных содержит альтернативные значения параметров для который компонент модели. Нажмите кнопку Variant, чтобы добавить больше.
Примечание
В этом примере приложение использует информацию о классификации из таблицы данных входных данных и сопоставляет столбец ID как сгруппированную переменную (заданный строкой group в таблице) и столбец Time как независимая переменная (заданный строкой independent в таблице). Это также идентифицировало CentralConc, PeripheralConc и TumorWeight как столбцы ответа.
В первой строке ответа, рядом с CentralConc, дважды кликают ячейку Component и вводят Central.Drug как соответствующий компонент модели для того столбца данных измерения.
Точно так же сопоставьте столбец PeripheralConc с Peripheral.Drug.
Сопоставьте TumorWeight с [Tumor Growth Model].tumor_weight.
Сопоставьте столбец Dose с Central.Drug, чтобы указать, что разновидность Drug в отсеке Central дозируется.
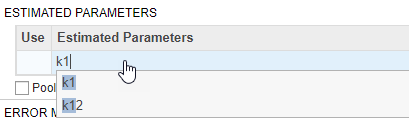
Задайте параметры модели, чтобы оценить в таблице Estimated Parameters. Дважды кликните пустую ячейку в столбце Estimated Parameters и введите k1. Приложение показывает компоненты модели с соответствием с именами. Выберите k1 из списка.
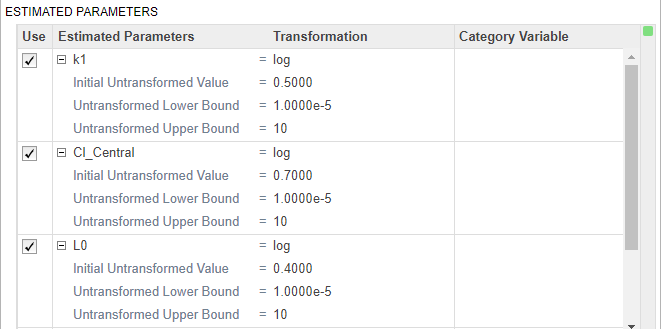
По умолчанию параметр преобразовывается в журнал, как обозначено преобразованием log. Можно изменить преобразование ни в какое преобразование none, probit, или logit преобразование. Для получения дополнительной информации смотрите Преобразования Параметра. В данном примере сохраните журнал по умолчанию, преобразовывают, потому что он часто улучшает сходимость. Initial Untransformed Value автоматически установлен в значение модели, которое является 0.5.
Осуществите биологические параметры, чтобы остаться положительными путем определения Untransformed Lower Bound и Untransformed Upper Bound как 1e-5 и 10, соответственно.
Точно так же добавьте следующие параметры: Cl_Central, L0, L1, k12 и k21.
Выберите Pooled fit, чтобы оценить один набор параметров для всех пациентов (подгонка населения). Если вы не выбираете Pooled fit, приложение оценивает один набор параметров для каждого пациента (отдельная подгонка).
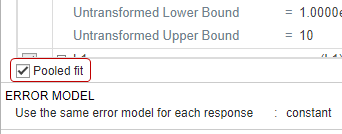
Ошибочная модель по умолчанию является постоянной ошибочной моделью. Поддержки SimBiology постоянные, пропорциональные, экспоненциальные, и объединенные ошибочные модели. Для получения дополнительной информации см. Ошибочные Модели. На данный момент используйте постоянную ошибочную модель.
Сохраните остальную часть подходящих настроек неизменной. Эти настройки
Метод оценки — метод по умолчанию lsqnonlin если у вас есть Optimization Toolbox. Если вы не делаете, использование приложения fminsearch.
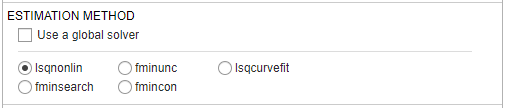
Для получения дополнительной информации см. Поддерживаемые Методы для Оценки Параметра SimBiology.
Настройки алгоритма — Наиболее распространенные опции для метода оценки. Щелкните, чтобы расширить раздел и видеть опции. Чтобы видеть описание каждой опции, кликните по информационному значку справа от заголовка.
Усовершенствованные Настройки Алгоритма — Расширенные настройки для метода оценки. Таблица пуста по умолчанию.
После того, как вы установите подходящие опции, можно запустить шаг Fit.
Наверху шага Fit нажмите кнопку Run this program step.
По умолчанию шаг Fit показывает прогресс оценки параметра отдельную фигуру. График прогресса показывает живое состояние оценки параметра и подходящих качественных мер, таких как логарифмическая вероятность. Для получения дополнительной информации см. График Прогресса.
График прогресса показывает, что подгонка сходилась. Можно закрыть график прогресса.
Если вы используете fminsearch, подгонка может не сходиться из-за достижения максимального количества итераций. Можно увеличить MaxIter в Algorithm Settings, но в целях этого примера, можно продолжить завершать шаги, не делая так.
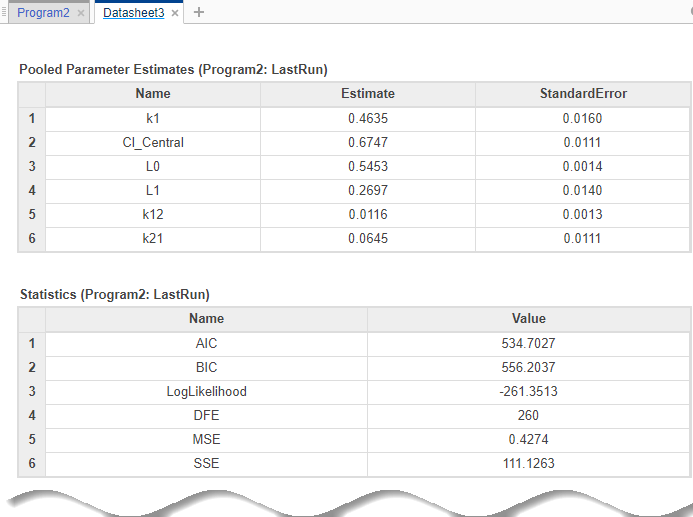
Если оценка параметра завершена, результаты подгонки показывают в новой таблице данных. Таблица данных содержит оценки параметра и другую информацию, связанную с подходящими качественными мерами, такими как AIC и BIC, который может быть полезным, чтобы сравнить эффективность различных ошибочных моделей.
В дополнение к качественной статистике можно также просмотреть различные подходящие графики, такой как фактические по сравнению с предсказанными графиками и остаточными графиками распределения.
В панели Browser расширьте Program2> LastRun, который содержит results и simdataI. results содержит оцененные значения параметров и подходящую статистику. simdataI содержит симулированные ответы модели для каждого индивидуума (пациент или группа) использование предполагаемых значений параметров.
Нажмите results. Доступные подходящие графики автоматически перечислены в разделе Plot по вкладке Home. Затем выберите Act vs Pred из списка.
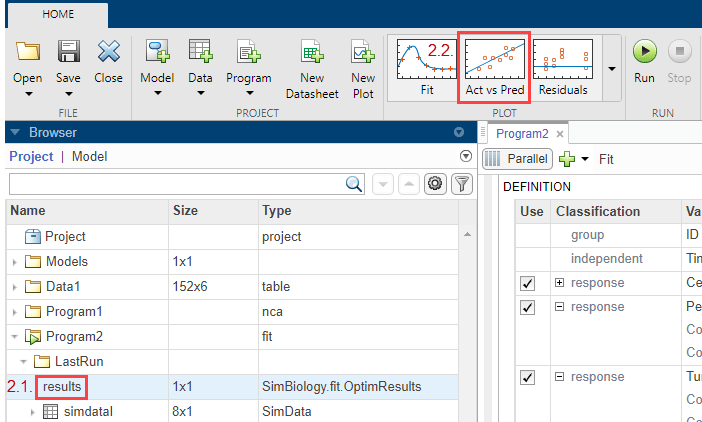
Фактическое по сравнению с предсказанным графиком появляется на отдельной вкладке. Предсказанные ответы построены на x - ось и наблюдаемые (экспериментальные) ответы построены на y - ось.
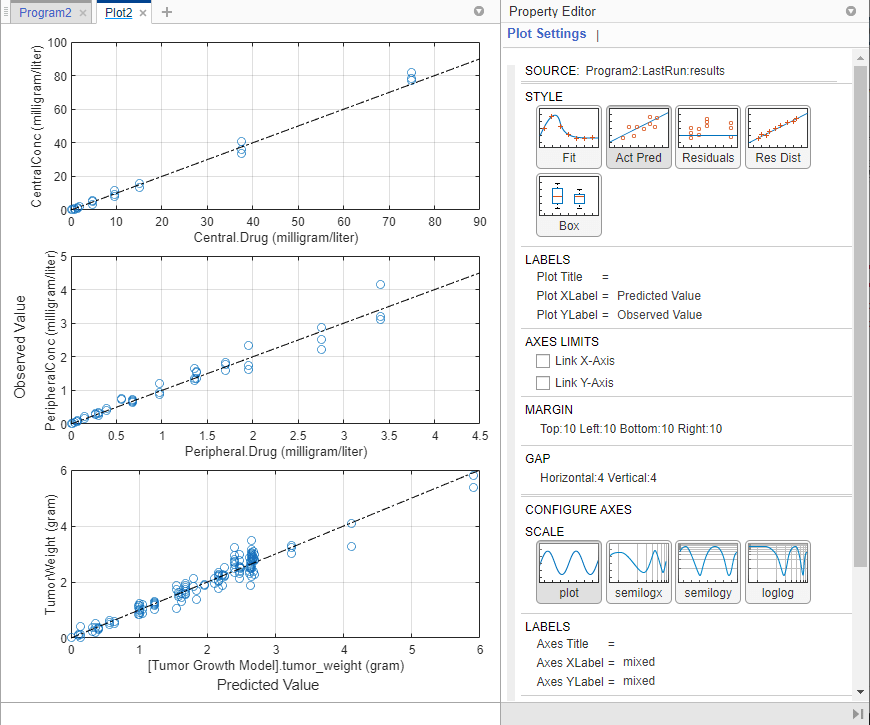
Можно изменить график в другие поддерживаемые графики путем выбора одного из графиков от раздела Style в Property Editor. Если вы хотите новый график на его собственной отдельной вкладке, и вы не хотите снова использовать существующую вкладку графика, выбирать график из раздела Plot по вкладке Home.
Измените график в остаточный график распределения путем выбора Res Dist в разделе Style.
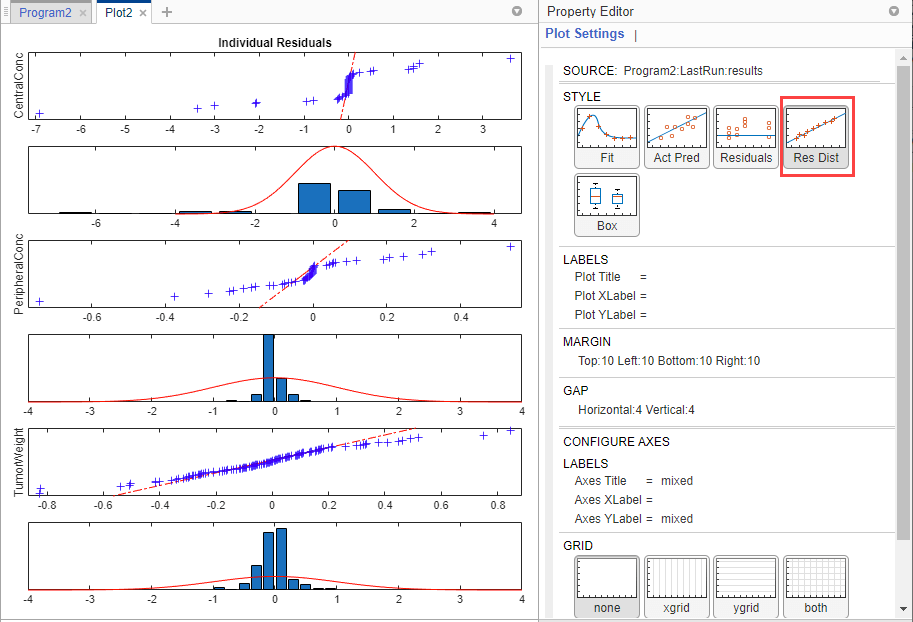
График показывает, нормально распределены ли остаточные значения для каждого ответа. В идеальном графике нормального распределения остаточных значений остаточные значения выстраиваются в линию вдоль диагональной линии через график, и гистограммы указывают на нормальную подгонку. Однако из графика, остаточные значения для всех трех ответов, особенно CentralConc и PeripheralConc, кажется, не нормально распределены. Это могла быть индикация, что постоянное ошибочное предположение модели является неправильным.
Следующие шаги показывают, как изменить ошибочную модель в экспоненциальную ошибочную модель, чтобы соответствовать данным снова и сравнить подходящую статистику двух различных ошибочных моделей.
Сохраните Результаты подгонки. Перед подгонкой данных снова с помощью экспоненциальной ошибочной модели, сохраните постоянный ошибочный результат модели в отдельной папке. В противном случае, программа, по умолчанию, перезаписи следуют из папки LastRun каждый раз, когда вы запускаете подгонку.
Щелкните правой кнопкой по папке LastRun подходящей программы в панели Browser.
Выберите Save Data.
В диалоговом окне Save Data введите fit_constant как имя данных.
Повторно выполните Подгонку С Экспоненциальной Ошибочной Моделью. После того, как вы сохраните данные, можно повторно выполнить подходящую программу с различной ошибочной моделью.
Возвратитесь к подходящей программе путем нажатия на вкладку Program2. В разделе Error Model выберите exponential.
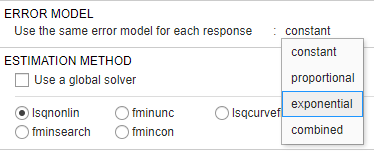
Наверху шага Fit нажмите кнопку Run this program step.

Закройте график прогресса после того, как подгонка завершится.
Если вы закрыли предыдущую таблицу данных (Datasheet3), который содержит подходящую статистику от предыдущей подгонки, вновь откройте таблицу данных. Для этого в панели Browser, расширьте Program2> fit_constant. Затем дважды кликните Datasheet3.
От папки LastRun перетащите results на Datasheet3. Новые столбцы (Program2_LastRun), содержащий последние результаты подгонки, добавляются рядом с предыдущими результатами подгонки (Program2_fit_constant).
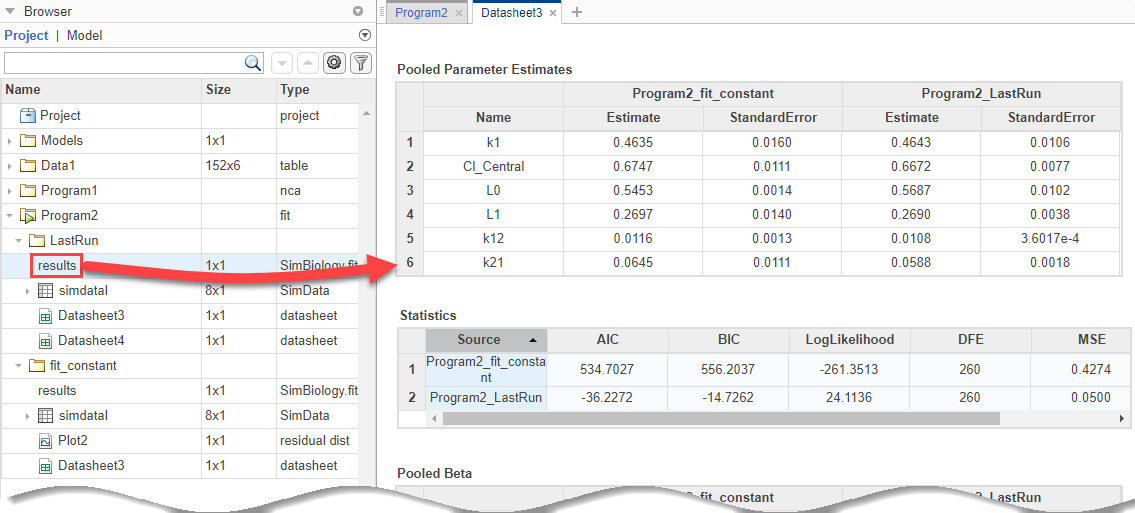
Таблица Statistics сравнивает подходящие качественные меры. От сравнения и AIC и BIC подгонки с помощью экспоненциальной ошибочной модели меньше, чем те из предыдущей подгонки. Это указывает, что экспоненциальная ошибочная модель соответствует данным лучше, чем постоянная ошибочная модель. Большая логарифмическая вероятность экспоненциальной ошибочной модели также указывает, что это - лучшая подгонка.
Затем посмотрите на остаточный график распределения. Нажмите results от папки LastRun. Затем нажмите Residual Dist от раздела Plot по вкладке Home.
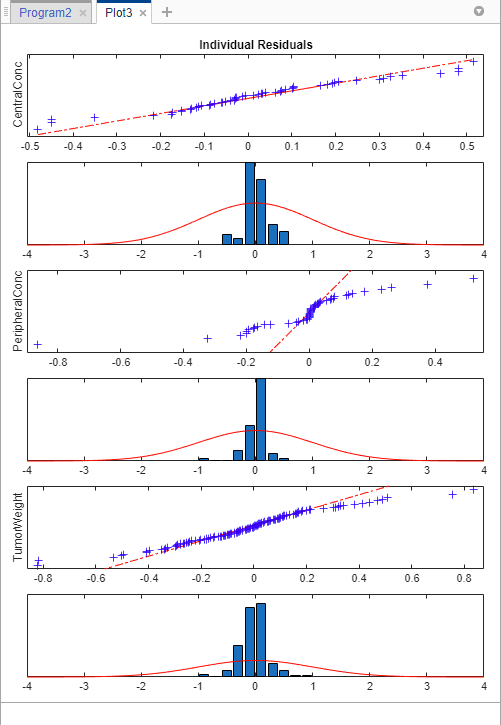
По сравнению с остаточными распределениями постоянной ошибочной модели остаточные распределения из экспоненциальной ошибочной модели выглядят более нормальными, указывая, что экспоненциальная ошибочная модель соответствует данным лучше.
Другой способ оценить качество результатов подгонки состоит в том, чтобы вычислить 95% доверительных интервалов для предполагаемых параметров и предсказаний модели — то есть, результаты симуляции модели с помощью предполагаемых параметров. Этот шаг требует Statistics and Machine Learning Toolbox™.
Возвратитесь к подходящей программе. Кликните (+) значок в верхнем левом углу и выберите Confidence Interval. Шаг Confidence Interval кажется выполняющим шаг Fit.
Наверху шага Confidence Interval отключите автоматическую генерацию графиков путем нажатия на значок графика. И для Parameter Confidence Intervals и для Prediction Confidence Intervals, используйте метод по умолчанию gaussian и 95% доверительный уровень. Нажмите кнопку Run this program step, чтобы вычислить доверительные интервалы.

Для доверительных интервалов параметра поддерживаемые методы являются гауссовыми, profileLikelihood, и начальная загрузка.
Для доверительных интервалов предсказания поддерживаемые методы являются гауссовыми и загружаются.
После того, как завершенный, результаты хранятся как parameterCI и predictionCI в папке LastRun программы. parameterCI содержит 95% доверительных интервалов для предполагаемых параметров. predictionCI содержит 95% доверительных интервалов для предсказаний модели.
Постройте 95% доверительных интервалов для предполагаемых параметров. Нажмите parameterCI в Browser разделяют на области и выбирают Confidence в разделе Plot.
Доверительный интервал для каждого предполагаемого параметра показывают в новом графике. График показывает успешный расчет доверительных интервалов для всех предполагаемых параметров.
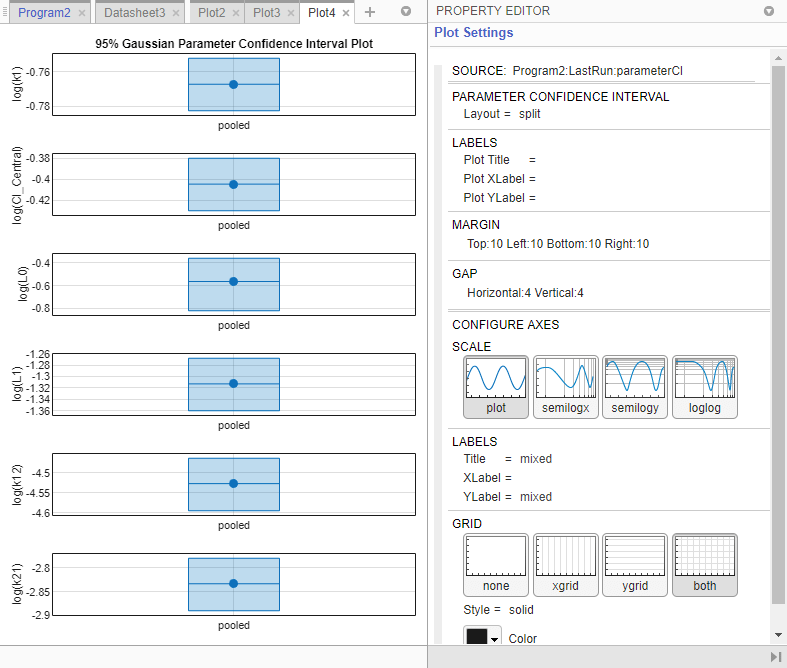
В зависимости от результата (состояние) оценки доверительного интервала приложение строит результаты по-другому.
Если состояние оценки доверительного интервала имеет успех (как в вышеупомянутом графике), приложение использует первый цвет по умолчанию (синий), чтобы построить график и точку в центре для каждой оценки параметра. Приложение также строит поле, чтобы указать на доверительные интервалы.
Если состояние ограничивается или допускающее оценку, приложение использует второй (красный) цвет по умолчанию и строит график, сосредоточенную точку и поле, чтобы указать на доверительные интервалы.
Если состояние не является допускающим оценку, приложение строит только график и крест в центре красного цвета.
Если существуют какие-либо преобразованные параметры с ориентировочными стоимостями, которые являются 0 (для log преобразуйте) и 0 или 1 (для probit или logit преобразуйте), никакие доверительные интервалы не построены для тех оценок параметра.
Для получения дополнительной информации об определениях различных состояний смотрите Состояние Оценки Доверительного интервала Параметра.
Можно также изменить Layout графика в Plot Settings.
'split' размещение отображает доверительный интервал для каждой оценки параметра на отдельной оси.
'grouped' размещение отображает все доверительные интервалы на одной оси, сгруппированной оценками параметра. Каждый предполагаемый параметр разделяется вертикальной черной линией.
В обоих случаях границы параметра, заданные в исходной подгонке, отмечены квадратными скобками. Приложение использует вертикальные пунктирные линии, чтобы сгруппировать доверительные интервалы оценок параметра, которые были вычислены в общей подгонке.
Точно так же постройте 95% доверительных интервалов для предсказаний модели. Нажмите predictionCI в Browser разделяют на области и выбирают Confidence в разделе Plot.
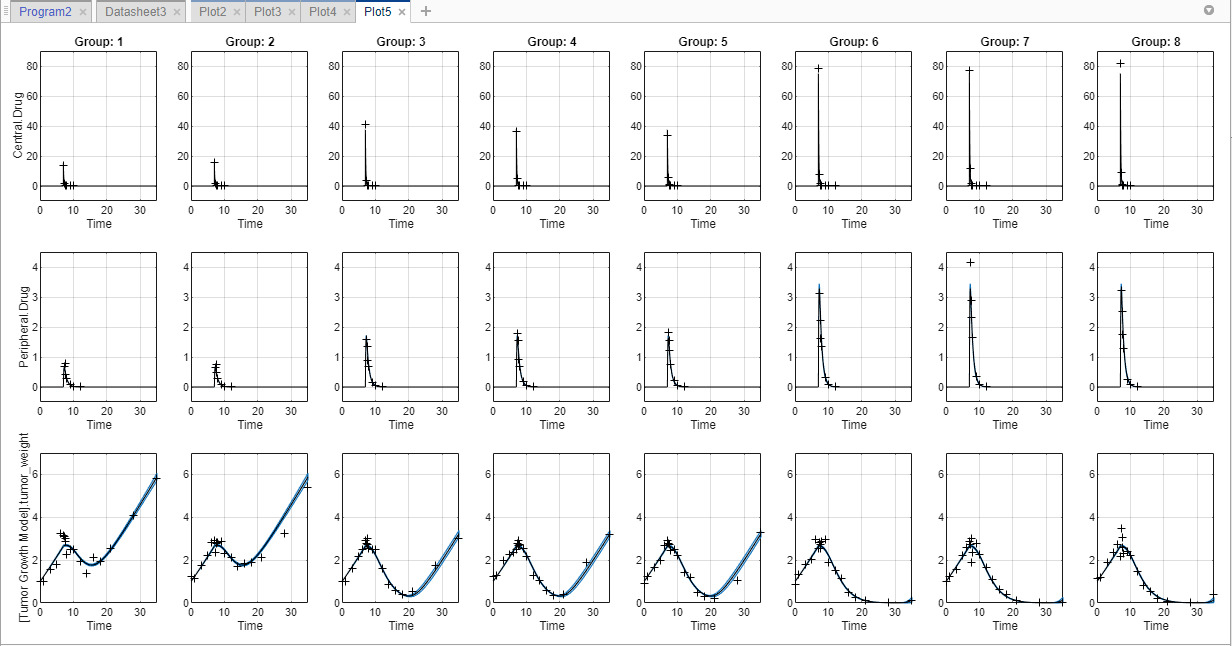
В этом графике доверительный интервал для каждой группы построен в отдельном столбце, и каждый ответ построен в отдельной строке. График показывает успешный расчет доверительных интервалов.
Поведение графического вывода отличается в зависимости от результата (состояние) вычисления доверительного интервала.
Если состояние ограничивается или не допускающее оценку, приложение использует второй цвет по умолчанию (красный), чтобы построить доверительные интервалы.
В противном случае приложение использует первый (синий) цвет по умолчанию и строит доверительные интервалы как заштрихованные области (как в вышеупомянутом графике).
Для получения дополнительной информации смотрите Гауссово Вычисление Доверительного интервала для Предсказаний Модели и Вычисление Доверительного интервала Начальной загрузки.
График процентили позволяет вам визуализировать результаты симуляции и статистику, которая может быть наложена с экспериментальными данными. Например, можно построить 5-е и 95-е кривые распределения данных моделирования в зависимости от времени вместо того, чтобы видеть отдельные графики временной зависимости. Можно также визуализировать среднее значение, стандартное отклонение, минимум и максимум данных моделирования и экспериментальных данных. Для получения дополнительной информации см. График Процентили.
Примечание
Если вы не завершили предшествующие шаги, которые сгенерировали необходимые результаты продолжиться, можно загрузить завершенный проект вместо этого.
Откройте приложение SimBiology Model Analyzer.
Нажмите Open и перейдите к папке matlabroot\examples\simbio\data\. matlabroot является папкой, где вы установили MATLAB. Выберите файл с именем проекта tumor_growth_fitPKPD_completed.sbproj.
В папке LastRun подходящей программы выберите simDataI> [Tumor Growth Model].tumor_weight.
На вкладке Home нажмите percentile.
![]()
График процентили показывает 5-е и 95-е кривые распределения по умолчанию.
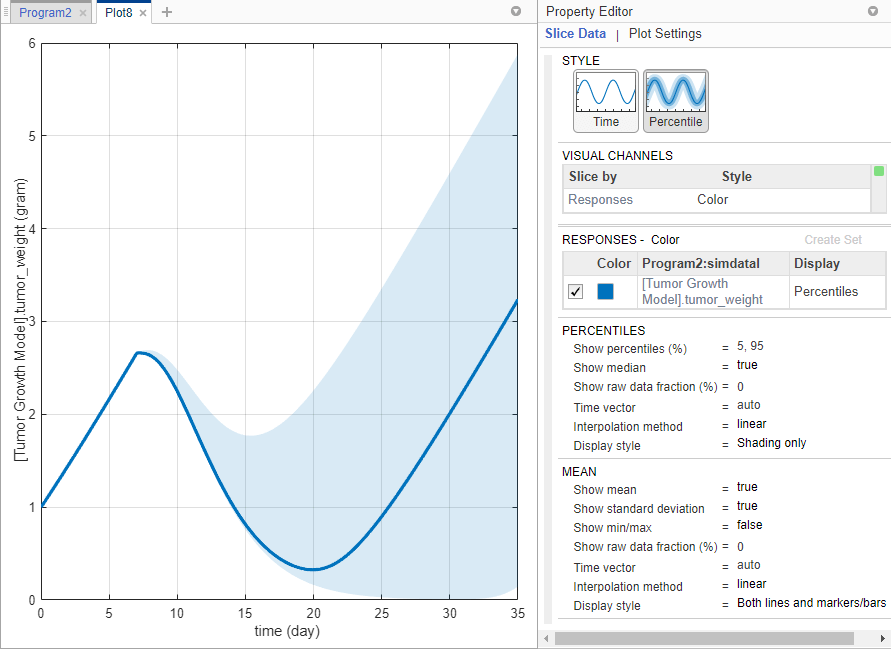
Выберите Data1> TumorWeight. Перетащите мышью его на графике.
По умолчанию тип Display экспериментальных данных автоматически установлен в Mean, который показывает среднее измерение каждый раз с ±1 стандартным отклонением. Если вы хотите визуализировать исходные точки данных, дважды кликнуть Mean и выбрать Raw Data. В данном примере сохраните отображение Mean.
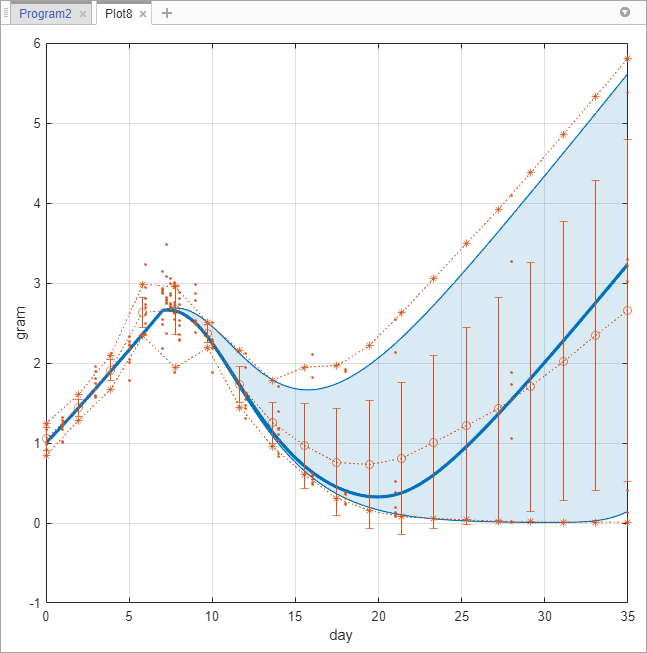
В разделе Percentiles, для опции Show percentiles (%), вводят 10,80 показать 10-е и 80-е процентили.
Измените Display style в Both lines and shading.

В разделе Mean покажите минимальные и максимальные точки данных ответа каждый раз установкой Show min/max к true. Также покажите все базовые точки необработанных данных путем изменения Show raw data fraction (%) в 100. Можно также ввести пользовательский номер процента.

График процентили теперь показывают можно следующим образом. Звездочки (*) представляйте расчетные минимальные и максимальные значения после интерполяции и точек (.) представляйте исходные точки данных. Поскольку эти минимальные и максимальные значения вычисляются на основе интерполированного общего временного вектора, значения не соответствуют точно с теми из необработанных данных. Для получения дополнительной информации см. Средние Опции.
После оценки параметра можно установить значения модели к оценкам параметра и выполнить другие исследования. Например, можно узнать важные параметры модели с помощью анализа чувствительности и варьироваться чувствительные параметры, чтобы исследовать изменчивость модели при помощи виртуальных пациентов.
[1] Simeoni, Моника, Паоло Магни, Криштиану Каммиа, Джузеппе Де Николао, Шафраны Valter, Энрико Пезенти, Массимилиано Джермани, Итало Поджези и Маурицио Роккетти. “Прогнозирующее Фармакокинетическо-фармакодинамическое Моделирование кинетики Роста Опухоли в Моделях Ксенотрансплантата после применения Противораковых Агентов”. Исследования рака 64, № 3 (1 февраля 2004): 1094–1101.