Передискретизируйте сигнал с peaks при сохранении peaks
[X, Intensities]
= msppresample(Peaklist, N)
msppresample(Peaklist, N,
...'Range', RangeValue, ...)
msppresample(Peaklist, N,
...'FWHH', FWHHValue,
...)
msppresample(Peaklist, N,
...'ShowPlot', ShowPlotValue, ...)
Peaklist | Любое из следующего:
СоветМожно использовать функцию |
N | Целое число, задающее количество равномерно распределенных точек (разделительные стоимости единицы) в передискретизируемом сигнале. |
RangeValue | 1 2 вектор, задающий минимальные и максимальные разделительные стоимости единицы для выходной матрицы Intensities. RangeValue должен быть в inputSU является конкатенированными разделительными стоимостями единицы от входа Peaklist. Значением по умолчанию является полный спектр |
FWHHValue | Значение, которое задает полную ширину на половине высоты (FWHH) в разделительных модулях. FWHH используется, чтобы преобразовать каждый пик в кривую гауссовой формы. Значением по умолчанию является inputSU является конкатенированными разделительными стоимостями единицы от входа Peaklist. Значением по умолчанию является грубое приближение разрешения, наблюдаемого во входных данных, Peaklist.СоветЧтобы гарантировать, что разрешение peaks сохраняется, устанавливает |
ShowPlotValue | Управляет отображением графика исходного и передискретизируемого сигнала. Выбором является true, false или I, целое число, задающее индекс сигнала в Intensities. Если вы устанавливаете на true, первый сигнал в Intensities построен. Значение по умолчанию:
|
X | Вектор равномерно распределенных, общих разделительных стоимостей единицы для набора сигналов с peaks. Число элементов в векторе равняется N или количеству строк в матричном Intensities. |
Intensities | Матрица восстановленных значений интенсивности для набора peaks, который совместно использует ту же область значений разделительного модуля. Каждая строка соответствует разделительной стоимости единицы, и каждый столбец соответствует или набору сигналов с peaks или время задержания. Количество строк равняется N или числу элементов в векторном X. |
Используйте следующие синтаксисы с данными из любого разделительного метода, который производит данные сигнала, такие как спектроскопия, NMR, электрофорез, хроматография или масс-спектрометрия.
[ передискретизирует X, Intensities]
= msppresample(Peaklist, N)Peaklist, пиковый список, путем преобразования центроидного peaks в полунепрерывный, необработанный сигнал, что консервы достигают максимума информация. Передискретизируемый сигнал имеет N равномерно распределенные точки. Вывод X является вектором элементов N, задающих равномерно распределенные, общие разделительные стоимости единицы для набора сигналов с peaks. Вывод Intensities является матрицей восстановленных значений интенсивности для набора peaks, который совместно использует ту же область значений разделительного модуля. Каждая строка соответствует разделительной стоимости единицы, и каждый столбец соответствует или набору сигналов с peaks или время задержания. Количество строк равняется N.
msppresample использует Гауссово ядро, чтобы восстановить сигнал. Интенсивность по любой данной разделительной стоимости единицы взята из максимальной интенсивности любого содействия (наложение) peaks.
msppresample полезен, чтобы подготовить набор сигналов для обработки изображений функций, таких как msheatmap и предварительная обработка функций, таких как msbackadj и msnorm.
msppresample(Peaklist, N, ... 'PropertyName', PropertyValue, ...) msppresample с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
msppresample( задает область значений разделительного модуля для выходной матрицы Peaklist, N,
...'Range', RangeValue, ...)Intensities с помощью минимальных и максимальных разделительных значений, заданных в 1 2 векторном RangeValue. RangeValue должен быть в [min(inputSU) max(inputSU)]inputSU является конкатенированными разделительными стоимостями единицы от входа Peaklist. Значением по умолчанию является полный спектр [min(inputSU) max(inputSU)]
msppresample( устанавливает полную ширину на половине высоты (FWHH) в разделительных модулях. FWHH используется, чтобы преобразовать каждый пик в кривую гауссовой формы. Значением по умолчанию является Peaklist, N,
...'FWHH', FWHHValue,
...)median(diff(inputSU))/2inputSU является конкатенированными разделительными стоимостями единицы от входа Peaklist. Значением по умолчанию является грубое приближение разрешения, наблюдаемого во входных данных, Peaklist.
Чтобы гарантировать, что разрешение peaks сохраняется, устанавливает FWHHValue на половину расстояния между двумя peaks интереса, которые являются самыми близкими друг к другу.
msppresample( управляет отображением графика исходного и передискретизируемого сигнала. Выбором является Peaklist, N,
...'ShowPlot', ShowPlotValue, ...)true, false или I, целое число, задающее индекс сигнала в Intensities. Если вы устанавливаете на true, первый сигнал в Intensities построен. Значение по умолчанию:
ложь Когда возвращаемые значения заданы.
tRUE Когда возвращаемые значения не заданы.
Загрузите MAT-файл, включенный с программным обеспечением Bioinformatics Toolbox™, которое содержит жидкостную хроматографию / переменные данных (LC/MS) масс-спектрометрии. Это включает peaks, массив ячеек пиковых списков, где каждый элемент является матрицей 2D столбца m/z значений и ионных значений интенсивности, и каждый элемент соответствует время задержания или спектру.
load lcmsdataПередискретизируйте данные, задавая 5 000 m/z значений в передискретизируемом сигнале. Затем создайте карту тепла данных LC/MS.
[MZ,Y] = msppresample(ms_peaks,5000); msheatmap(MZ,ret_time,log(Y))
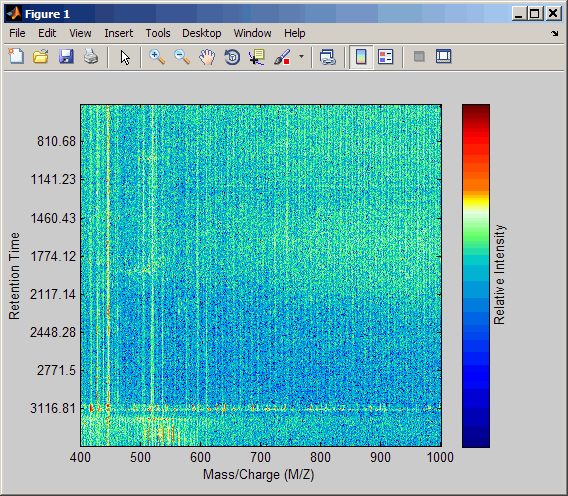
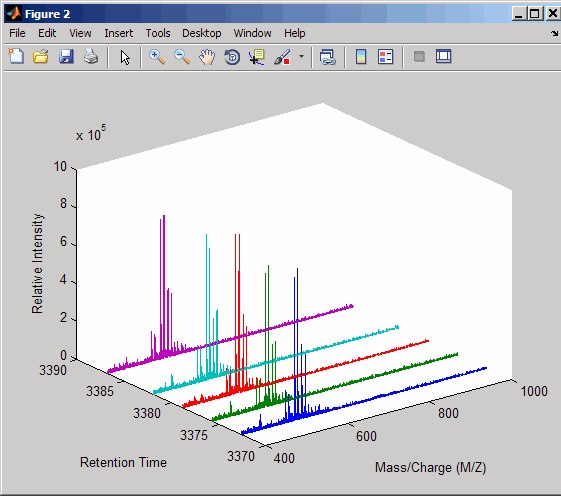
Постройте восстановленные спектры профиля между двумя разами задержания.
figure t1 = 3370; t2 = 3390; h = find(ret_time>t1 & ret_time<t2); [MZ,Y] = msppresample(ms_peaks(h),10000); plot3(repmat(MZ,1,numel(h)),repmat(ret_time(h)',10000,1),Y) xlabel('Mass/Charge (M/Z)') ylabel('Retention Time') zlabel('Relative Intensity')
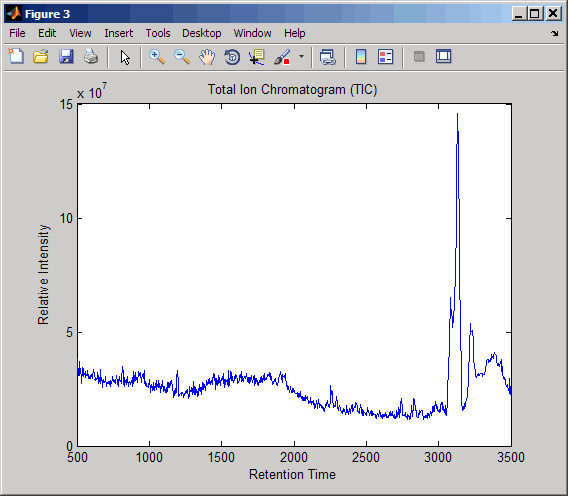
Передискретизируйте данные, чтобы построить Общую ионную хроматограмму (TIC).
figure [MZ,Y] = msppresample(ms_peaks,5000); plot(ret_time,sum(Y)) title('Total Ion Chromatogram (TIC)') xlabel('Retention Time') ylabel('Relative Intensity')
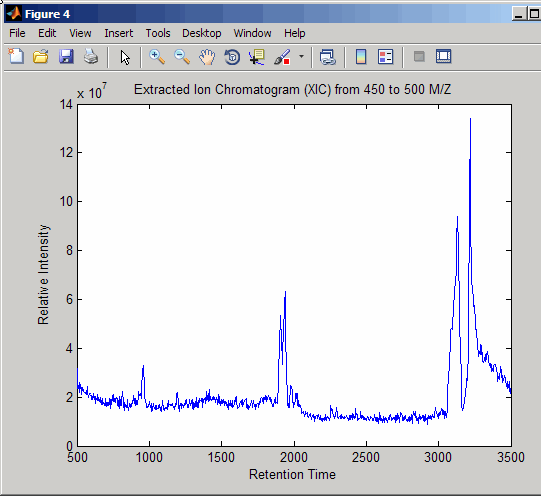
Передискретизируйте данные, чтобы построить Извлеченную Ионную Хроматограмму (XIC) в 450 - 500 областях значений m/z.
figure [MZ,Y] = msppresample(ms_peaks,5000,'Range',[450 500]); plot(ret_time,sum(Y)) title('Extracted Ion Chromatogram (XIC) from 450 to 500 M/Z') xlabel('Retention Time') ylabel('Relative Intensity')
msdotplot | mspalign | mspeaks | msresample | mzcdf2peaks | mzcdfread | mzxml2peaks | mzxmlread