Класс: geneont
Найти термины, которые являются предками указанного термина генной онтологии (GO)
AncestorIDs = getancestors(GeneontObj, ID)
[AncestorIDs, Counts] = getancestors(GeneontObj, ID)
... = getancestors(..., 'Height', HeightValue, ...)
... = getancestors(..., 'Relationtype', RelationtypeValue, ...)
... = getancestors(..., 'Exclude', ExcludeValue, ...)
AncestorIDs = getancestors(GeneontObj, ID)GeneontObj, объект geneont, для терминов GO, которые являются предками терминов GO, указанных ID, который является идентификатором термина GO или вектором идентификаторов. Возвращается AncestorIDsвектор идентификаторов терминов GO, включающий ID. ID - неотрицательное целое число или вектор, содержащий неотрицательные целые числа.
[ также возвращает число найденных предков. AncestorIDs, Counts] = getancestors(GeneontObj, ID)Counts - вектор столбца с тем же количеством элементов, что и члены в GeneontObj.
Совет
Counts возвращаемое значение полезно, когда вы подсчитываете количество в исследованиях по обогащению генов. Дополнительные сведения см. в разделе Обогащение генной онтологии в данных микрочипов.
... = getancestors(..., ' требования PropertyName', PropertyValue, ...)getancestors с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
... = getancestors(..., 'Height', выполняет поиск по указанному количеству уровней, HeightValue, ...)HeightValue, в генной онтологии. HeightValue является положительным целым числом. По умолчанию: Inf.
... = getancestors(..., 'Relationtype', поиск указанных типов отношений, RelationtypeValue, ...)RelationtypeValue, в генной онтологии. RelationtypeValue - символьный вектор. Варианты: 'is_a', 'part_of', или 'both' (по умолчанию).
... = getancestors(..., 'Exclude', элементы управления, исключая ExcludeValue, ...)ID, исходный запрашиваемый термин (ы), из выходных данныхAncestorIDs, если только термин не был достигнут во время поиска в генной онтологии. Варианты: true или false (по умолчанию).
GeneontObj | Генеонтный объект, например, созданный geneont функция конструктора. |
ID | Идентификатор термина GO или вектор идентификаторов. |
HeightValue | Положительное целое число, указывающее количество уровней для поиска вверх в генной онтологии. |
RelationtypeValue | Вектор символов, указывающий типы отношений для поиска в генной онтологии. Возможны следующие варианты:
|
ExcludeValue | Элементы управления, исключая ID, исходный запрашиваемый термин (ы), из выходных данныхAncestorIDs, если только термин не был достигнут во время поиска в генной онтологии. Варианты: true или false (по умолчанию). |
AncestorIDs | Вектор идентификаторов терминов GO, включая ID. |
Counts | Вектор столбца с тем же количеством элементов, что и члены в GeneontObj, указывая количество находок каждого предка. |
Загрузите текущую версию базы данных Gene Ontology из Интернета в объект geneont в программном обеспечении MATLAB ®.
GO = geneont('LIVE', true)Программное обеспечение MATLAB создает объект geneont и отображает количество терминов в базе данных.
Gene Ontology object with 24316 Terms.
Получить предков термина генной онтологии с идентификатором 46680.
ancestors = getancestors(GO,46680)
ancestors =
8150
9636
17085
42221
46680
50896Создание подчиненной генной онтологии.
subontology = GO(ancestors) Gene Ontology object with 6 Terms.
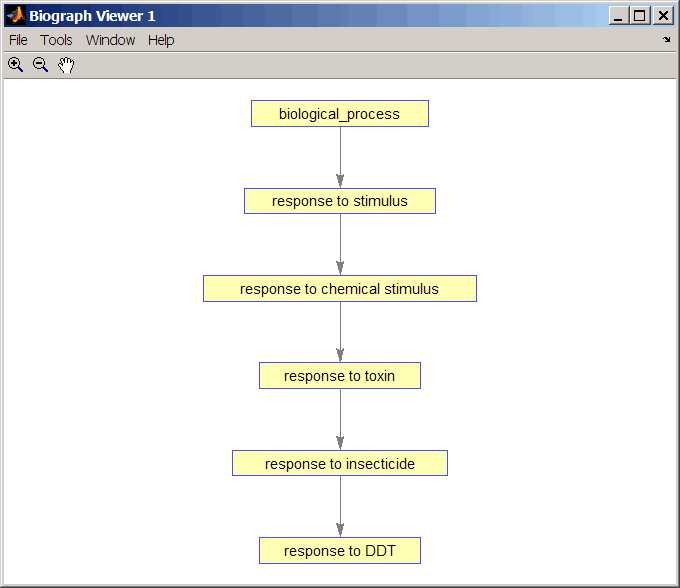
Создайте и отобразите отчет о зависимых терминах генной онтологии, который включает идентификатор и имя GO.
rpt = get(subontology.terms,{'id','name'})
[ 8150] 'biological_process'
[ 9636] 'response to toxin'
[17085] [1x23 char]
[42221] [1x29 char]
[46680] 'response to DDT'
[50896] [1x20 char]
Просмотр отношений подчиненной онтологии генов с помощью getmatrix метод создания матрицы соединений для передачи в biograph функция.
cm = getmatrix(subontology); BG = biograph(cm, get(subontology.terms, 'name')); view(BG)
goannotread | num2goid | term