Этот пример показывает, как добавить ингибирование SGLT2 гипотетическим соединением к существующей модели глюкоза-инсулин с использованием SimBiology Model Builder.
Эта модель является другой реализацией SimBiology модели глюкоза-инсулин, на которую ссылаются в примере моделирования ответа глюкоза-инсулин. Модель основана на публикации Dalla Man et al. В своей публикации 2007 года [1] авторы разработали модель ответа глюкоза-инсулин человека после еды. Эта модель описывает динамику системы с использованием обыкновенных дифференциальных уравнений. Авторы использовали свою модель для моделирования глюкозно-инсулиновой реакции после одного или нескольких приемов пищи для нормальных людей и для людей с различными видами нарушений инсулина.
Показано, что рецептор SGLT2 облегчает около 50% реабсорбции глюкозы в почках [3]. Этот пример предполагает наличие гипотетического соединения ингибитора SGLT2, которое ингибирует SGLT2 на 50%. Также предполагается приемлемый режим дозирования и свойства PK. В этом примере в модель глюкоза-инсулин включается фармакокинетика/фармакодинамика (PK/PD) этого соединения-ингибитора.
На следующих этапах моделируется поглощение соединения и клиренс гипотетического соединения ингибитора SGLT2 с помощью двух реакций.
Откройте приложение Построитель моделей SimBiology, щелкнув Построитель моделей SimBiology на вкладке Приложения или введя simBiologyModelBuilder в командной строке.
На вкладке Домашняя страница приложения выберите Открыть.
Перейти к папке matlabroot\examples\simbio\data\. matlabroot - папка, в которой установлен MATLAB. Вход matlabroot в командной строке указывается путь к корневой папке. Выберите файл проекта с именем SGLT2_model_incomplete.sbproj. Щелкните Открыть (Open).
Примечание
В macOS вместо клавиши Ctrl используйте клавишу командной строки.
Перетащите два блока видов с панели инструментов вкладки «Схема».
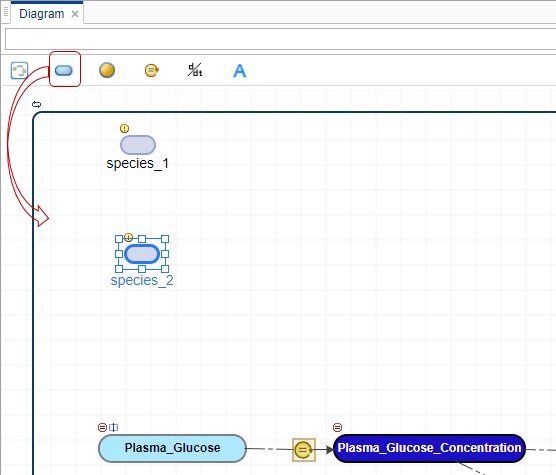
Нажмите Ctrl и перетащите линию из первого вида во второй вид. Между ними появляется реакционный блок. Эта реакция представляет собой абсорбцию соединения.
Измените имя вида по умолчанию, дважды щелкнув его. Переименовать species_1 кому GI_SGLT2_Inhib и species_2 кому Plasma_SGLT2_Inhib.
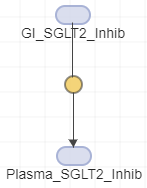
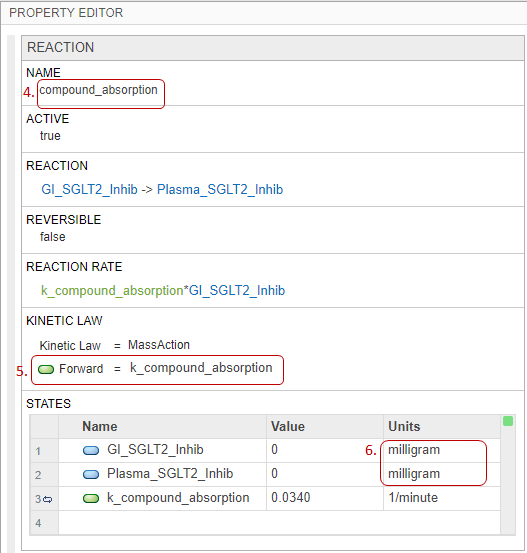
Щелкните блок реакции. В правой панели редактора свойств измените имя реакции на compound_absorption.
В разделе Kinetic Law измените автоматически созданный параметр Forward rate kf на k_compound_absorption. Модель уже имеет параметр форвардной скорости k_compound_absorption который был создан ранее. Приложение использует зеленый текст для имен параметров и синий текст для имен видов в выражении скорости реакции.
Совет
Чтобы изменить конфигурации реакции по умолчанию, щелкните Настройки (Preferences) на вкладке Главная (Home). В диалоговом окне «Установки» выберите «Модель здания». В разделе Здание реакции (Reaction Building) имеется три варианта изменения кинетического закона по умолчанию, создания параметров для кинетического закона и изменения объема создаваемых параметров.
В таблице Штатов установить единицы двух видов как milligram.
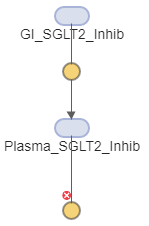
Перетащите другой блок реакции с панели инструментов диаграммы, чтобы смоделировать составной зазор.
Нажмите клавишу Ctrl и перетащите строку из Plasma_SGLT2_Inhib на реакцию.
Совет
Если блоки не выровнены, их можно выровнять с помощью инструментов выравнивания. На вкладке Главная выберите Инструменты схемы > Diagram Alignment Tools.
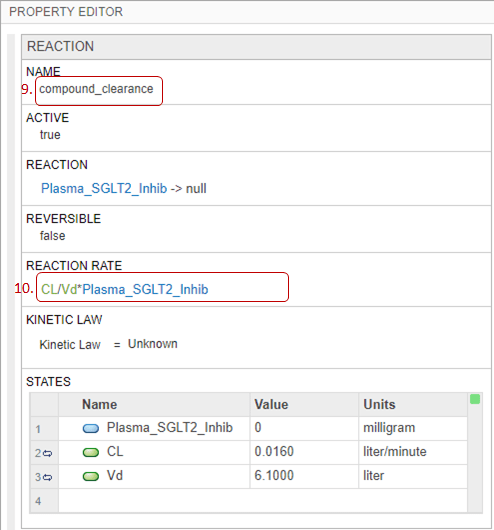
Щелкните блок реакции. На панели «Редактор свойств» измените имя реакции на compound_clearance.
Обновить скорость реакции до CL/Vd*Plasma_SGLT2_Inhib. CL и Vd указывают параметры модели для зазора и объема распределения соответственно.
Совет
Кинетический закон для только что добавленной реакции по умолчанию настроен на MassAction, и приложение SimBiology Model Builder автоматически создает и отображает виды и параметры, необходимые для скорости реакции. Для других кинетических законов создаются и отображаются только параметры. Необходимо создать и отобразить вид вручную. Для определения пользовательской скорости реакции с собственными параметрами используется кинетический закон Unknown. Необходимо определить и добавить виды и параметры, необходимые для пользовательской ставки.
Кинетические законы MassAction и Unknown могут иметь разные результаты моделирования, даже если скорость реакции одинакова. Это может произойти, когда происходит обратимая реакция с видами в разных отсеках. Разница в результатах моделирования обусловлена масштабированием объема, выполненным SimBiology во время анализа размеров. Для получения дополнительной информации см. Деривация ОДУ из реакций. В частности, для MassAction SimBiology использует соответствующие объемы отсека для умножения прямой и обратной скоростей. Однако для неизвестных и других встроенных кинетических законов SimBiology умножает всю скорость только на один отсек, который содержит реагенты. Чтобы точно увидеть, какие объемы отсека используются для масштабирования, откройте вкладку «Уравнения» и проверьте раздел ODE.
SimBiology позволяет определить математическое выражение для определения или обновления значения величины модели во время моделирования. Дополнительные сведения см. в разделе Определения и оценки правил в моделях SimBiology. На следующих этапах добавляется правило повторного назначения для включения фармакодинамики ингибитора путем определения почечного порога, при котором глюкоза в плазме выводится на основе эффективности соединения.
На панели «Обозреватель» щелкните значок «плюс» на панели инструментов обозревателя и выберите Add Repeated Assignment. Приложение перемещает фокус на последнюю пустую строку в таблице Повторяющиеся назначения.
Дважды щелкните строку и введите следующее выражение, представляющее ингибирование соединения на основе уравнения Хилла:
renal_threshold = basal_renal_threshold*(1-compound_Imax*Plasma_SGLT2_Inhib^2/(compound_IC50^2+Plasma_SGLT2_Inhib^2))
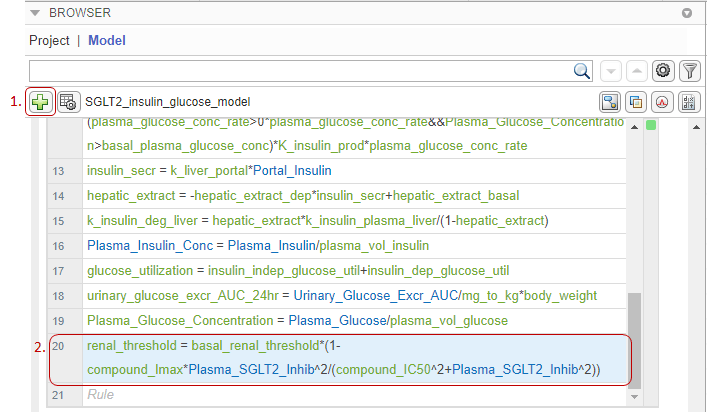
На вкладке Схема теперь отображается повторяющийся блок правил назначения для renal_threshold параметр.
Совет
Чтобы просмотреть всю модель и выполнить панорамирование, разверните «Инструменты оценки модели» на панели «Обозреватель» и нажмите «Обзор».
Примечание
Приложение показывает только блок параметров для параметра, который находится в левой части (LHS) повторяющегося правила назначения, правила скорости или функции события.
Приложение показывает только блоки правил для повторяющихся назначений и правил тарифов.
Приложение использует линии тире-точки для соединения величин в правой части правила. По умолчанию эти строки не отображаются. Чтобы отобразить строки, щелкните блок правил. На панели «Редактор свойств» в разделе «Блок» задайте для параметра «Строки выражения» значение «Показать».
Реакция почечной экскреции модели в настоящее время определяется как Plasma_Glucose -> Urinary_Glucose_Excr_AUC с параметром скорости реакции glucose_excretion. Параметр скорости определяется повторяющимся правилом назначения как glucose_excretion = (Plasma_Glucose>renal_threshold)*GFR*(Plasma_Glucose-renal_threshold), где GFR - скорость клубочковой фильтрации, которая определяет поток реакции и оказывает влияние на эффект ингибирования SGLT2.
В следующих шагах вы обновите реакцию почечной экскреции до Plasma_Glucose + Plasma_SGLT2_Inhib -> Plasma_SGLT2_Inhib + Urinary_Glucose_Excr_AUC, где соединение-ингибитор Plasma_SGLT2_Inhib является как реагентом, так и продуктом реакции.
На вкладке «Схема» щелкните серый квадратный блок реакции с именем Renal excretion.
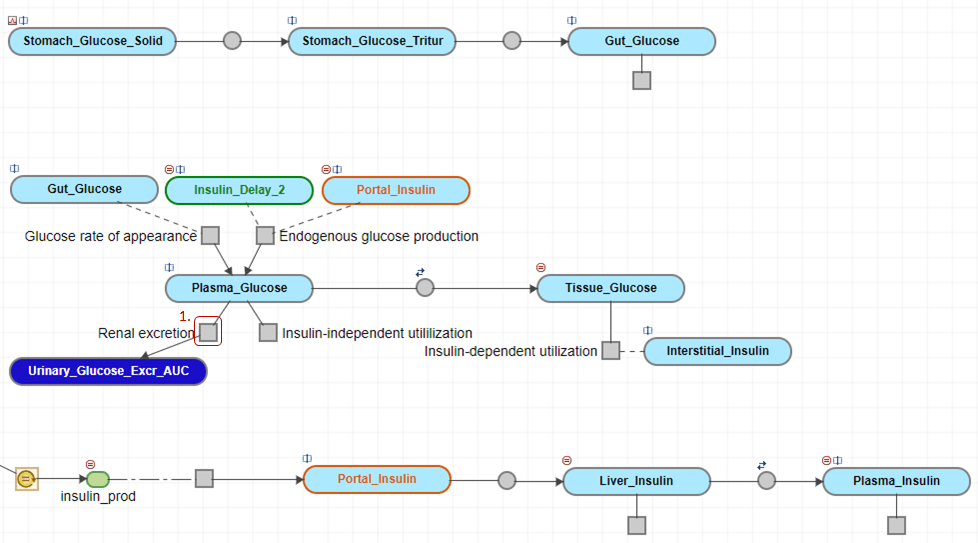
На панели «Редактор свойств» обновите строку «Реакция» до Plasma_Glucose + Plasma_SGLT2_Inhib -> Plasma_SGLT2_Inhib + Urinary_Glucose_Excr_AUC. Теперь подключается пунктирная линия Plasma_SGLT2_Inhib к блоку реакции на вкладке «Схема».
Примечание
SimBiology использует пунктирную линию, чтобы указать, что разновидность является как реагентом, так и продуктом реакции и не потребляется реакцией.
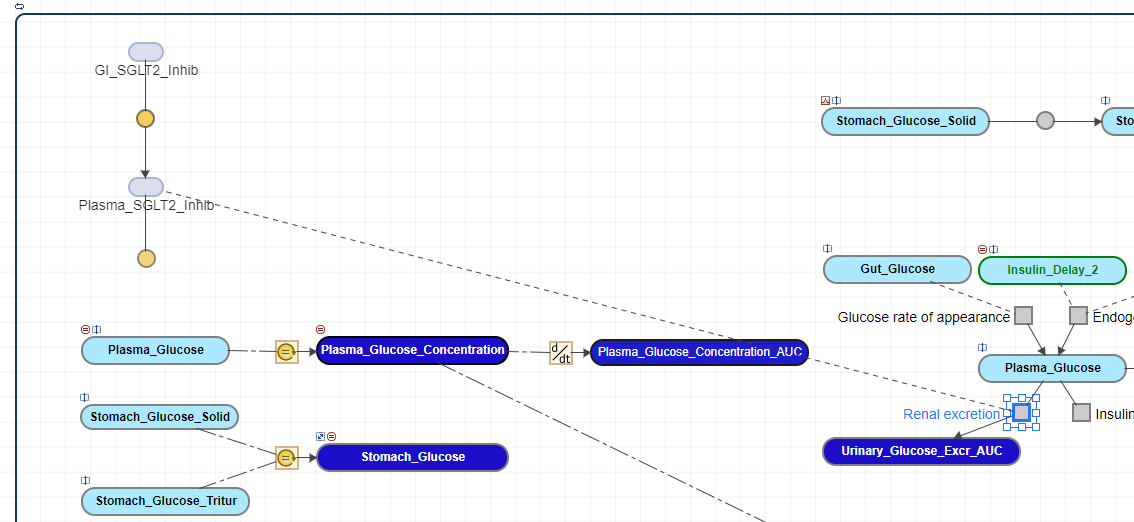
При наличии нескольких ссылок на одну и ту же величину к блоку подключается несколько линий. Чтобы сделать схему более четкой, можно разделить блок, то есть создать копии одного и того же блока, чтобы каждая ссылка была соединена с другой копией блока. Можно также клонировать блок, чтобы добавить для него другое использование. Например, можно сначала клонировать видовой блок для ссылки в нескольких выражениях. Затем можно использовать каждый клон в каждом выражении при построении модели.
На следующих шагах выполняется клонирование Plasma_SGLT2_Inhib блок. Эти шаги являются необязательными и не влияют на поведение модели.
Щелкните значок Plasma_SGLT2_Inhib блок на схеме.
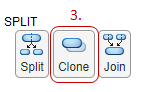
На панели «Редактор свойств» выполните прокрутку до раздела «Разделить».
Щелкните Клон (Clone).
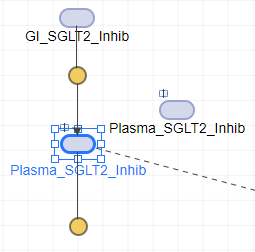
На вкладке «Схема» рядом с исходным блоком появляется клонированный блок. Теперь каждый блок имеет индикатор клона.
Теперь можно переместить пунктирную линию в клонированный блок. Сначала щелкните пунктирной линией. Нажмите клавишу Ctrl и перетащите пунктирную линию в клонированный блок. Когда линия находится рядом с клонированным блоком, появляется зеленый значок «плюс». Отпустите мышь, чтобы прикрепить линию к клонированному блоку.
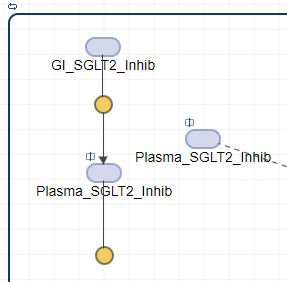
Теперь можно переместить клонированный блок ближе к Renal excretion блок реакции, чтобы облегчить считывание диаграммы.
Можно моделировать внезапные изменения в поведении модели на основе заданного условия. Например, можно сбросить количество видов в определенный момент времени или при пересечении определенного порога концентрации. SimBiology позволяет моделировать такие изменения с помощью компонента моделирования, называемого событием. Событие позволяет указать дискретные переходы в значениях количества, которые возникают, когда пользовательское условие становится верным. Такое условие называется триггером события. Как только условие становится истинным, выполняется одна или несколько функций события. Дополнительные сведения см. в разделе События в моделях SimBiology.
В следующих шагах вы сбрасываете общее количество глюкозы в моче на ноль каждые 24 часа, добавляя один триггер события и три функции события.
Щелкните значок «плюс» на панели инструментов браузера. Выбрать Add Event. Приложение перемещает фокус на последнюю пустую строку в таблице События.
Введите следующий триггер события: floor(time/timeDay)>num_day.
В следующей строке EventFcn введите Urinary_Glucose_Excr_AUC = 0.
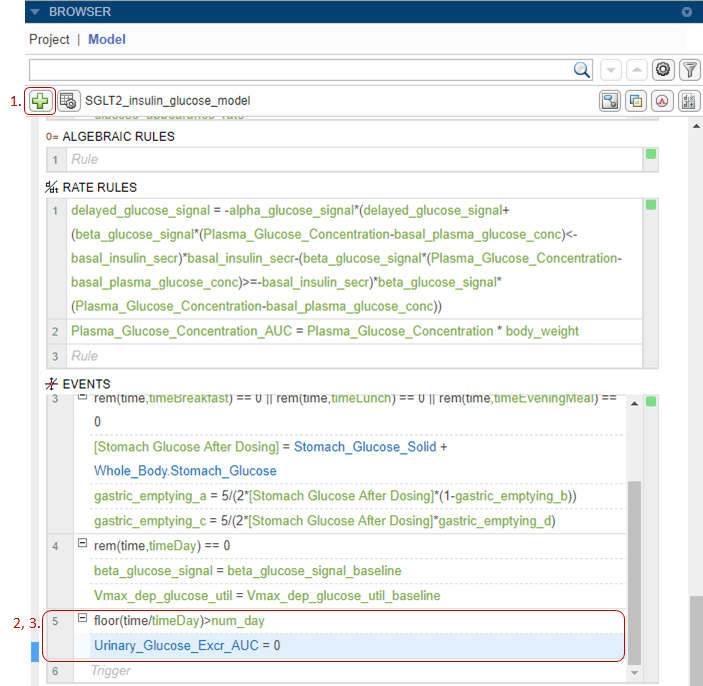
Чтобы добавить вторую функцию события к тому же событию, перейдите на панель редактора свойств события. В таблице Event Fcns дважды щелкните пустую строку и введите следующее для сброса количества дней: num_day = floor(time/1440).
Добавьте третью функцию события, чтобы сбросить общую концентрацию глюкозы в плазме, введя Plasma_Glucose_Concentration_AUC = 0.
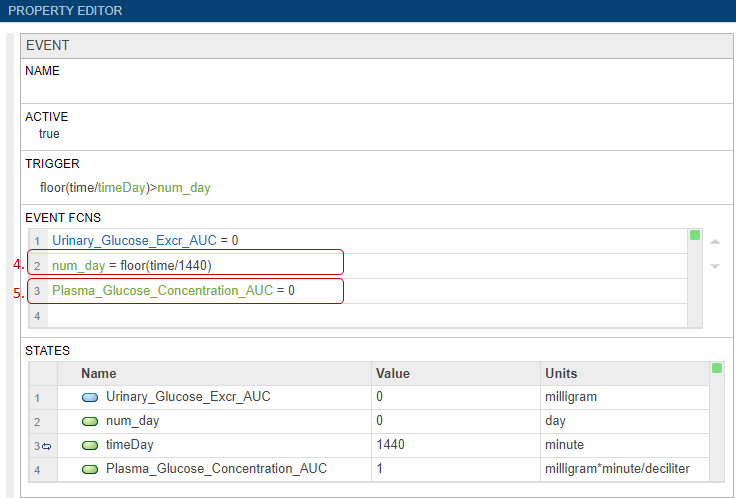
SimBiology позволяет моделировать увеличение количества вида за счет стимула, такого как пероральное или внутривенное введение препарата. Для моделирования такого увеличения видового количества используйте компонент моделирования дозы. На следующих шагах вы моделируете потребление препарата-ингибитора, например, один раз в день в течение x дней, путем дозирования GI_SGLT2_inhib виды.
На панели «Обозреватель» щелкните значок «Показать дозы» на панели инструментов.
![]()
Откроется вкладка Дозы (Doses). В разделе «Дозы» каждая строка представляет дозу. В столбце Тип (Type) можно выбрать опцию Повторная доза (Repeat Dose) (по умолчанию) и Запланированная доза (Schedule Dose) В столбце Активный (Active) можно выбрать дозы, применяемые при моделировании модели.
Дважды щелкните столбец «Имя» в последней пустой строке и введите SGLT2 Inhib QD.
В разделе «Свойства» в поле «Имя цели» введите GI_SGLT2_Inhib и выбрать Whole_Body.GI_SGLT2_Inhib.
В разделе Доза введите следующее:
Сумма = 300
Ставка = 0
StartTime = 60
Интервал = 1440
RepeatCount = 7
В разделе «Единицы» введите следующее:
Единицы измерения = milligram
RateUnits =
TimeUnits = minute
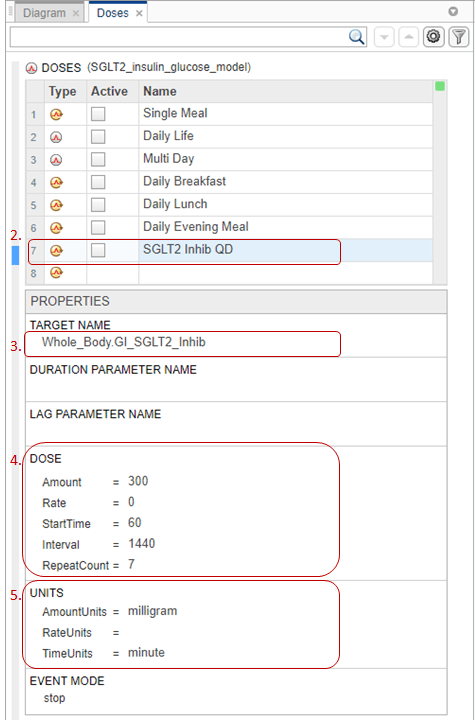
Биологическую изменчивость можно моделировать с помощью компонента моделирования, называемого вариантом. Вариант - это набор количеств с альтернативными значениями. Например, в этом примере можно иметь набор значений параметров для пациента с диабетом 2 типа и другой набор значений для пациента без диабета 2 типа.
Для целей этого примера модель уже имеет два варианта. На следующих шагах открывается вкладка Исполнения, на которой можно править или добавлять дополнительные исполнения.
На панели «Обозреватель» щелкните значок «Показать варианты» на панели инструментов.
![]()
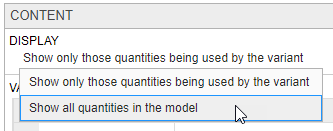
Появится вкладка Варианты (Variants). В разделе Исполнения каждая строка представляет вариант. Столбец Активный (Active) позволяет выбрать варианты для применения при моделировании модели. Можно выбрать несколько вариантов, и при наличии повторяющихся спецификаций для значения количества во время моделирования используется последнее вхождение значения в массиве вариантов. Приложение применяет варианты в том порядке, в котором они отображаются в таблице сверху вниз. Переупорядочивание вариантов может изменить начальные условия, поскольку варианты применяются в новом порядке. Убедитесь, что при моделировании модели в приложении Model Analyzer указан правильный порядок. В столбце Значение (Value) раздела Содержимое (Content) отображается итоговое значение количества после применения всех выбранных вариантов.
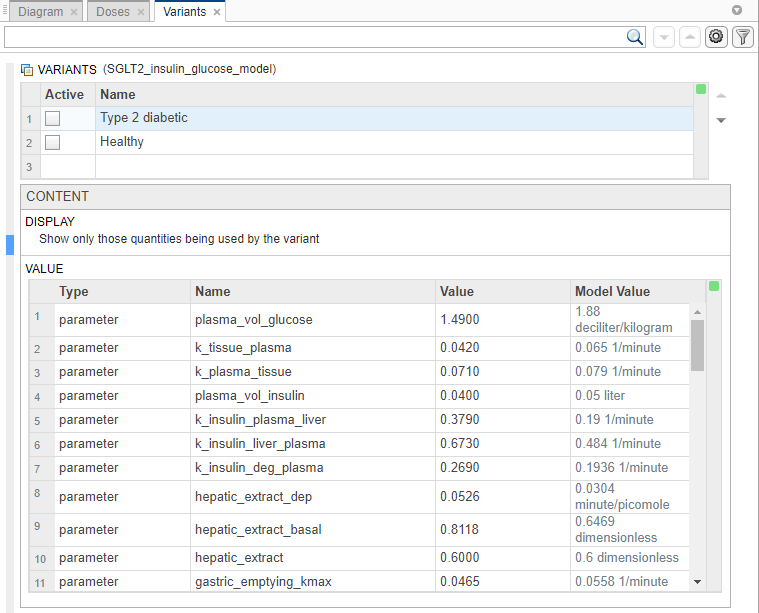
По умолчанию в разделе Содержимое отображаются только те количества, которые изменяются вариантом. Для просмотра всех величин модели выберите Show all quantities in the model в разделе «Отображение».
Можно просмотреть базовую систему уравнений, а именно обычные дифференциальные уравнения (ОДУ) и правила, представляющие модель. SimBiology извлекает ОДУ из реакций модели, а ОДУ определяют, какие величины необходимо интегрировать во время моделирования модели. Дополнительные сведения см. в разделе Моделирование модели.
Для отладки модели можно использовать уравнения модели и начальные условия. Например, можно проверить начальные условия ODE, чтобы проверить, инициализированы ли значения количества в соответствии с ожиданиями. Можно также увидеть, как SimBiology корректирует размеры ОДУ, разделяя правые стороны уравнений объемами отсека. Информация о коррекции громкости может помочь в отладке неожиданных результатов моделирования, особенно при использовании многоотсековой модели с различными объемами отсека.
Для просмотра уравнений модели щелкните значок «Показать уравнения модели» на панели инструментов обозревателя.
![]()
Приложение открывает вкладку Уравнения.
По умолчанию приложение встраивает потоки реакций, когда они отображаются в уравнениях модели. Снимите флажок «Встроить потоки», чтобы увидеть раздел «Потоки» отдельно.
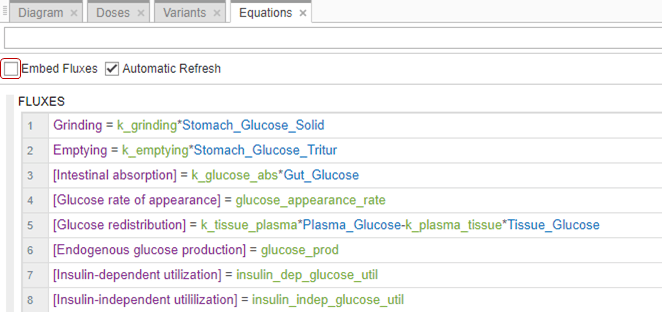
Обычно реакционные потоки эквивалентны скоростям реакции, за исключением того, что размеры потоков всегда amount/time. Размеры скоростей реакции могут быть в concentration/time или amount/time. Для получения дополнительной информации см. Деривация ОДУ из реакций.
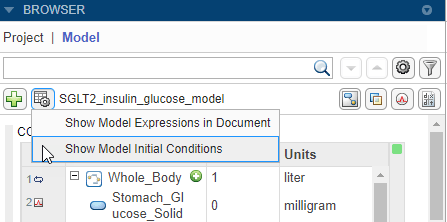
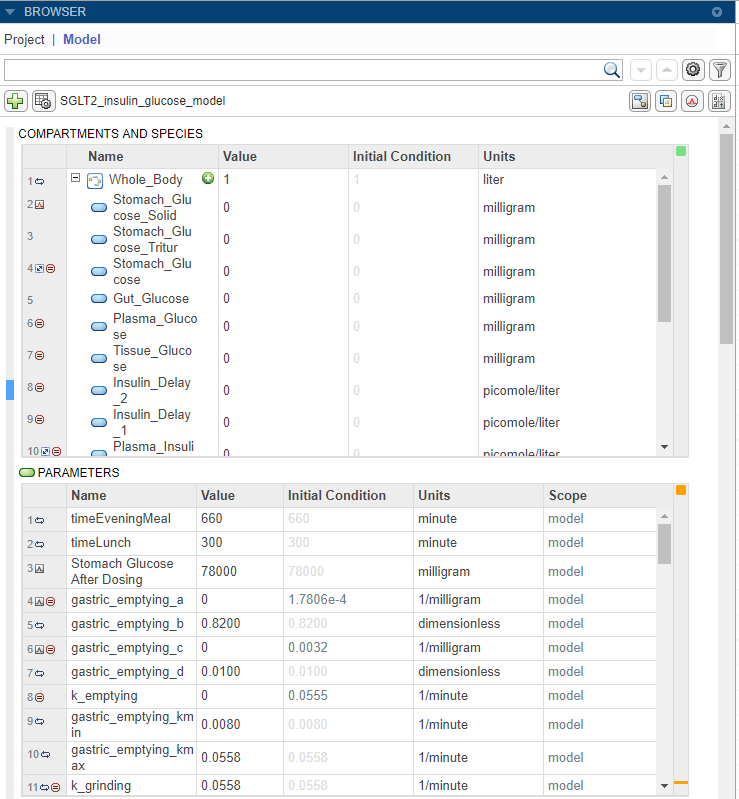
Можно просмотреть начальные условия величин модели, а именно отделения, виды и параметры. Начальными условиями являются значения количества в момент моделирования = 0. На панели инструментов панели «Обозреватель» выберите «Параметры просмотра документов модели» > «Показать начальные условия модели».
Приложение добавляет столбец с именем Начальное условие в таблицу Отделения и Виды и таблицу Параметры.
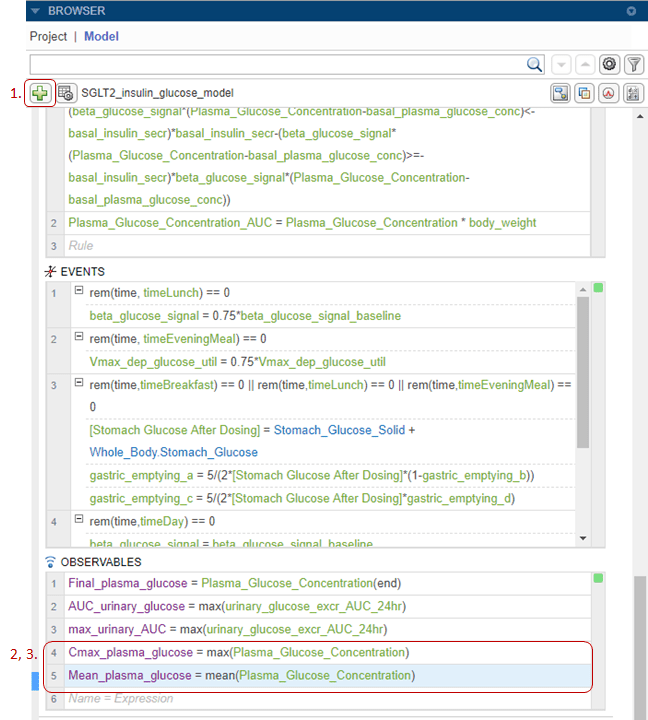
SimBiology позволяет выполнять вычисления постсимуляции путем определения и оценки пользовательских выражений. Такое выражение называется Observable. На следующих шагах в модель добавляются наблюдаемые выражения для вычисления Cmax и средних значений профиля концентрация-время глюкозы в плазме.
Щелкните значок «плюс» на панели инструментов браузера. Выбрать Add Observable. Приложение перемещает фокус на последнюю пустую строку в таблице Наблюдаемые.
Дважды щелкните пустую строку и введите следующее выражение, чтобы получить значение Cmax: Cmax_plasma_glucose = max(Plasma_Glucose_Concentration).
Дважды щелкните следующую пустую строку и введите следующее выражение, чтобы получить среднее значение: Mean_plasma_glucose = mean(Plasma_Glucose_Concentration).
Совет
Чтобы увидеть значения наблюдаемых выражений, смоделируйте модель с помощью приложения SimBiology Model Analyzer. Запустите программу Simulate Model с шагом постобработка: расчет наблюдаемых. В папке LastRun результатов моделирования программы разверните результаты. Векторные наблюдаемые перечислены в результатах. Скалярные наблюдаемые, такие как Cmax и mean в этом примере перечислены в подгруппе с именем scalars. Можно перетащить данные на лист данных или график. Дополнительные примеры анализа модели см. в разделе Анализатор модели SimBiology.
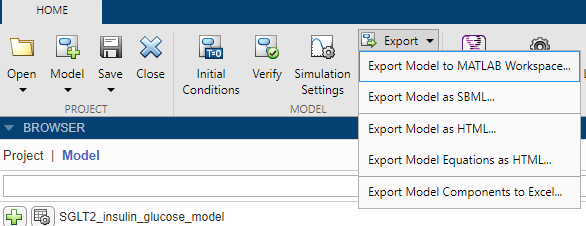
SimBiology Model Builder позволяет экспортировать модель в различные форматы файлов. Вы можете:
Экспортируйте модель в рабочую область MATLAB. Когда модель находится в рабочей области, над ней можно работать программно. Дополнительные примеры командной строки см. в разделе Создание моделей.
Экспортируйте модель в файл SBML.
Экспорт модели в HTML-файл.
Экспорт уравнений модели в HTML-файл.
Экспорт компонентов модели в файлы Excel ®.
На вкладке Главная (Home) в разделе Модель (Model) выберите Экспорт (Export).
Совет
Завершенную модель для этого примера можно найти в matlabroot\examples\simbio\data\SGLT2_model.sbproj, где matlabroot - папка, в которой установлен MATLAB.
[1] Далла Ман, Кьяра, Роберт А. Рицца и Клаудио Кобелли. «Имитационная модель еды системы глюкоза-инсулин». IEEE Transactions on Biomedical Engineering 54, No 10 (октябрь 2007): 1740-49. https://ieeexplore.ieee.org/document/4303268.
[2] Далла Ман, Кьяра, М. Камиллери и К. Кобелли. «Системная модель перорального поглощения глюкозы: валидация данных золотого стандарта». IEEE Transactions on Biomedical Engineering 53, No 12 (декабрь 2006): 2472-78. https://ieeexplore.ieee.org/document/4015600.
[3] Райт, Эрнест М., Дональд Ф. Лоо и Брюс А. Хираяма. «Биология переносчиков глюкозы натрия человека». Физиологические обзоры 91, № 2 (апрель 2011): 733-94. https://journals.physiology.org/doi/full/10.1152/physrev.00055.2009.
Анализатор модели SimBiology | Построитель моделей SimBiology