В этом примере показано, как определить важные параметры модели для модели роста опухоли [1]. В этом примере вычисляются зависящие от местного времени чувствительности роста опухоли по параметрам модели для противоопухолевого препарата.
Модель, используемая в этом примере, представляет собой реализацию SimBiology ® фармакокинетической/фармакодинамической (PK/PD) модели Simeoni et al. Он количественно оценивает влияние противораковых препаратов на кинетику роста опухоли из исследований на животных in vivo. Фармакокинетика препарата описана двухкамерной моделью с внутривенным дозированием болюса и линейным элиминированием (ke) из центрального отделения. Рост опухоли представляет собой двухфазный процесс с начальным экспоненциальным ростом с последующим линейным ростом. Скорость роста пролиферирующих опухолевых клеток описана
w)
L0, L1 и Ψ - параметры роста опухоли, x1 - вес распространяющихся опухолевых клеток, и w - общий вес опухоли. При отсутствии каких-либо лекарств опухоль состоит только из пролиферирующих клеток, то есть w = x1. В присутствии противоракового агента часть пролиферирующих клеток трансформируется в непролиферирующие клетки. Предполагается, что скорость этого превращения зависит от концентрации лекарственного средства в плазме и фактора эффективности k2. Непролиферирующие клетки x2 проходят через ряд транзитных стадий (x3 и x4) и в конечном итоге удаляются из системы. Поток транзитных отсеков моделируется как процесс первого порядка с постоянной скоростью k1.
Модель SimBiology вносит эти коррективы в фармакодинамику роста опухоли:
Вместо определения веса опухоли как суммы x1, x2, x3 и x4 модель определяет вес опухоли по реакции, называемой увеличением, null → tumor_weight, со скоростью реакции * tumor_weight.
tumor_weight - общий вес опухоли, x1 - вес пролиферирующих опухолевых клеток, L0 и L1 - параметры роста опухоли.
Аналогично, модель определяет уменьшение веса опухоли по реакции под названием Распад, tumor_weight → null, со скоростью реакции k1*x4. Константа k1 является параметром прямой скорости, а x4 является последним видом в серии транзитных сокращений веса опухоли.
ke - функция зазора и объема центрального отсека: ke = Cl_Central/Central.
Анализ чувствительности позволяет определить, какие константы скорости и концентрации в модели оказывают значительное влияние на общее поведение модели. В этом примере показано, как исследовать, какие параметры модели чувствительны к росту опухоли с помощью встроенной программы анализа чувствительности. В частности, в этом примере показано, как вычислить чувствительность видов tumor_weight к параметрам модели следующим образом:
∂ (k1),...
Предположим, что вы уже откалибровали параметры модели роста опухоли к экспериментальным данным PK/PD и сохранили оценки параметров и начальные условия как вариант, называемый parameterEstimates в вашей модели. В качестве последующего анализа необходимо выяснить, какие параметры модели чувствительны к росту опухоли. Следующие шаги показывают, как это сделать с помощью встроенной программы анализа чувствительности.
В командной строке MATLAB ® загрузите модель (m1), введя :
sbioloadproject tumor_growth_vpop_sa.sbprojОткройте модель в приложении SimBiology Model Analyzer, введя:
simBiologyModelAnalyzer(m1)
Можно также открыть приложение, щелкнув Анализатор модели SimBiology на вкладке Приложения. Затем загрузите модель из приложения, выбрав Модель > Import Model from MATLAB.
На вкладке Главная выберите Программа > Вычислить чувствительность. Откроется новая программа.
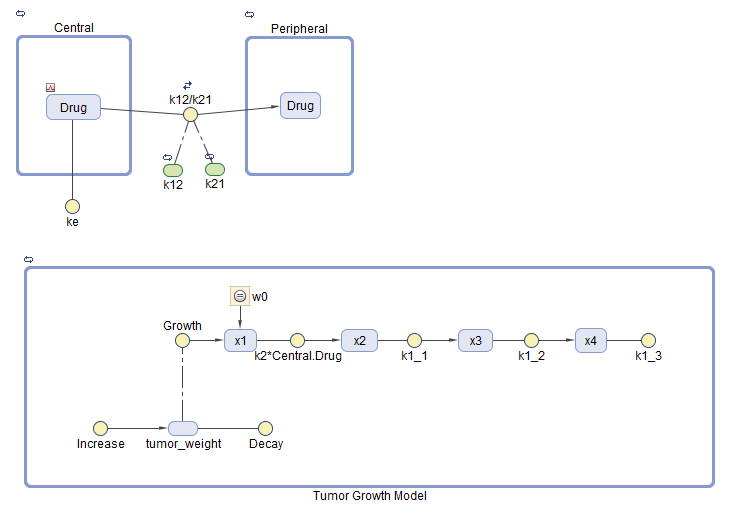
В разделе Исполнения (Variants) шага Модель (Model) (шаг настройки) программы выберите parameterEstimates. Этот вариант содержит предварительно оцененные значения параметров и исходные условия для препарата.
В разделе Дозы выберите interval_dose. Эта повторная доза составляет 30 мг препарата каждые 4 дня, начиная с 7 дня, в общей сложности пять раз.
В разделе Состояния для регистрации выберите [Модель роста опухоли] .tumor _ weight only.
Совет
Контекстные меню таблиц Исполнения (Variants), Дозы (Doses) и Состояния в журнал (States To Log) содержат опции для добавления соответствующих компонентов модели и отображения сведений об этих компонентах в приложении Построитель моделей (Model Builder).
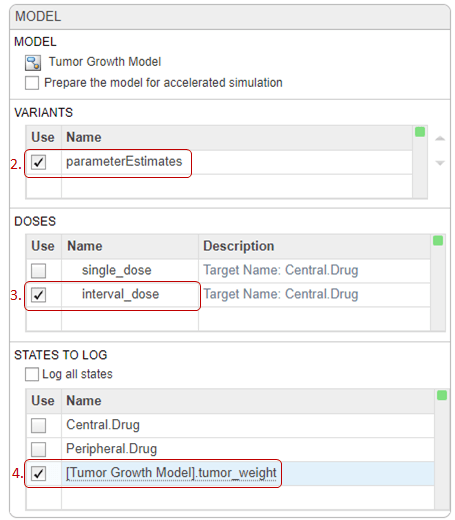
На этапе моделирования (этап выполнения) по умолчанию метод нормализации имеет значение Full (полная дедименсионализация), что означает, что приложение полностью нормализует чувствительность, чтобы их можно было сравнить друг с другом. Для получения более подробной информации см. Normalization.
Совет
Каждый шаг выполнения программы можно выполнить отдельно. Шаг выполнения включает в себя кнопку запуска рядом с именем шага. Выполнение отдельного шага особенно полезно, если программа содержит несколько шагов и требуется просмотреть промежуточные результаты определенного шага. Таким образом, можно внести необходимые изменения перед выполнением следующего шага или всей программы. Чтобы запустить всю программу, нажмите кнопку Выполнить на вкладке Главная.
В разделе Чувствительность к вычислению (Sensitivities to Compute) щелкните правой кнопкой мыши в любом месте таблицы и выберите Добавить все постоянные параметры (Add All Constant Parameters). Очистить ke, потому что он определен как функция Central том и Cl_Central, которые уже являются входными данными. Щелкните последнюю пустую строку и введите tumor. Затем выберите [Tumor Growth Model].tumor_weight из списка. Затем в качестве выхода добавляют tumor_weight разновидности.
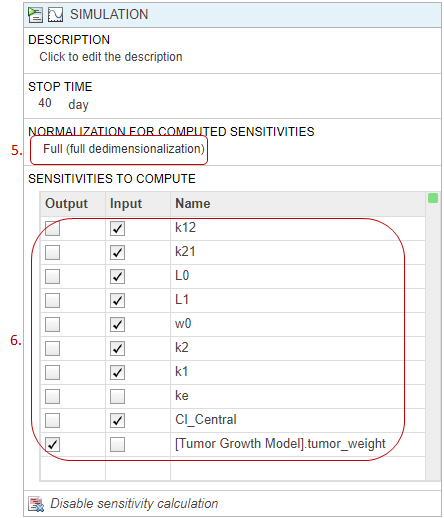
Перед моделированием модели можно просмотреть начальные условия величин модели, а именно отделения, виды и параметры. Начальными условиями являются значения количества в момент моделирования = 0 после применения правил присвоения, вариантов и доз (если таковые имеются в момент времени = 0). Дополнительные сведения см. в разделе Моделирование модели. Эту информацию можно использовать для отладки модели и проверки инициализации значений количества в соответствии с ожиданиями перед моделированием модели.
На панели «Обозреватель» нажмите «Модель».
Щелкните правой кнопкой мыши в любом месте таблицы Виды и отсеки или таблицы Параметры. Выберите Показать исходные условия модели (Show Model Initial Conditions).
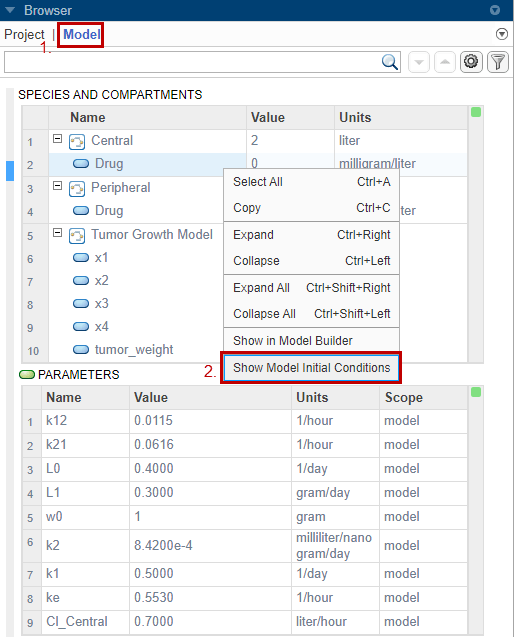
Приложение показывает столбец Начальное условие (Initial Condition) в обеих таблицах количества. Эти значения используются программой в момент времени = 0 при моделировании.
Затем запустите программу, щелкнув Выполнить (Run) на вкладке Главная страница (Home).
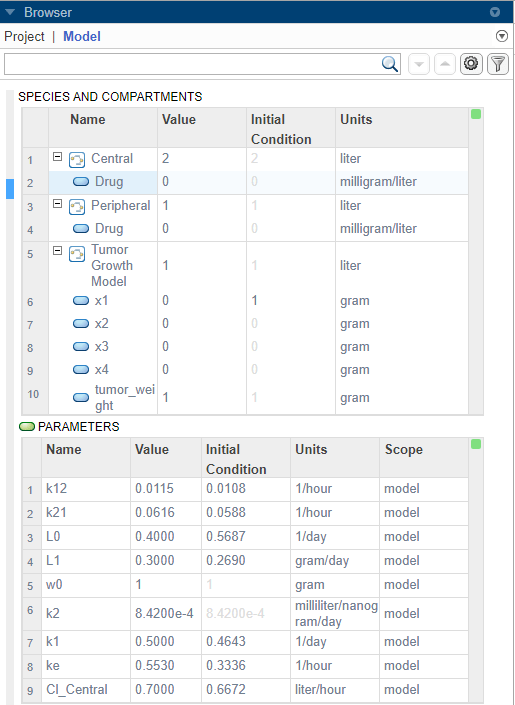
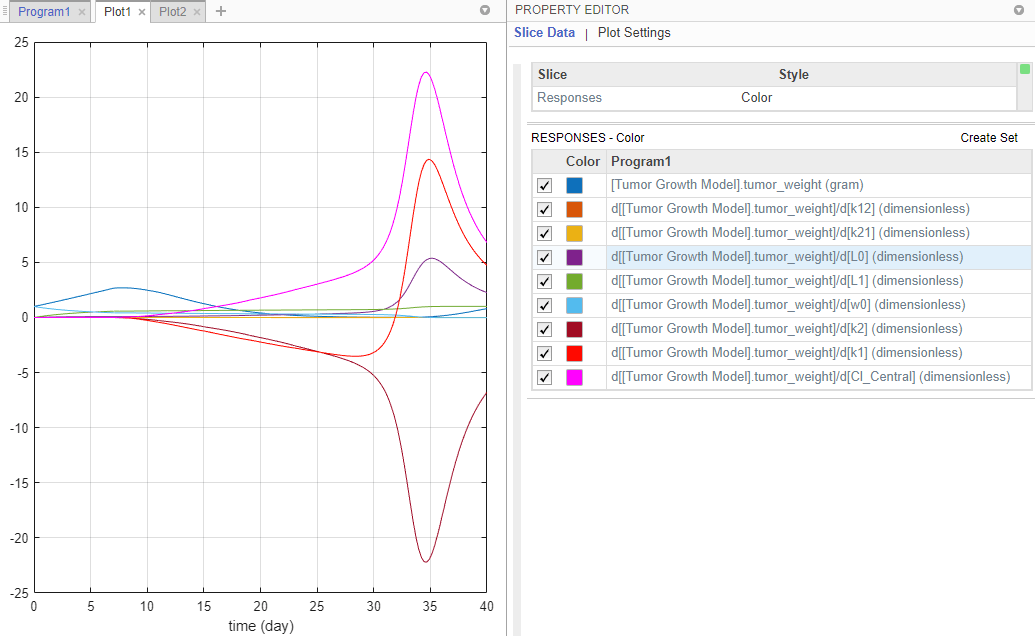
После анализа чувствительности программа автоматически создает два графика. Перейдите на вкладку «Plot1» для просмотра графика временного течения веса опухоли и других значений чувствительности. d[tumor_weight]/d[parameter]. Графики времени для d[tumor_weight]/d[k21] и d[tumor_weight]/d[k12] оставаться относительно плоским по сравнению с другими чувствительностью, что указывает на то, что рост опухоли не чувствителен к параметрам k21 и k12. Редактор свойств позволяет выбрать, какие графики следует отображать.
Совет
Графики подкрепляются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками. Когда данные (экспериментальные данные или результаты моделирования) удаляются или изменяются, графики также обновляются в соответствии с изменениями в базовых данных.
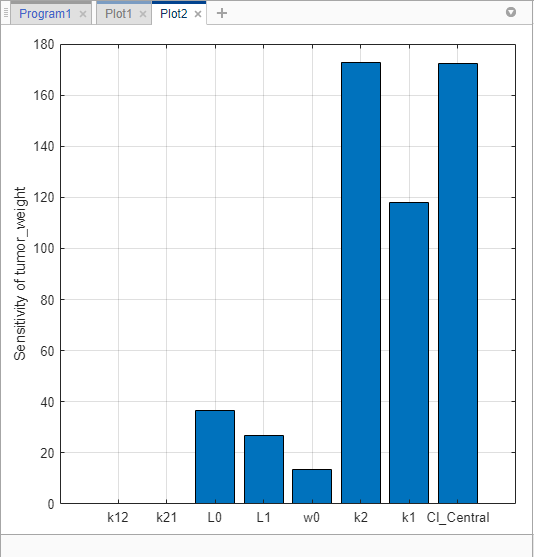
Можно также исследовать те же самые данные чувствительности, проверяя величину вычисленных чувствительности, интегрированных во времени. Второй сгенерированный график (Plot2) показывает штрих-график, содержащий такую информацию. График показывает, что вес опухоли чувствителен к L0, L1, w0, k2, k1 и Cl_Central, но не к k12 и k21.
Сохраните результаты анализа чувствительности в отдельной папке.
Нажмите «Проект» на панели «Обозреватель». Разверните папку Program1.
Щелкните правой кнопкой мыши папку LastRun. Выбрать Save Data.
В диалоговом окне «Сохранение данных» введите sa_data в качестве имени данных.
Результаты анализа чувствительности показывают, что вес опухоли не чувствителен к модельным значениям k12 и k21. Узнайте, приводит ли изменение значений этих параметров к различным результатам чувствительности.
Вары к12. Сначала измените значения параметра k12.
Перейдите на вкладку «Program1». Щелкните значок (+) плюс в верхней части программы и выберите Generate Samples.
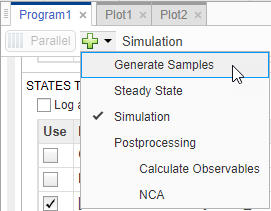
Появится шаг Создать образцы (Generate Samples). В разделе «Набор параметров» шага дважды щелкните пустую ячейку в поле «Имя компонента» и введите k12. Задайте следующие параметры:
Тип - диапазон значений
Интервал - линейный
Мин - 1е-3
Макс. - 1
Количество шагов - 25
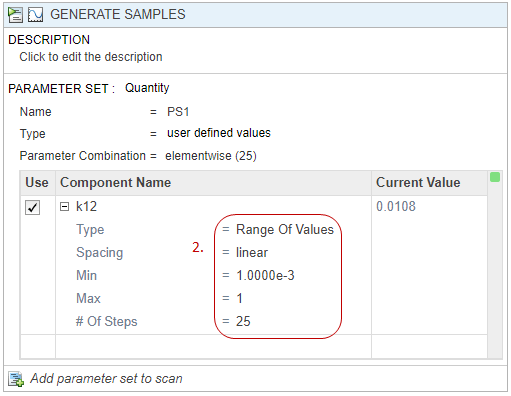
Отключите генерацию графика по умолчанию, нажав кнопку графика в верхней части шага.
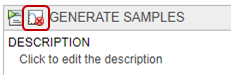
На шаге Моделирование (Simulation) в разделе Чувствительность к вычислению (Sensitivities to Compute) очистите все входные данные, кроме k12. Сохранить tumor_weight в качестве выходных данных. Кроме того, отключите генерацию графика по умолчанию, нажав кнопку графика в верхней части шага.
На вкладке Главная нажмите кнопку Выполнить, чтобы запустить всю программу.
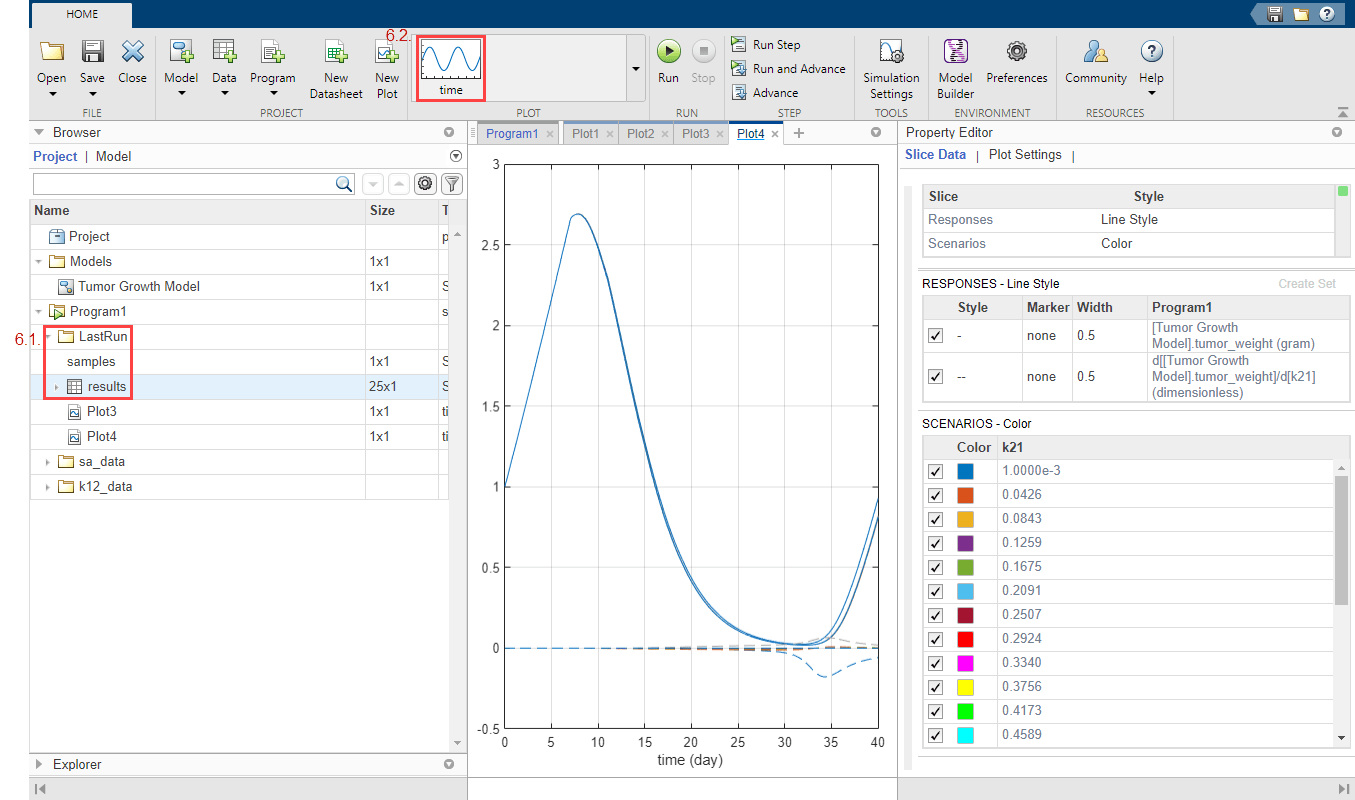
После завершения моделирования результаты чувствительности сохраняются в папке LastRun. Разверните папку LastRun. Щелкните результаты. Затем выберите время в разделе График (Plot) на вкладке Главная страница (Home).
Ответы на графике соответствуют массе опухоли и чувствительности массы опухоли по отношению к k12. Каждый ответ печатается с использованием различных стилей линий. Сценарии соответствуют каждому параметру сканирования (сценарий моделирования). Каждый сценарий строится с использованием различных цветов.
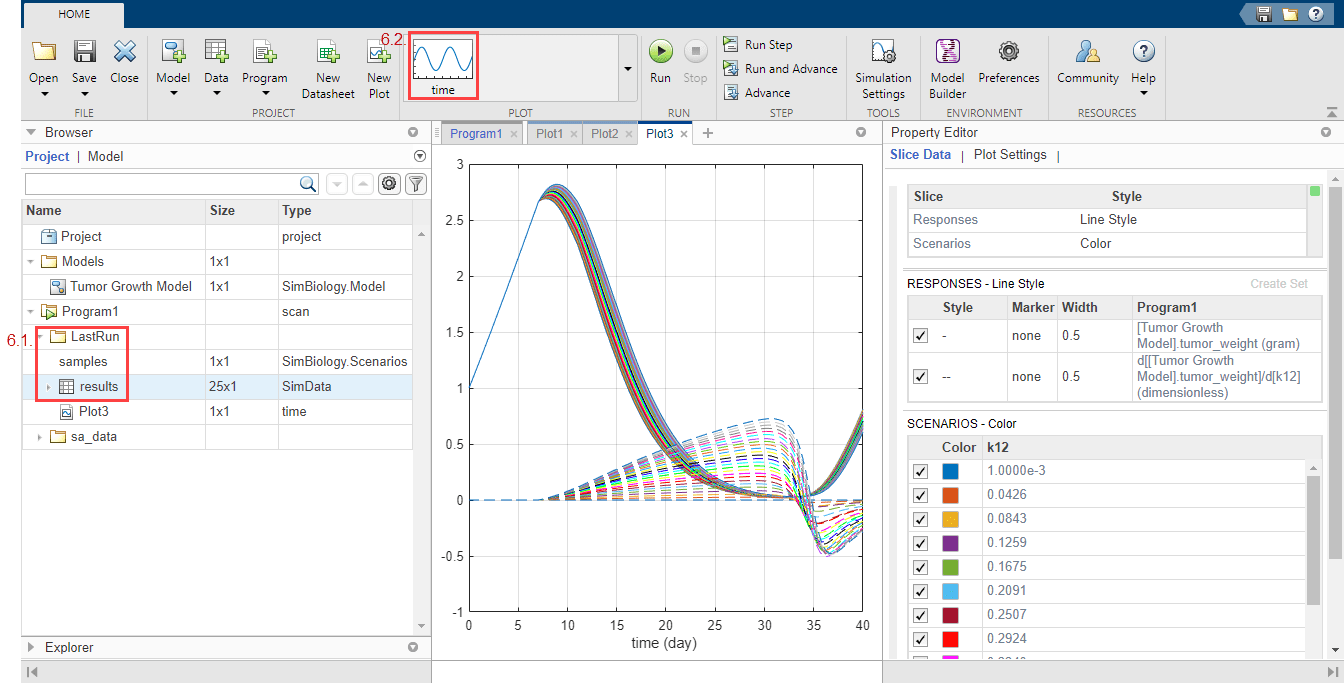
Настройте график таким образом, чтобы вместо него для каждого ответа использовался другой цвет. В таблице Данные фрагмента (Slice Data) снимите флажок Стиль сценариев (Style of Scriptions), выбрав пустой. Задать отклики на цвет.
В таблице Ответы снимите флажок tumor_weight и сохранять только результаты чувствительности.
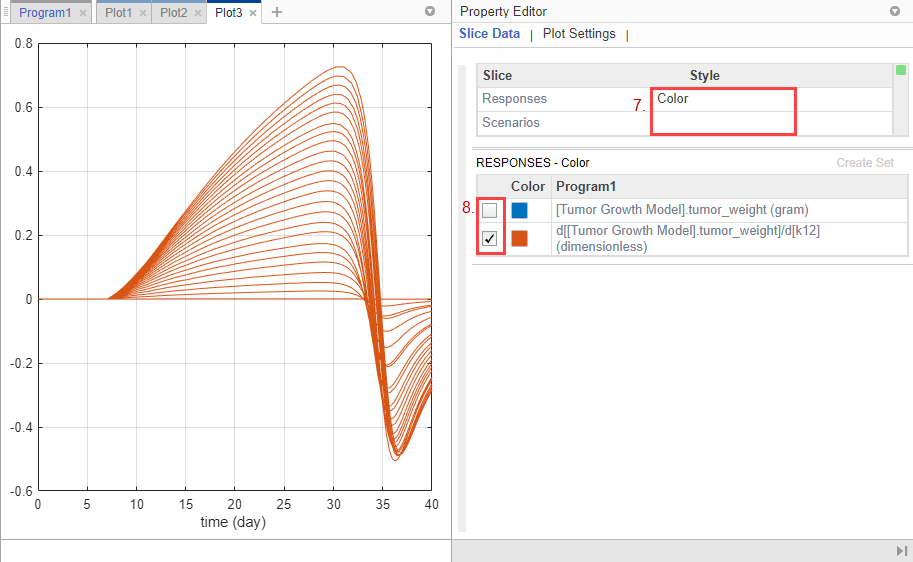
График предполагает, что вес опухоли чувствителен к некоторым значениям параметров k12, когда исследуются варианты модели, отличные от оцененных значений параметров.
Постройте график чувствительности k12 вместе с предыдущими результатами чувствительности, чтобы увидеть, насколько чувствителен рост опухоли к k12 относительно других параметров модели. На панели «Обозреватель» разверните sa_data папки, сохраненную ранее. Затем разверните результаты.
Множественный выбор (Shift + Click) записей данных чувствительности для L0, L1, w0, k2, k1 и Cl_Central. Затем перетащите их на Plot3 графика чувствительности k12.
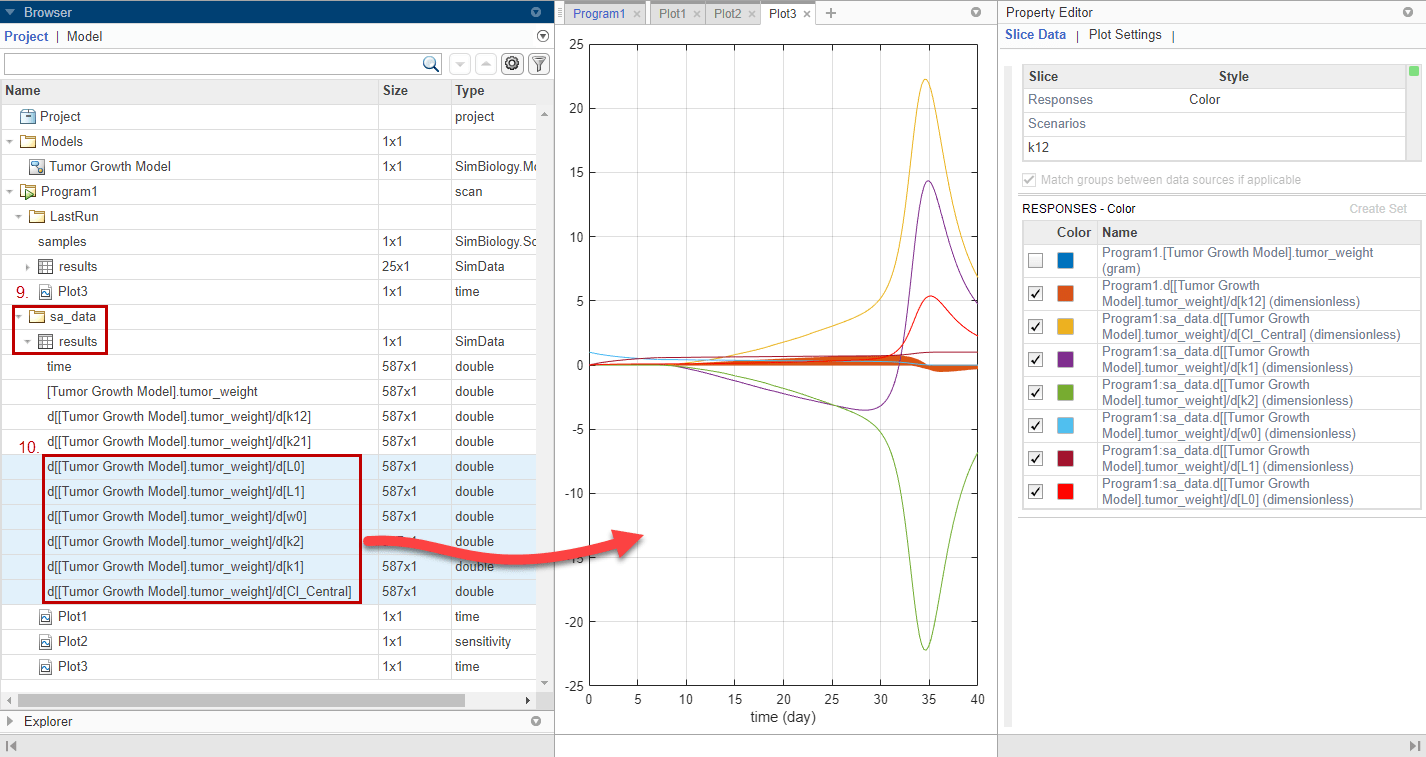
График предполагает, что рост опухоли все еще более чувствителен к большинству других параметров, чем k12.
Сохраните результаты в отдельной папке. Щелкните правой кнопкой мыши LastRun и выберите Save Data. Войти k12_data в качестве имени данных.
Вары к21. Можно выполнить аналогичный анализ, изменяя k21 и сравнивая его результаты с чувствительностью других параметров.
Вернуться к Program1. На шаге Создать образцы щелкните Добавить набор параметров для сканирования. Появится новый раздел «Набор параметров».
Дважды щелкните пустую ячейку в поле «Имя компонента» и введите k21. Установите те же параметры, что и k12:
Тип - диапазон значений
Интервал - линейный
Мин - 1е-3
Макс. - 1
Количество шагов - 25
Деактивизируйте первый набор параметров (PS1). Укажите верхний угол раздела «Набор параметров» PS1 и нажмите кнопку действия. Затем выберите Disable Parameter Set.
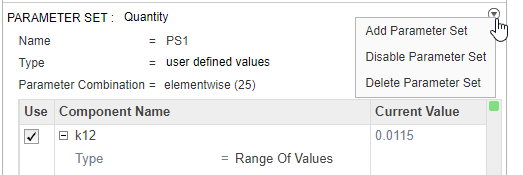
После этого набор параметров будет выделен серым цветом, что указывает на успешное отключение набора параметров.
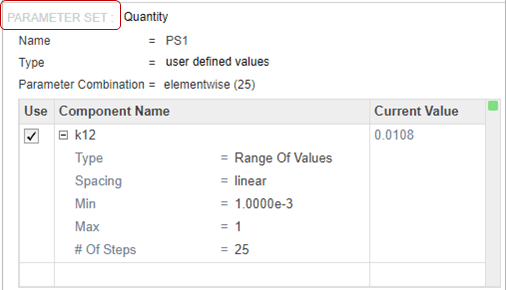
На шаге Моделирование (Simulation) выберите k21 как единственный вход и tumor_weight как выход. Отключите генерацию графика по умолчанию, нажав кнопку графика в верхней части шага.
Нажмите кнопку Выполнить на вкладке Главная, чтобы снова запустить программу.
После завершения моделирования результаты чувствительности сохраняются в папке LastRun. Разверните папку LastRun. Щелкните результаты. Затем выберите время в разделе График (Plot) на вкладке Главная страница (Home).
В таблице Данные фрагмента (Slice Data) снимите флажок Стиль сценариев (Style of Scriptions), выбрав пустой. Задать отклики на цвет.
В таблице Ответы снимите флажок tumor_weight и сохранять только результаты чувствительности.
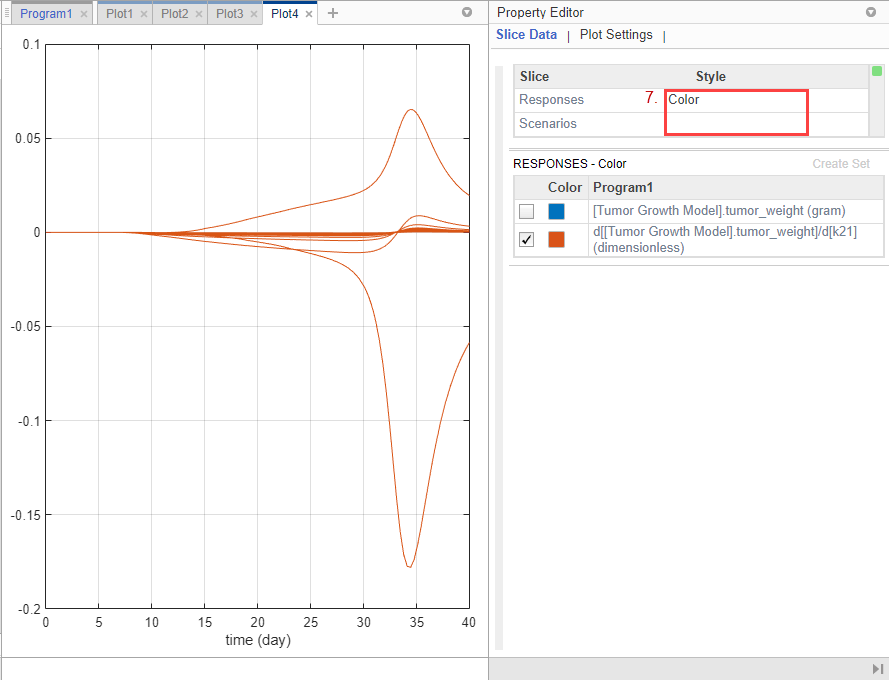
График предполагает, что вес опухоли чувствителен к некоторым значениям параметров k21, когда исследуются варианты модели, отличные от оцененных значений параметров.
Постройте график чувствительности k21 вместе с предыдущими результатами чувствительности для сравнения. Нажмите кнопку Plot3. На панели «Обозреватель» разверните папку LastRun. Затем разверните результаты.
Перетащите данные чувствительности k21 d [[Модель роста опухоли] .tumor _ weight ]/d [k21] на Plot3.
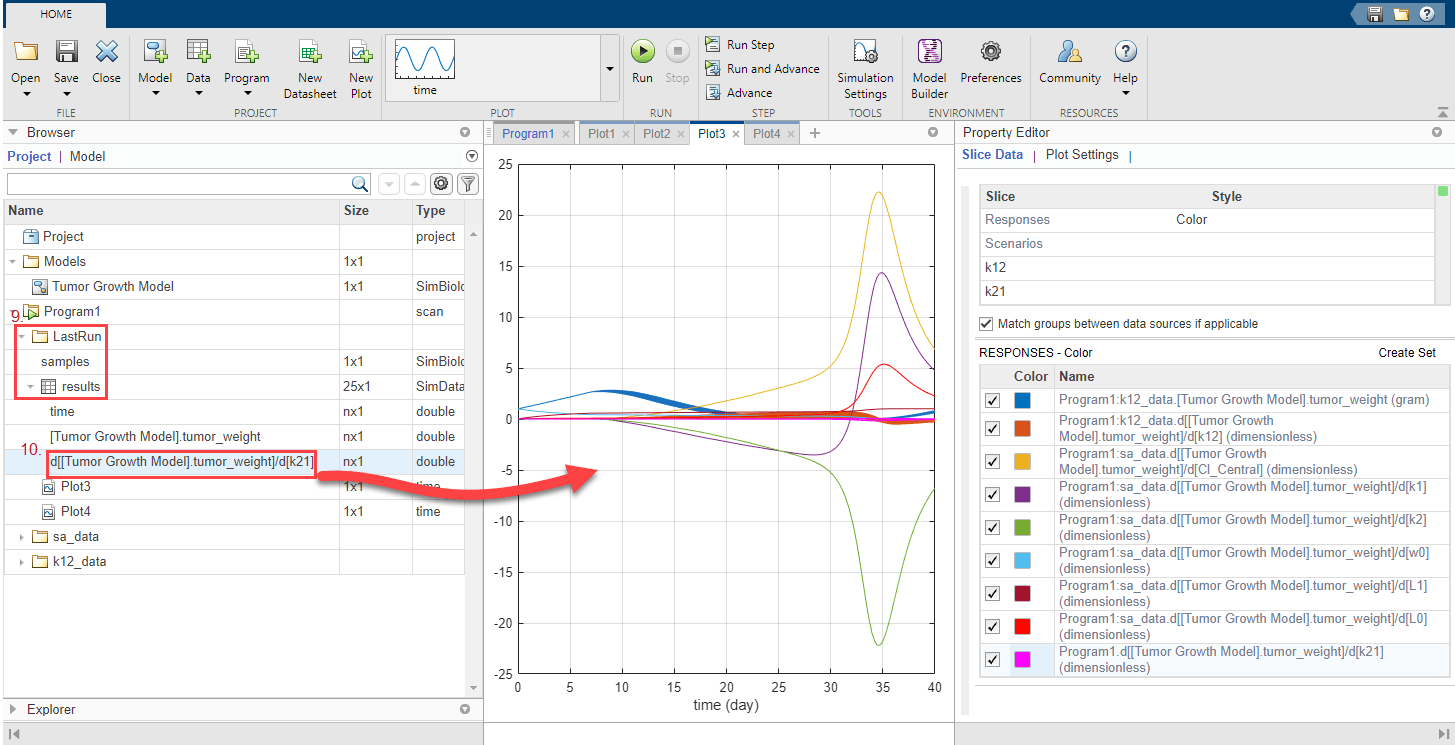
График предполагает, что рост опухоли относительно более чувствителен к большинству других параметров, чем k21 и k12.
Этот пример показывает, как выполнить анализ чувствительности, чтобы найти важные параметры модели, к которым чувствителен рост опухоли. Первоначальный анализ делает вывод, что масса опухоли не чувствительна к некоторым параметрам. Затем в примере исследуется пространство параметров этих менее чувствительных параметров и сравниваются результаты чувствительности с результатами других параметров модели. Можно использовать такую информацию о чувствительности для других анализов, например, для моделирования Монте-Карло путем изменения чувствительных параметров для изучения динамики модели и биологической изменчивости.
[1] Симеони, М., П. Магни, К. Каммия, Г. Де Николао, В. Крочи, Э. Песенти, М. Германи, И. Поггези и М. Роккетти. 2004. Прогностическое фармакокинетико-фармакодинамическое моделирование кинетики роста опухоли на моделях ксенотрансплантата после введения противораковых агентов. Исследования рака. 64:1094-1101.