В этом примере показано, как генерировать и моделировать виртуальных пациентов с помощью приложения SimBiology Model Analyzer. В этом примере виртуальный пациент представлен как единая реализация параметров модели. В примере используется модель роста опухоли [1], чтобы исследовать изменчивость некоторых параметров модели, которые влияют на рост опухоли, и исследовать различные режимы дозирования для ее контроля.
В этом примере требуется Toolbox™ статистики и машинного обучения.
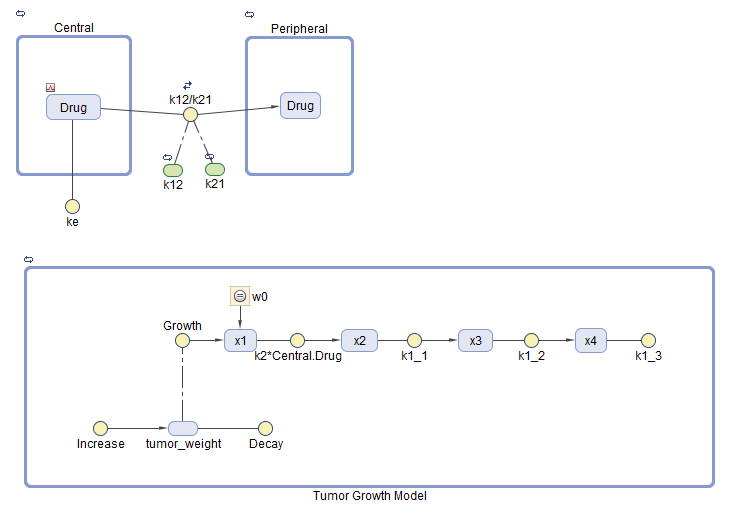
Модель, используемая в этом примере, представляет собой реализацию SimBiology ® фармакокинетической/фармакодинамической (PK/PD) модели Simeoni et al. Он количественно оценивает влияние противораковых препаратов на кинетику роста опухоли из исследований на животных in vivo. Фармакокинетика препарата описана двухкамерной моделью с внутривенным дозированием болюса и линейным элиминированием (ke) из центрального отделения. Рост опухоли представляет собой двухфазный процесс с начальным экспоненциальным ростом с последующим линейным ростом. Скорость роста пролиферирующих опухолевых клеток описана
w)
L0, L1 и Ψ - параметры роста опухоли, x1 - вес распространяющихся опухолевых клеток, и w - общий вес опухоли. При отсутствии каких-либо лекарств опухоль состоит только из пролиферирующих клеток, то есть w = x1. В присутствии противоракового агента часть пролиферирующих клеток трансформируется в непролиферирующие клетки. Предполагается, что скорость этого превращения зависит от концентрации лекарственного средства в плазме и фактора эффективности k2. Непролиферирующие клетки x2 проходят через ряд транзитных стадий (x3 и x4) и в конечном итоге удаляются из системы. Поток транзитных отсеков моделируется как процесс первого порядка с постоянной скоростью k1.
Модель SimBiology вносит эти коррективы в фармакодинамику роста опухоли:
Вместо определения веса опухоли как суммы x1, x2, x3 и x4 модель определяет вес опухоли по реакции, называемой увеличением, null → tumor_weight, со скоростью реакции * tumor_weight.
tumor_weight - общий вес опухоли, x1 - вес пролиферирующих опухолевых клеток, L0 и L1 - параметры роста опухоли.
Аналогично, модель определяет уменьшение веса опухоли по реакции под названием Распад, tumor_weight → null, со скоростью реакции k1*x4. Константа k1 является параметром прямой скорости, а x4 является последним видом в серии транзитных сокращений веса опухоли.
ke - функция зазора и объема центрального отсека: ke = Cl_Central/Central.
Виртуальная популяция в этом примере представлена виртуальными пациентами, определенными как отдельные наборы значений параметров для конкретного пациента. Предположим, что, основываясь на предыдущих знаниях или анализе чувствительности модели, рост опухоли чувствителен к этим параметрам модели: L0, L1, w0, k1, k2 и Cl_Central. Предполагая, что эти биологические параметры следуют логнормальному распределению (и всегда должны быть положительными), вы можете генерировать виртуальных пациентов, которые представляют различные значения параметров, полученные из совместного логнормального распределения.
Логнормальное распределение использует следующие параметры:
mu - Среднее логарифмических значений
sigma - стандартное отклонение логарифмических значений
Дополнительные сведения см. в разделе Lognormal Distribution (Statistics and Machine Learning Toolbox).
В этом примере предполагается, что сигма составляет 0,01 для всех параметров. Для малого значения сигма среднее логнормального распределения приблизительно равно логарифму значения модели. Следовательно, в этом примере предполагается, что mu для каждого параметра является логарифмом значения модели.
В этом примере противоопухолевый препарат используют для контроля роста опухоли. Каждый виртуальный пациент получает одинаковое количество препарата по одному и тому же графику. Реакция на рост опухоли моделируется для каждого пациента. Затем количества доз варьируются, чтобы найти диапазон количеств доз, которые могут подавлять рост опухоли многих виртуальных пациентов в популяции.
Следующие шаги показывают, как извлечь значения выборки из совместного распределения вероятности для параметров модели, чувствительных к росту опухоли, чтобы представить группу виртуальных пациентов.
В командной строке MATLAB ® загрузите модель (m1), введя :
sbioloadproject tumor_growth_vpop_sa.sbprojОткройте модель в приложении SimBiology Model Analyzer, введя:
simBiologyModelAnalyzer(m1)
Можно также открыть приложение, щелкнув Анализатор модели SimBiology на вкладке Приложения. Затем загрузите модель из приложения, выбрав Модель > Import Model from MATLAB.
Создайте программу для получения значений образцов из совместного логнормального распределения для параметров, чувствительных к росту опухоли: L0, L1, w0, k1, k2 и Cl_Central.
На вкладке Главная выберите Программа > Создать образцы. Откроется новая программа.
В разделе Исполнения (Variants) на шаге Модель (Model) программы щелкните опцию для просмотра применяемых исполнений. Затем выберите parameterEstimates, который содержит оценочные значения параметров.
На шаге Создать образцы (Generate Samples) нажмите кнопку графика, чтобы отключить генерацию графика по умолчанию. Вы постройте график образцов позже.
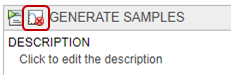
В разделе «Набор параметров» задайте для параметра «Тип» значение values from a distribution.
Задать количество образцов равным 25.
Дважды щелкните пустую ячейку в столбце «Имя компонента» и введите L0.
Изменить распределение для L0 на Lognormal. По определению, mu - среднее логарифмических значений. Итак, измените mu на -0.5644, который является log(Current Value) или log(0.5687). Измените сигму на 0,01. Для малого значения сигмы среднее логнормального распределения приблизительно равно log(Current Value). Дополнительные сведения см. в разделе Lognormal Distribution (Statistics and Machine Learning Toolbox).
Дважды щелкните пустую ячейку в столбце «Имя компонента» и добавьте L1. Повторите тот же процесс, чтобы изменить распределение на логнормальное и установить значения mu и sigma. Аналогично, добавьте w0, k1, k2 и ke. В этой таблице перечислены соответствующие значения mu этих параметров, которые можно скопировать и вставить в программное обеспечение. Измените сигму на 0,01 для каждого параметра.
Параметр | Текущее значение | mu или Log (текущее значение) |
|---|---|---|
L0 | 0.5687 | -0.5644 |
L1 | 0.2690 | -1.3130 |
w0 | 1 | 0 |
k1 | 0.4643 | -0.7672 |
k2 | 8.4200e-4 | -7.0797 |
Cl_Central | 0.6672 | -0.4047 |
Для параметра «Выборка» используйте опцию по умолчанию: random sampling with rank correlation matrix, где матрица является единичной матрицей.
На следующем рисунке показан раздел «Набор параметров» со всеми настроенными параметрами. Красные числа соответствуют предыдущим шагам.
(Необязательно) Сохраните проект под новым именем, выбрав «Сохранить» > «Сохранить проект как» на вкладке «Главная».
Добавьте информацию о дозировке в качестве другого набора параметров, указав различное количество дозы - в частности, шесть количеств дозы, которые равноудалены от 5 до 35 мг. Затем объедините дозы с первым набором параметров (виртуальные пациенты) с помощью декартова комбинированного метода. Этот метод объединяет каждого виртуального пациента с каждым количеством дозы, чтобы создать в общей сложности 150 итераций (или сценариев моделирования). Дополнительные сведения см. в разделе Объединение сценариев моделирования в SimBiology.
В нижней части шага Создать образцы щелкните Добавить набор параметров для сканирования. Появится другой набор параметров (PS2).
Дважды щелкните пустую ячейку в столбце «Имя компонента» и введите interval. Появится список вариантов. Выбрать interval_dose.Amount.
Задать тип для Individual Values и задайте для параметра «Значения» значение [5:6:35].
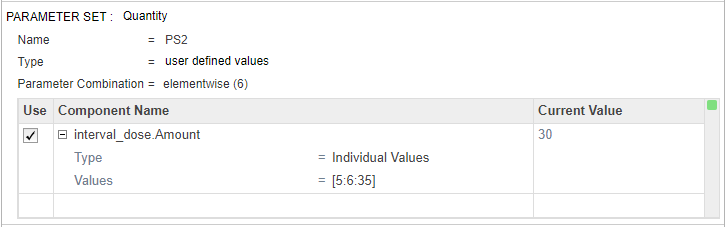
В верхней части шага Создать образцы (Generate Samples) в разделе Комбинации наборов параметров (Parameter Set Combinations) убедитесь, что для типа Комбинация (Combination) задано значение cartesian объединиться PS1 и PS2.

После определения совместного логнормального распределения для параметров модели, диапазона доз и метода комбинации можно выполнить шаг для генерации различных параметров и комбинаций доз.
Нажмите кнопку Run this program step, чтобы создать образцы.
Совет
Каждый шаг выполнения программы можно выполнить отдельно. Шаг выполнения включает в себя кнопку запуска рядом с именем шага. Выполнение отдельного шага особенно полезно, если программа содержит несколько шагов и требуется просмотреть промежуточные результаты определенного шага. Таким образом, можно внести необходимые изменения перед выполнением следующего шага или всей программы. Чтобы запустить всю программу, нажмите кнопку Выполнить на вкладке Главная.
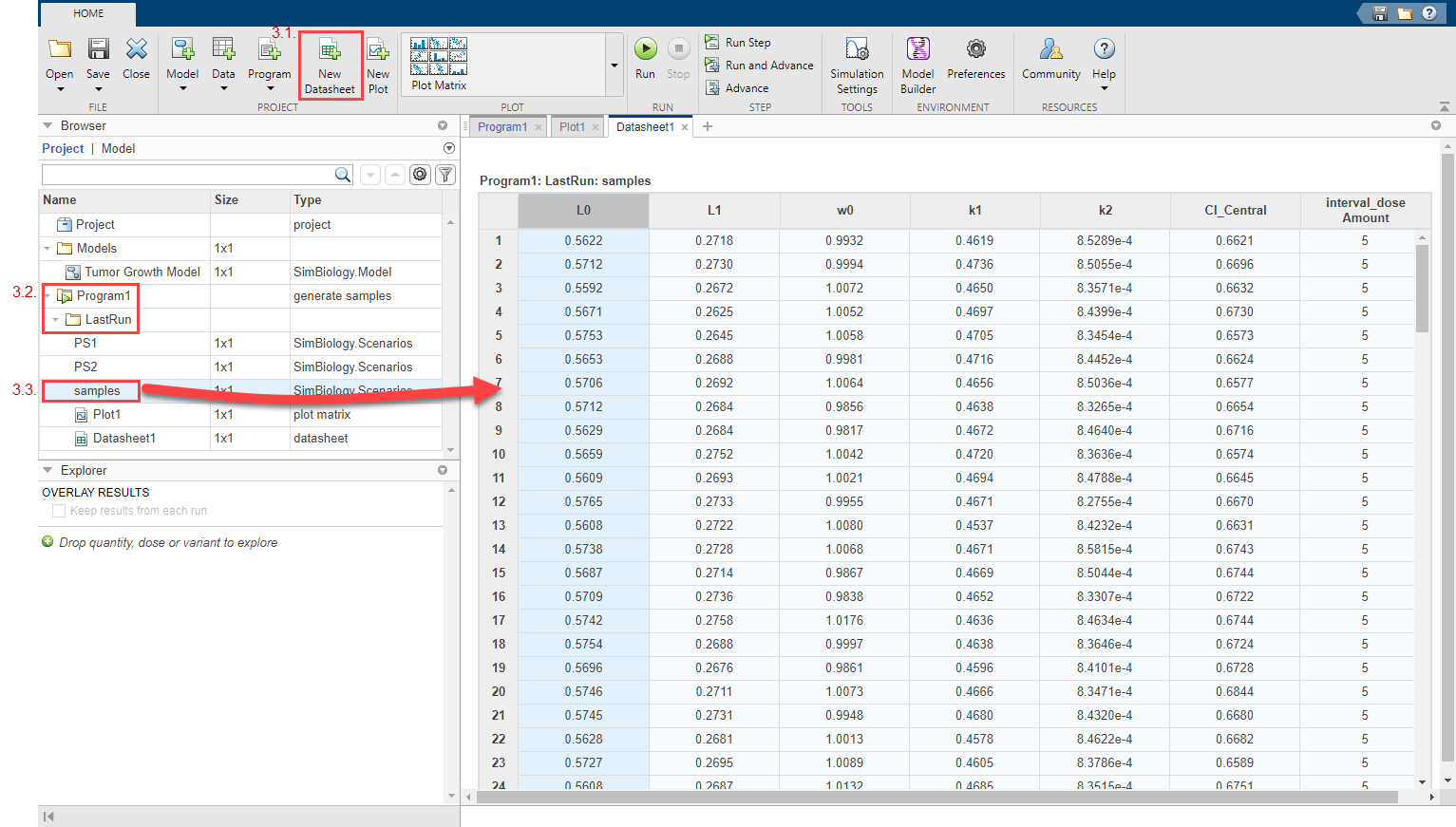
По умолчанию созданные образцы хранятся в папке LastRun программы.
Визуализация созданных образцов. В левой панели браузера разверните папку Program1. Разверните папку LastRun и щелкните образцы. В разделе Печать (Plot) на вкладке Главная (Home) щелкните Матрица печати (Plot Matrix). График показывает распределение каждого параметра, изменяющегося вокруг его модельного значения, за исключением количеств доз, которые появляются в однородном диапазоне. Обратите внимание, что график может отличаться от графика, показанного здесь, из-за случайности.
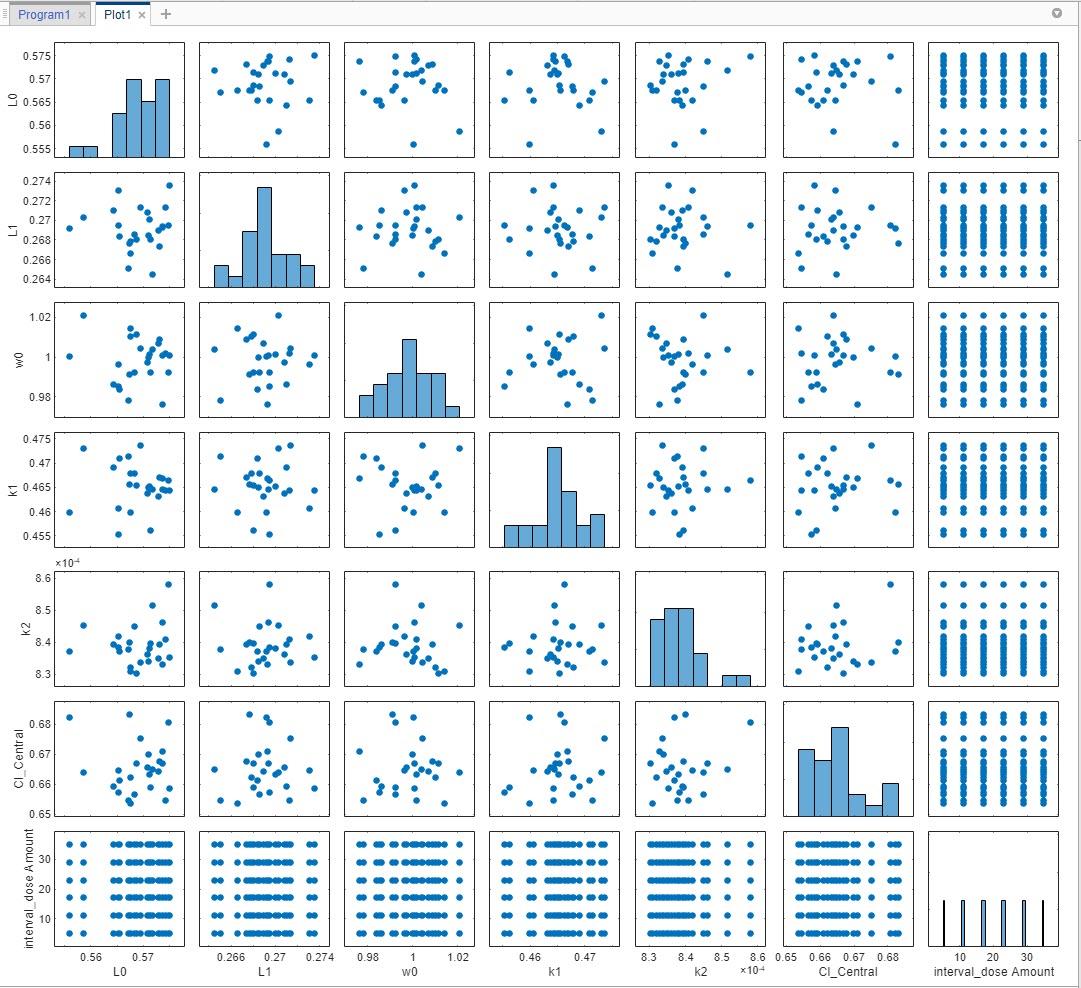
Примечание
Можно включить генерацию графика по умолчанию для автоматического отображения графика при каждом выполнении шага. Для этого нажмите кнопку графика в верхней части шага Создать образцы (Generate Samples). Имейте в виду, что если на шаге программы много образцов, построение графика всех образцов может занять много времени.
Отображение созданных виртуальных пациентов и комбинаций доз численно в табличном формате. На вкладке Главная щелкните Создать таблицу. Из папки LastRun программы перетащите образцы в новую таблицу данных. Каждая строка таблицы представляет сценарий моделирования с различными значениями параметров модели и дозами.
Как только вы подготовите образцы, вы можете смоделировать модель, чтобы исследовать рост опухоли у виртуальных пациентов, получающих разное количество дозы.
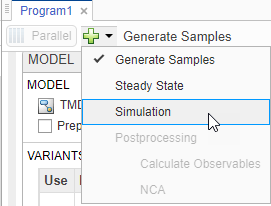
Вернитесь к программе, щелкнув вкладку «Program1».
Щелкните значок (+) в верхнем левом углу и выберите «Моделирование».
На шаге настройки модели в разделе Исполнения убедитесь, что выбран параметр parameterEstimates. В разделе Состояния для регистрации установите флажок для просмотра состояний и снимите все флажки, кроме [Модель роста опухоли] .tumor _ weight.
В верхней части шага Моделирование (прокрутите вниз, чтобы увидеть этот шаг) нажмите кнопку графика, чтобы отключить генерацию графика по умолчанию.
Нажмите кнопку Выполнить этот шаг программы (Run this program step), чтобы смоделировать модель.
Приложение моделирует созданные сценарии на шаге Создать образцы, выполненном ранее. Повторный запуск шага не требуется.
После завершения моделирования приложение сохраняет результаты моделирования в папке LastRun.
В папке LastRun на панели «Обозреватель» щелкните результаты. В разделе Печать на вкладке Главная щелкните время. Каждая линия на графике представляет профиль веса опухоли для каждого сценария моделирования.
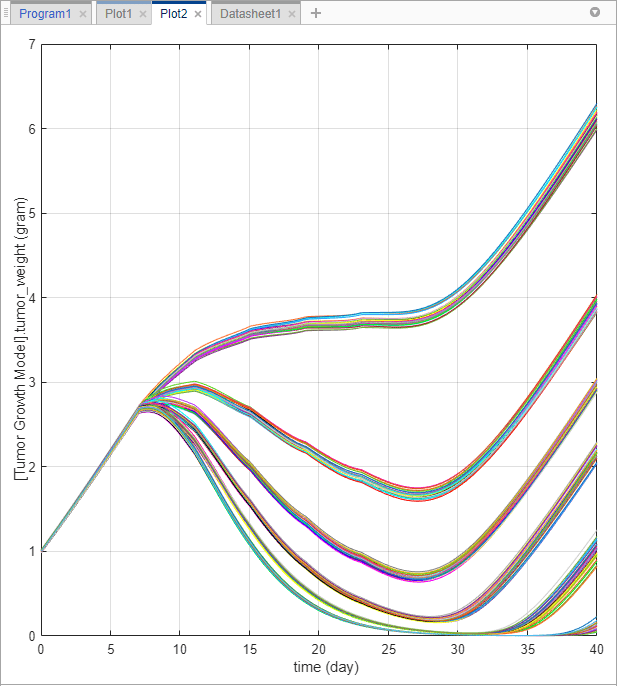
График показывает, что профили веса опухоли появляются в группах, соответствующих различным дозам. Чтобы лучше визуализировать это наблюдение, можно разрезать данные по количеству доз. Для этого можно использовать таблицу Данные среза (Slice Data), содержащую сводку переменных среза, которые в настоящее время используются при печати, и соответствующие стили печати.
Совет
Графики подкрепляются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками. Когда данные (экспериментальные данные или результаты моделирования) удаляются или изменяются, графики также обновляются в соответствии с изменениями в базовых данных.
В таблице «Данные фрагмента» дважды щелкните L0 и выбрать interval_dose Amount.
Совет
Данные среза можно разделить с помощью различных переменных среза. Каждая переменная среза отображается на графике с различным визуальным стилем (или каналом), таким как цвет, стиль линий и положение осей. Переменные среза могут представлять атрибуты данных, такие как ответы или сценарии (т.е. группы или прогоны моделирования). Разделяющие переменные также могут быть ковариатами или значениями параметров, связанными со сценарием или группой. По умолчанию приложение предоставляет разделенные переменные для различных переменных ответа и различных сценариев в построенных данных. Можно добавить другие визуальные стили (или каналы) для наборов ответов и связанных переменных параметров или ковариат.
Дважды щелкните пустую ячейку в столбце «Стиль» и выберите Grid.
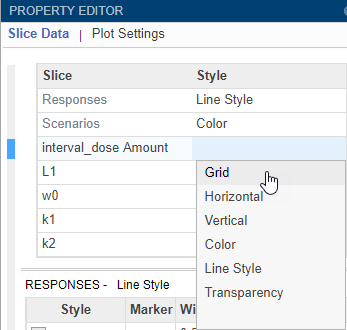
Приложение обновляет Plot2, чтобы показать каждое количество дозы на своих осях.
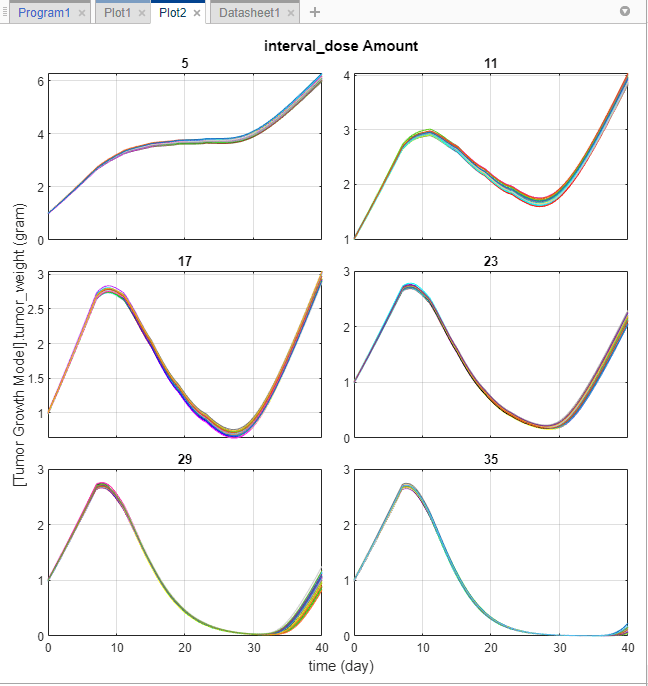
График показывает, что различные количества доз играют критическую роль в росте опухоли виртуальных пациентов. На этом графике можно получить некоторое начальное представление об оптимальных дозах и планировании доз. Например, предположим, что целевой вес опухоли составляет 0,5 грамма. Результаты моделирования показывают, что количество дозы в 23 миллиграмма или больше может достичь этой цели (можно дополнительно отрегулировать диапазон количеств дозы, настроив его на шаге «Генерировать образцы» программы). Вы можете использовать эту информацию в сочетании с существующей информацией о токсичности лекарств (не обсуждается в этом примере), чтобы получить режим дозирования, который удовлетворяет как требованиям эффективности, так и безопасности, например.
Вы также можете повторно обработать результаты моделирования, чтобы посмотреть на корреляцию между количеством дозы и весом опухоли другим способом.
Вернитесь к программе, щелкнув вкладку «Program1». Щелкните значок (+) в верхней части программы и выберите Рассчитать наблюдаемые в разделе Постобработка.
Шаг Постобработка: Расчет наблюдаемых (Postprocessing: Calculate Observables) отображается под шагом Моделирование (Simul
Дважды щелкните первую ячейку в столбце «Имя». Войти stat1.
В столбце «Выражение» введите следующее выражение: min(vertcat(nan,tumor_weight(time>=7))). Экспрессия возвращает минимальный вес опухоли после первой дозы, применяемой на 7 день, из каждого моделирования.
Примечание
При каждом добавлении выражения в таблицу Наблюдаемые (Observables) на шаге выражение автоматически добавляется как observable объект к соответствующей модели.
В столбце «Единицы» введите gram.
Повторно запустите этап моделирования, нажав кнопку запуска в верхней части шага. Приложение моделирует и оценивает stat1 выражение для каждого сценария моделирования или итерации и создает следующий график. Ось x представляет параметры, а ось y представляет минимальный вес опухоли. Каждая точка представляет сценарий моделирования. График также показывает, что нет корреляции между размером опухоли и значениями различных параметров модели, за исключением количеств дозы (самый правый подграф).
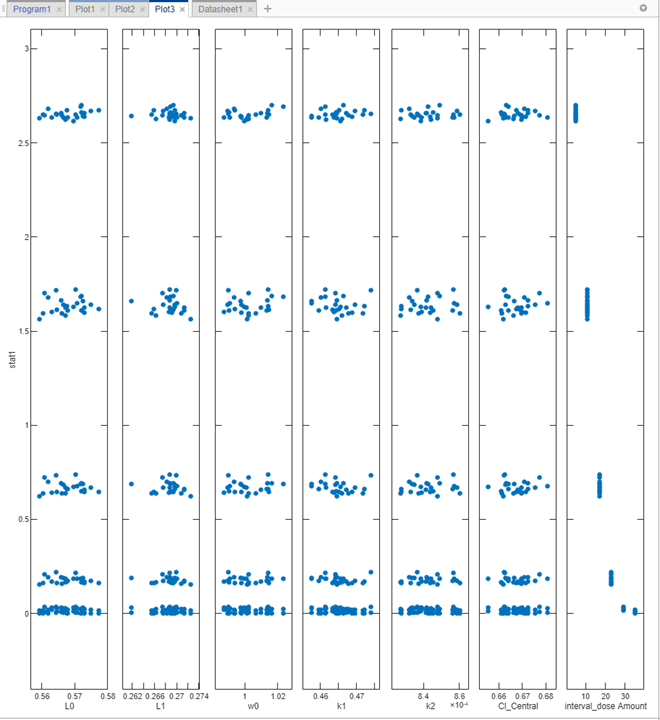
Показать только субплот количества дозы. В окне «Параметры печати» снимите все флажки в таблице «Параметры», кроме interval_dose Amount.
В разделе «Сетка» выберите оба параметра, чтобы показать сетки для осей X и Y.
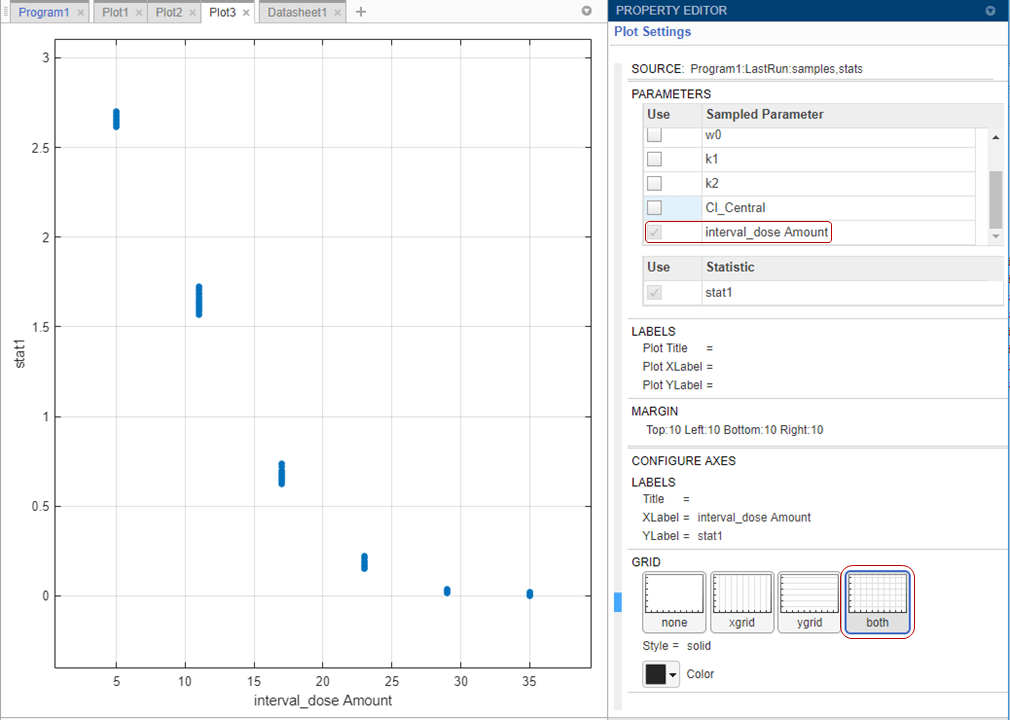
График подтверждает, что более высокие количества доз лучше контролируют рост опухоли. Эти первоначальные результаты моделирования показывают, что количества дозы 23 мг или более могут достигать гипотетического порога массы опухоли 0,5 г или ниже. На шаге Генерировать образцы (Generate Samples) можно дополнительно скорректировать диапазон доз.
Совет
Для интерактивного просмотра данных печати экспортируйте график в отдельное окно рисунка, выбрав команду «Экспорт графика» в контекстном меню графика.
В этом примере показано, как генерировать образцы для представления виртуальных пациентов и выполнять моделирование Монте-Карло для изучения реакции модели на рост опухоли при различных дозах. Результаты моделирования указывают диапазон количеств доз и графиков доз, которые контролируют рост опухоли для различных виртуальных пациентов. Вы можете дополнительно скорректировать схемы дозирования, чтобы дозы оставались в пределах некоторых известных порогов эффективности и токсичности. Аналогичный анализ см. в примере Схемы дозирования с использованием приложения SimBiology Model Analyzer.
[1] Симеони, М., П. Магни, К. Каммия, Г. Де Николао, В. Крочи, Э. Песенти, М. Германи, И. Поггези и М. Роккетти. 2004. Прогностическое фармакокинетико-фармакодинамическое моделирование кинетики роста опухоли на моделях ксенотрансплантата после введения противораковых агентов. Исследования рака. 64:1094-1101.
Observable | Анализатор модели SimBiology