Агломерирующее иерархическое дерево кластера
Вычисления linkage(y) может быть медленным, когда y - векторное представление матрицы расстояний. Для 'centroid', 'median', и 'ward' методы, linkage проверяет, y - евклидово расстояние. Избегайте этой трудоемкой проверки, проходя X вместо y.
'centroid' и 'median' методы могут создавать дерево кластера, которое не является монотонным. Этот результат возникает, когда расстояние от объединения двух кластеров, r и s, до третьего кластера меньше, чем расстояние между r и s. В этом случае в дендрограмме, нарисованной с ориентацией по умолчанию, путь от листа к корневому узлу делает некоторые шаги вниз. Чтобы избежать этого результата, используйте другой метод. На этом рисунке показано немонотонное дерево кластера.
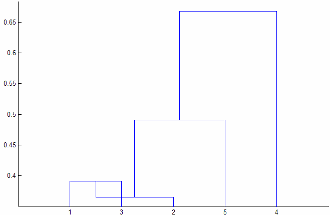
В этом случае кластер 1 и кластер 3 объединяются в новый кластер, и расстояние между этим новым кластером и кластером 2 меньше, чем расстояние между кластером 1 и кластером 3. В результате получается немонотонное дерево.
Можно предоставить выходные данные Z к другим функциям, включая dendrogram для отображения дерева, cluster назначение точек кластерам, inconsistent для вычисления противоречивых показателей, и cophenet вычисляют коэффициент кофенетической корреляции.
cluster | clusterdata | cophenet | dendrogram | inconsistent | kmeans | pdist | silhouette | squareform