В этом примере показано, как выполнять продольный анализ с использованием mvregress.
Загрузите образец продольных данных.
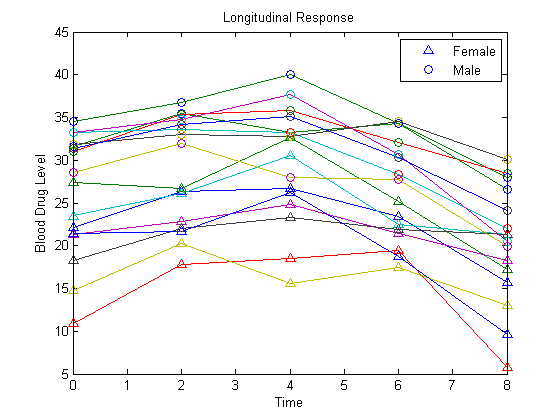
load longitudinalDataМатрица Y содержит данные ответа для 16 человек. Ответ представляет собой уровень лекарственного средства в крови, измеренный в пять временных точек (t = 0, 2, 4, 6 и 8). Каждая строка Y соответствует отдельному объекту, и каждый столбец соответствует временному моменту. Первые восемь субъектов - женщины, а вторые восемь субъектов - мужчины. Это смоделированные данные.
Постройте график данных по всем 16 предметам.
figure() t = [0,2,4,6,8]; plot(t,Y) hold on hf = plot(t,Y(1:8,:),'^'); hm = plot(t,Y(9:16,:),'o'); legend([hf(1),hm(1)],'Female','Male','Location','NorthEast') title('Longitudinal Response') ylabel('Blood Drug Level') xlabel('Time') hold off
Пусть yij обозначает ответ для отдельного i = 1,..., n, измеренный в моменты времени tij, j = 1,..., d. В этом примере n = 16 и d = 5. Пусть Gi обозначает пол отдельного i, где Gi = 1 для мужчин и 0 для женщин.
Рассмотрите возможность установки квадратной продольной модели с отдельным наклоном и пересечением для каждого пола.
β5Gi × tij2 + αij,
где ∼MVN (0, Λ). Корреляция ошибок учитывает кластеризацию внутри отдельного пользователя.
Подгонка этой модели с помощью mvregressданные ответа должны быть в n-за-d матрице. Y уже находится в правильном формате.
Затем создайте массив ячеек длиной n матриц конструкции d-by-K. Для этой модели существует K = 6 параметров.
Для отдельных i матрица проектирования 5 на 6
),
соответствующий вектору параметров
Матрица X1 имеет матрицу дизайна для самки, и X2 имеет матрицу дизайна для самца.
Создание массива ячеек матриц проектирования. Первые восемь особей - самки, а вторые восемь - самцы.
X = cell(8,1);
X(1:8) = {X1};
X(9:16) = {X2};
Подгоните модель, используя максимальную оценку правдоподобия. Отображение расчетных коэффициентов и стандартных ошибок.
[b,sig,E,V,loglikF] = mvregress(X,Y); [b sqrt(diag(V))]
ans =
18.8619 0.7432
13.0942 1.0511
2.5968 0.2845
-0.3771 0.0398
-0.5929 0.4023
0.0290 0.0563Коэффициенты на условиях взаимодействия (в последних двух строках b) не кажутся значимыми. Для этой подгонки можно использовать значение целевой функции loglikeability, loglikF, чтобы сравнить эту модель с моделью без условий взаимодействия, используя тест отношения правдоподобия.
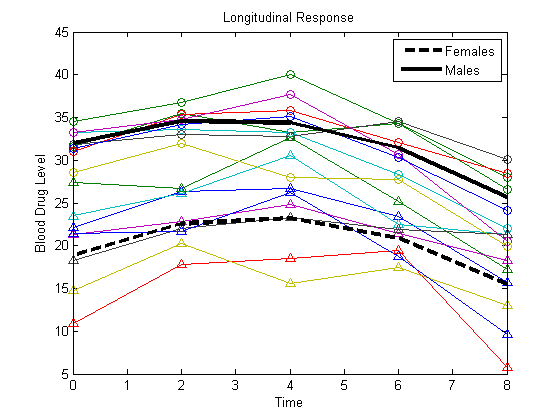
Постройте подгоняемые линии для самок и самцов.
Yhatf = X1*b; Yhatm = X2*b; figure() plot(t,Y) hold on plot(t,Y(1:8,:),'^',t,Y(9:16,:),'o') hf = plot(t,Yhatf,'k--','LineWidth',3); hm = plot(t,Yhatm,'k','LineWidth',3); legend([hf,hm],'Females','Males','Location','NorthEast') title('Longitudinal Response') ylabel('Blood Drug Level') xlabel('Time') hold off
Подогнать модель без условий взаимодействия,
β3tij2 + αij,
где ∼MVN (0, Λ).
Эта модель имеет четыре коэффициента, которые соответствуют первым четырем столбцам матриц проектирования X1 и X2 (для женщин и мужчин соответственно).
X1R = X1(:,1:4);
X2R = X2(:,1:4);
XR = cell(8,1);
XR(1:8) = {X1R};
XR(9:16) = {X2R};
Подгоните эту модель, используя максимальную оценку правдоподобия. Отображение расчетных коэффициентов и их стандартных ошибок.
[bR,sigR,ER,VR,loglikR] = mvregress(XR,Y); [bR,sqrt(diag(VR))]
ans =
19.3765 0.6898
12.0936 0.8591
2.2919 0.2139
-0.3623 0.0283Сравните две модели, используя тест отношения правдоподобия. Нулевая гипотеза заключается в том, что уменьшенная модель достаточна. Альтернатива заключается в том, что уменьшенная модель является неадекватной (по сравнению с полной моделью с терминами взаимодействия).
Статистика теста отношения правдоподобия сравнивается с хи-квадратным распределением с двумя степенями свободы (для двух понижаемых коэффициентов).
LR = 2*(loglikF-loglikR); pval = 1 - chi2cdf(LR,2)
pval =
0.08030.0803 указывает, что нулевая гипотеза не отвергается на уровне значимости 5%. Поэтому нет достаточных доказательств того, что дополнительные термины улучшают прилегание.