Вычислите количество чтений, сопоставленных с геномными функциями
T = featurecount(GTFfile,Inputfile)Inputfile которые сопоставляются с геномными функциями, заданными в GTF-форматированном файле GTFfile. GTFfile задает файл аннотации. Inputfile задает имена учитываемых файлов BAM или SAM. Область выхода T - таблица, в которой строки соответствуют функциям, а столбцы - файлам входа. Элементы таблицы состоят из количества чтений, сопоставленных с каждой функцией для данного входного файла.
[___] = featurecount(___, использует дополнительные опции, заданные одним или несколькими Name,Value)Name,Value аргументы в виде пар.
Счетчик считывает данные из выборки файла SAM, который сопоставлен с функциями, включенными в файл GTF. По умолчанию featurecount отображает показания на экзоны и суммирует общее количество показаний на уровне генов.
[t,s] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam');
Processing GTF file Dmel_BDGP5_nohc.gtf ... Processing SAM file rnaseq_sample1.sam ... Processing reference chr2L ... Processing reference chr2R ... Processing reference chr3L ... Processing reference chr3R ... Processing reference chr4 ... Processing reference chrX ... Done.
Отображение первых 10 строк подсчета данных.
t(1:10,:)
ans=10×3 table
ID Reference rnaseq_sample1
_______________ _________ ______________
{'FBgn0002121'} {'chr2L'} 9
{'FBgn0067779'} {'chr2L'} 2
{'FBgn0005278'} {'chr2L'} 4
{'FBgn0031220'} {'chr2L'} 4
{'FBgn0025683'} {'chr2L'} 13
{'FBgn0053635'} {'chr2L'} 2
{'FBgn0016977'} {'chr2L'} 22
{'FBgn0086902'} {'chr2L'} 27
{'FBgn0031245'} {'chr2L'} 2
{'FBgn0024352'} {'chr2L'} 2
The ID столбец содержит имена функций (гены в этом примере). The Reference в столбце перечислены имена ссылочных последовательностей для функций. Третий столбец содержит общее количество чтений, сопоставленных с каждой функцией для данного файла SAM, то есть rnaseq_sample1.sam. По умолчанию в таблице показаны только те функции (строки) и файлы SAM (столбцы) с ненулевым количеством чтений. Установите значение true 'ShowZeroCounts', чтобы включить эти строки и столбцы со всеми счетчиками нуля в таблицу выхода.
s содержит сводную статистику назначенных и неназначенных чтений из каждого файла SAM. Для образца, TotalEntries строка указывает общее количество записей выравнивания из данного файла SAM и Assigned строка включает количество чтений, назначенных функциям в файле GTF. Для получения дополнительной информации о каждой строке см. раздел «Выходные аргументы» страницы с описанием.
s
s=9×1 table
rnaseq_sample1
______________
TotalEntries 33354
Assigned 16399
Unassigned_ambiguous 167
Unassigned_filtered 0
Unassigned_lowMappingQuality 0
Unassigned_multiMapped 0
Unassigned_noFeature 16788
Unassigned_supplementary 0
Unassigned_unmapped 0
Считывание счетчиков без суммирования и отключение отображения сообщений о прогрессе.
[t2,s2] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'Summarization',false,'Verbose',false);
Обратите внимание, что в столбце идентификатора таблицы выхода теперь отображается атрибут функции, за которым следуют начальное и стоповое положения каждой функции, разделенные подчеркиванием.
t2(1:10,:)
ans=10×3 table
ID Reference rnaseq_sample1
_____________________________ _________ ______________
{'FBgn0002121_12286_12928' } {'chr2L'} 3
{'FBgn0002121_13683_14874' } {'chr2L'} 1
{'FBgn0002121_14933_15711' } {'chr2L'} 3
{'FBgn0067779_67044_67507' } {'chr2L'} 2
{'FBgn0005278_108588_108809'} {'chr2L'} 1
{'FBgn0005278_110755_110877'} {'chr2L'} 1
{'FBgn0005278_112690_113369'} {'chr2L'} 1
{'FBgn0031220_117079_117759'} {'chr2L'} 2
{'FBgn0031220_118361_118874'} {'chr2L'} 1
{'FBgn0031220_118931_119076'} {'chr2L'} 1
Можно выбрать, как назначить чтение конкретному элементу, когда чтение перекрывается с несколькими функциями, установив опцию 'OverlapMethod'. Например, если вы хотите считать только чтение, если оно полностью перекрывает функцию, используйте опцию 'full'.
[tFull, sFull] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'OverlapMethod','full','Verbose',false);
Если у вас есть парные данные, можно считать чтения как фрагменты.
[tFrag,sFrag] = featurecount('Dmel_BDGP5_nohc.gtf','rnaseq_sample1.sam', ... 'CountFragments',true,'Verbose',false);
Можно также подсчитать фрагменты из нескольких файлов SAM.
[t2,s2] = featurecount('Dmel_BDGP5_nohc.gtf',... {'rnaseq_sample1.sam','rnaseq_sample2.sam'},'CountFragments',true, ... 'Verbose',false);
Используйте следующие опции для подсчета показаний в парном конце, где, по крайней мере, один из сопутствующих чтений выше определенного порога качества отображения.
[t3,s3] = featurecount('Dmel_BDGP5_nohc.gtf',... 'rnaseq_sample1.sam','CountFragments',true,'MinMappingQuality',20, ... 'Verbose',false);
Если показания получены из любого специфического для цепи анализа, можно задать такую специфичность цепи во время подсчета. Для образца, если протокол заблокирован, цепь функции сравнивается со строкой чтения. Затем подсчитываются только те чтения, которые имеют ту же строку, что и перекрываемую функцию.
[t4,s4] = featurecount('Dmel_BDGP5_nohc.gtf',... 'rnaseq_sample1.sam','StrandSpecificity','stranded','Verbose',false);
GTFfile - имя файла в формате GTFGTF-форматированное имя файла, заданное как вектор символов или строка.
Пример: 'Dmel_BDGP5_nohc.gtf'
Inputfile - имя файла в формате BAM или SAM;Имя файла в формате BAM или SAM, заданное как вектор символов, строка, строковый вектор или массив ячеек векторов символов.
Пример: 'rnaseq_sample1.sam'
Задайте необязательные разделенные разделенными запятой парами Name,Value аргументы. Name - имя аргумента и Value - соответствующее значение. Name должны находиться внутри кавычек. Можно задать несколько аргументов в виде пар имен и значений в любом порядке Name1,Value1,...,NameN,ValueN.
'CountFragments',true задает, что счетчик считывает как пары пар.'Feature' - Тип функции'exon' (по умолчанию) | вектор символов | строкаТип функции, заданный как вектор символов или строка. Это используется, чтобы решить, какую функцию следует учитывать из файла GTF. По умолчанию это 'exon'.
'Metafeature' - Тип атрибута'gene_id' (по умолчанию) | вектор символов | строкаТип атрибута, заданный как вектор символов или строка. Это используется, чтобы решить, какой атрибут следует учитывать из файла GTF для группировки функций в метафункции и суммирования количества чтений.
'Summarization' - Логическая переменная, указывающая, суммировать ли ее на метафункцияtrue (по умолчанию) | falseЛогическая переменная, указывающая, нужно ли суммировать на уровне метафункции, заданная как true или false.
По умолчанию это true, что означает, что функция группирует функции в метафункции и сообщает счетчики чтения для метафункции.
'Alias' - Имя файла, содержащего псевдонимы ссылочных именИмя файла, содержащего псевдонимы имен ссылки, заданное как вектор символов или строка. Файл должен быть файлом с разделителем табуляций, где первый столбец соответствует именам ссылки, используемым в файле GTF, а второй столбец соответствует именам ссылки, используемым в вход файлов (файлов ) (ах). Имена учитывают регистр. Необходимо включать только имена ссылок, которые отличаются в файле GTF и в файле входа. Файл должен содержать только один псевдоним для любой ссылки, перечисленной во входном файле. По умолчанию имена ссылок в файле GTF и во входных файлах приняты одинаковыми.
'CountFragments' - Логическая переменная, указывающая, считать ли чтения как пары парfalse (по умолчанию) | trueЛогическая переменная, указывающая, считать ли чтения как фрагменты, заданная как true или false. Парные чтения должны иметь тот же идентификатор для поля QNAME в вход файле и взаимный порядок пар выводится соответствующим битом в FLAG поле в файле входа. Чтения, которые не имеют допустимого совмещения либо потому, что совмещение не сопоставлено, либо отфильтрованы по входным критериям, все еще подсчитываются, если они удовлетворяют перекрывающимся критериям.
По умолчанию это false, то есть считывания считываются как одноконцевые чтения, и их информация о сопряжении игнорируется.
'StrandSpecificity' - Специфичность цепочки протокола секвенирования'unstranded' (по умолчанию) | 'stranded' | 'reverse'Специфика цепи протокола секвенирования, заданная как 'unstranded' (по умолчанию), 'stranded', или 'reverse'.
Если 'unstranded', строка чтений (или фрагментов) игнорируется.
Если 'stranded', рассматривается прядь чтений (или фрагментов), и подсчитываются только те, которые имеют ту же прядь, что и функцию, который они перекрывают.
Если 'reverse', рассматривается противоположное направление цепи чтений (или фрагментов), и подсчитываются только те, которые имеют противоположную цепь в качестве функции, который они перекрывают.
При подсчете фрагментов (парно-конечные чтения) прядь первого помощника рассматривается как прядь всего фрагмента. Взаимный порядок пар (первый или второй) выводится из соответствующего бита в FLAG поле входного файла.
'MinOverlap' - Требуется минимальное количество перекрывающихся основ;1 (по умолчанию) | положительное целое числоМинимальное количество перекрывающихся основ, необходимых для назначения чтения функции, заданное в виде положительного целого числа. При подсчете фрагментов сумма перекрытий с каждого конца используется как минимальное количество перекрываемых основ.
'MinMappingQuality' - Минимальное качество отображения для заданного чтения0 (по умолчанию) | неотрицательное целое числоМинимальное качество отображения для данного чтения будет рассмотрено для подсчета, заданное как неотрицательное целое число. Это соответствует MAPQ поле в файле входа. Если подсчет фрагментов, по крайней мере, один из читаемых помощников должен удовлетворять этому критерию в порядок, чтобы учитываться для подсчета.
'CountMultiOverlap' - Логическая переменная, указывающая, считать ли чтения, перекрывающие несколько функцииfalse (по умолчанию) | trueЛогическая переменная, указывающая, считать ли чтения, перекрывающие несколько функции, заданная как true или false (по умолчанию).
Если trueчтение (или фрагмент), перекрывающий несколько функции, отсчитывается несколько раз. Во время суммирования на уровне метафункции чтение (или фрагмент) отсчитывается только один раз, если оно перекрывается с несколькими функциями, принадлежащими одной и той же метафункции, пока это не перекрывается с другими метеоресурсами.
'CountMultiMapped' - Опция подсчета для чтений, имеющих несколько местоположений отображения во входном файле'primary' (по умолчанию) | 'none' | 'all'Параметр подсчета для чтений, имеющих несколько местоположений сопоставления в файле входа, заданный как 'primary' (по умолчанию), 'none', или 'all'.
Если 'primary', учитывается только первичное выравнивание считывания с несколькими отображениями. Соответствующий бит в файле входа используется для идентификации первичных выравниваний.
Если 'none'Все выравнивания многосветного чтения игнорируются. Тег NH используется для идентификации показаний с несколькими отображениями.
Если 'all'Все выравнивания многомассового чтения рассматриваются и подсчитываются несколько раз.
'BothEndsMapped' - Логическая переменная, указывающая, должны ли быть сопоставлены оба фрагментаfalse (по умолчанию) | trueЛогическая переменная, указывающая, должны ли быть сопоставлены оба фрагмента, заданная как true или false. Информация о сопоставлении извлекается из FLAG поле в файле входа. По умолчанию это false.
'ProperlyPaired' - Логическая переменная, указывающая, должен ли фрагмент быть правильно спаренfalse (по умолчанию) | trueЛогическая переменная, указывающая, должен ли фрагмент быть правильно соединен в пару, заданная как true или false. Информация о сопряжении извлекается из FLAG поле в файле входа. По умолчанию это false.
'ShowZeroCounts' - Логическая переменная, указывающая, сообщать ли о функциях или метафункциях с нулевым количествомfalse (по умолчанию) | trueЛогическая переменная, указывающая, сообщать ли о функциях или метафункциях с нулевым количеством для каждого входного файла в выходной таблице, заданная как true или false.
По умолчанию это falseто есть в выходную таблицу включаются только строки с ненулевыми счетчиками и столбцы с ненулевыми счетчиками.
'OverlapMethod' - Метод, используемый при присвоении данного чтения метафункции'partial' (по умолчанию) | 'full' | 'max' | 'hits'Метод, используемый при назначении заданного чтения метафункции, заданный как 'partial', 'full', 'max', или 'hits'. Если 'Summarization' установлено в false, затем чтение назначается функциям, вместо метафункции, на основе заданного метода.
В следующей таблице R ссылается на чтение или фрагмент, а M - на метафункцию.
| Метод | Описание |
|---|---|
'partial' | R назначается, чтобы M, если R перекрывается (даже частично) только с M. В противном случае R рассматривается неоднозначным. |
'full' | R присвоено M, если R полностью отображена только внутри M, то есть полностью перекрывается только M. В противном случае R рассматривается как неоднозначное |
'max' | R назначается M, удовлетворяет ли R критериям перекрытия только M, или если R удовлетворяет критериям перекрытия с несколькими метафункциями, но полностью перекрывается только с M. |
'hits' | R назначается, чтобы M, если R перекрывается даже частично только M, или если M является единственной метафункцией с наибольшим количеством функций, пораженных R; в противном случае R рассматривается неоднозначным. |
Следующая принципиальная схема и таблица иллюстрируют результаты этих методов в сочетании с 'CountMultiOverlap' аргумент пары "имя-значение". На рисунке чтение относится к последовательности короткого чтения из файла входа, а функция A и функция B - к функциям, перечисленным в файле GTF.
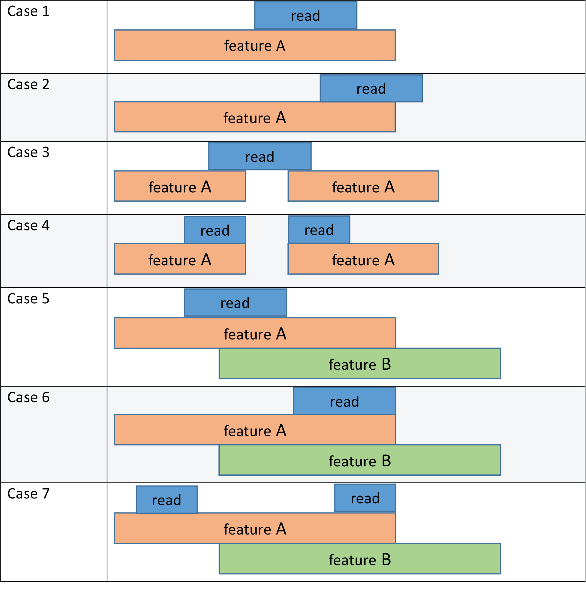
В каждом столбце метода перечислены функции, которым назначено чтение на основе соответствующего метода. The 'CountMultiOverlap' столбец указывает, установлена ли эта пара "имя-значение" true или false и если это имеет какой-либо эффект на результат каждого метода.
'CountMultiOverlap' | 'partial' | 'full' | 'max' | 'hits' | |
|---|---|---|---|---|---|
| Случай 1 | Эффект отсутствует, поскольку чтение преобразуется только в одну функцию (элемент A). | функция A | функция A | функция A | функция A |
| Дело 2 | Эффект отсутствует, поскольку чтение преобразуется только в одну функцию (элемент A). | функция A | нет функции | функция A | функция A |
| Дело 3 | Эффект отсутствует, поскольку чтение преобразуется только в одну функцию (элемент A). | функция A | нет функции | функция A | функция A |
| Дело 4 | Эффект отсутствует, поскольку чтение преобразуется только в одну функцию (элемент A). | функция A | функция A | функция A | функция A |
| Дело 5 | false | неоднозначный | функция A | функция A | неоднозначный |
true | функция A, функция B | функция A | функция A | функция A, функция B | |
| Дело 6 | false | неоднозначный | неоднозначный | неоднозначный | неоднозначный |
true | функция A, функция B | функция A, функция B | функция A, функция B | функция A, функция B | |
| Дело 7 | false | Неоднозначный | функция A | функция A | функция A |
true | функция A, функция B | функция A | функция A | функция A |
отсутствие функции означает, что чтение не назначено ни одному элементу. Если вы задали вторую выходную таблицу S, его Unassigned_noFeature строка для такого вхождения увеличивается на единицу. неоднозначное означает, что чтение не назначено никакой функции, поскольку оно удовлетворяет перекрывающимся критериям для нескольких функций и Unassigned_ambiguous строка для такого вхождения увеличивается на единицу.
'UseParallel' - Логическая переменная, указывающая, вычислять ли параллельноfalse (по умолчанию) | trueЛогическая переменная, указывающая, вычислять ли параллельно, задается как true или false.
В порядок выполнения расчета параллельно, вы должны иметь Toolbox™ Parallel Computing. Если MATLAB® параллельный пул не существует, он создается автоматически, когда опция автоматического создания включена в ваших параллельных настройках. В противном случае расчет выполняется в последовательном режиме.
По умолчанию это false, то есть последовательный режим.
'Verbose' - Логическая переменная, указывающая, отображать ли прогресс расчетовtrue (по умолчанию) | falseЛогическая переменная, указывающая, отображать ли прогресс расчетов, заданная как true или false.
T - Результаты, содержащие чтения последовательности, сопоставлены с геномными функциямиРезультаты, содержащие последовательности, считываются сопоставленными с геномными функциями, возвращаемыми как таблица. Строки соответствуют функциям, а столбцы соответствуют входным файлам. Элементы таблицы состоят из количества чтений, сопоставленных с каждой функцией для данного входного файла. В таблице также указывается идентификатор каждой функции и ссылочная последовательность для функции.
Когда 'Summarization' установлено значение false, столбец ID таблицы сообщает атрибут метафункции, за которым следуют начальное и стоповое положения каждой функции, разделенные символами подчеркивания.
S - Сводные данные назначенных и неназначенных записей выравниванияСводные данные назначенных и неназначенных записей выравнивания, возвращенная в виде таблицы. Каждый столбец таблицы соответствует каждому предоставленному входному файлу. Таблица имеет следующие строки:
| Имена строк | Описание |
|---|---|
TotalEntries | Количество записей (или выравнивания) в файле входа |
Assigned | Количество чтений или фрагментов, назначенных функциям |
Unassigned_ambiguous | Количество неприсоединенных чтений или фрагментов, перекрывающих несколько функции или метафункции |
Unassigned_filtered | Количество записей выравнивания, отфильтрованных по входным критериям |
Unassigned_lowMappingQuality | Количество записей выравнивания, отфильтрованных из-за низкого качества отображения |
Unassigned_multiMapped | Количество записей выравнивания, не назначенных из-за соответствующих чтений, сопоставленных с несколькими местоположениями |
Unassigned_noFeature | Количество чтений или фрагментов, не присвоенных каким-либо функциям |
Unassigned_supplementary | Количество записей выравнивания, не назначенных, поскольку они помечены как дополнительные записи для химерных выравниваний |
Unassigned_unmapped | Количество записей выравнивания, не назначенных, поскольку соответствующие чтения не сопоставлены |
Чтобы запустить параллельно, установите 'UseParallel' на true.
Для получения дополнительной информации смотрите 'UseParallel' аргумент пары "имя-значение".
У вас есть измененная версия этого примера. Вы хотите открыть этот пример с вашими правками?
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.