Отображение и манипуляция 3-D структурой молекулы
molviewer
molviewer(File)
molviewer(pdbID)
molviewer(pdbStruct)
FigureHandle =
molviewer(...)
File | Вектор символов или строка, задающая одно из следующего:
Файл-ссылка является файлом модели молекулы, таким как файл в формате Protein Data Bank (PDB) (текстовый файл ASCII). Допустимые типы файлов включают:
|
pdbID | Вектор символов или строка, задающая уникальный идентификатор для записи структуры белка в базе данных PDB. Примечание Каждая структура в базе данных PDB представлена четырехсимвольным алфавитно-цифровым идентификатором. Для примера, |
pdbStruct | Структура, содержащая поле для каждой записи PDB, например, возвращенная getpdb или pdbread функция. |
FigureHandle | Указатель на фигуру в Molecule Viewer. |
molviewer открывает приложение Molecule Viewer. Можно отобразить 3-D молекулярные структуры, выбрав File > Open, File > Load PDB ID или File > Open URL.
molviewer( считывает данные в файле модели молекулы, File)File, и открывает приложение Molecule Viewer, отображающее молекулярную структуру 3-D для просмотра и манипуляции.
molviewer( извлекает структурные данные белка, pdbID)pdbID, из базы данных PDB и открывает приложение Molecule Viewer, отображающее молекулярную структуру 3-D для просмотра и манипуляции.
molviewer( считывает данные из pdbStruct)pdbStruct, структуру, содержащую поле для каждой записи PDB, и открывает приложение Molecule Viewer, отображающее 3-D молекулярную структуру для просмотра и манипуляции.
FigureHandle =
molviewer(...)
Совет
Вы можете пройти FigureHandle в evalrasmolscript функция, которая отправляет команды скрипта RasMol в окно Molecule Viewer.
Совет
Если вы получаете какие-либо ошибки, связанные с памятью или Java® Пространство в куче, попробуйте увеличить пространство в Java, как описано на https://www.mathworks.com/support/solutions/en/data/1-18I2C/.
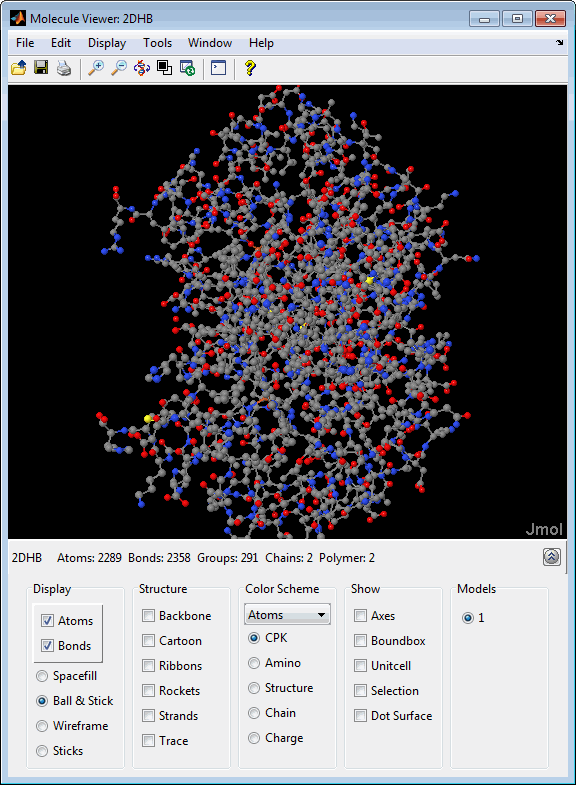
После отображения структуры молекулы 3-D, можно:
Наведите мышь на подкомпонент молекулы, чтобы отобразить идентификационную метку для нее.
Вращайте и вращайте молекулу под разными углами, перетаскивая ее щелчком мыши.
Прокрутите молекулу в плоскости x-z нажатием.![]()
Вращайте молекулу в плоскости x-y, нажимая и удерживая клавишу Shift, затем перетаскивая мышью влево и вправо.
Бесступенчатое масштабирование путем нажатия и удержания клавиши Shift, а затем перетаскивания мыши вверх и вниз.
Пошагово масштабировать, нажав на рисунок, затем повернув колесо прокрутки мыши или нажав следующие кнопки:
![]() или
или ![]()
Переместите молекулу, нажав и удерживая Ctrl + Alt, затем щелкните мышью - перетаскивание.
Измените цвет фона между черным и белым щелчком мыши.![]()
Сбросьте положение молекулы нажатием.![]()
Отобразить или скрыть панель управления можно нажав кнопку.![]()
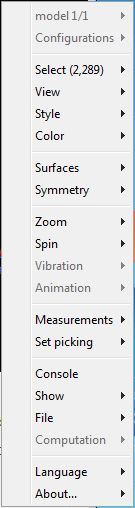
Управляйте и аннотируйте структуру 3-D, выбрав опции на панели управления или, для полного списка опций, щелкнув правой кнопкой мыши окно Molecule Viewer, чтобы выбрать команды:
Отобразить консоль Скрипт Console можно нажав кнопку.![]()
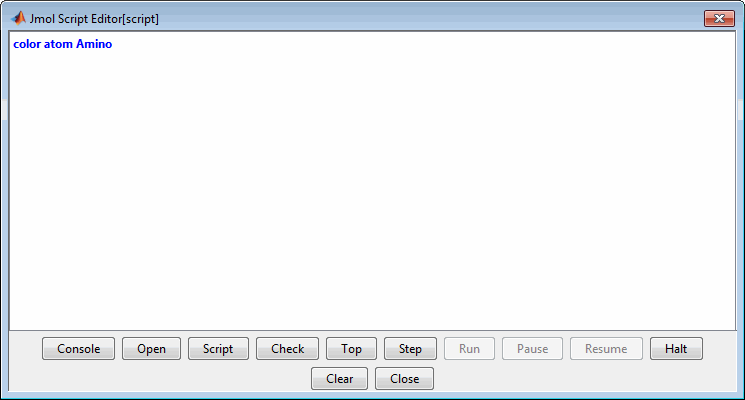
Примечание
Известна ошибка с кнопкой Open редактора скриптов, препятствующая интерактивной загрузке скрипта Rasmol. Вместо этого используйте evalrasmolscript функция, которая отправляет команды скрипта RasMol в Molecule Viewer приложение. Также можно скопировать и вставить команды скрипта в консоль скрипта.
Просмотрите молекулу ацетилсалициловой кислоты (аспирин), структурная информация которой содержится в файле молекулы Elsevier MDL aspirin.mol.
molviewer('aspirin.mol')Просмотрите молекулу гемагглютинина вируса H5N1 гриппа, структурная информация которой находится в www.rcsb.org/pdb/files/2FK0.pdb.gz.
molviewer('http://www.rcsb.org/pdb/files/2FK0.pdb.gz') Просмотрите молекулу с идентификатором PDB 2DHB.
molviewer('2DHB')Просмотрите молекулу с идентификатором PDB 4hhb, и создайте указатель на рисунок для Molecule Viewer.
FH = molviewer('4hhb')Используйте getpdb функция для извлечения данных структуры белка из базы данных PDB и создания структуры MATLAB. Затем посмотрим молекулу белка.
pdbstruct = getpdb('1vqx')
molviewer(pdbstruct) evalrasmolscript | getpdb | pdbread | pdbsuperpose | pdbtransform | pdbwrite