После секвенирования части ДНК одной из первых задач является исследование содержимого нуклеотидов в последовательности. Начиная с последовательности ДНК, этот пример использует функции статистики последовательностей для определения содержимого моно-, ди- и тринуклеотидов и для определения местоположения открытых систем координат считывания.
Следующая процедура иллюстрирует, как использовать MATLAB® Браузер документации для поиска информации в Интернете. В этом примере вы заинтересованы в изучении митохондриального генома человека. В то время как многие гены, которые кодируют белки митохондрий, находятся в ядре камеры, митохондриал имеет гены, которые кодируют белки, используемые для производства энергии.
Первая исследовательская информация о митохондриях человека и найти нуклеотидную последовательность для генома. Затем рассмотрим содержимое нуклеотидов для всей последовательности. И, наконец, определить открытые системы координат считывания и извлечь специфические последовательности генов.
Для исследования Интернета используйте браузер справки MATLAB. В Командном Окне MATLAB введите
web('http://www.ncbi.nlm.nih.gov/')
Откроется отдельное окно браузера с домашней страницей для веб-сайта NCBI.
Поиск информации на веб-сайте NCBI. Например, чтобы найти геном митохондриона человека, из списка Search выберите Genome , и в списке Search введите mitochondrion homo sapiens.
Веб-поиск NCBI возвращает список ссылок на соответствующие страницы.
Выберите страницу результатов. Например, щелкните ссылку, помеченную NC_012920.
Браузер MATLAB Help отображает страницу NCBI для митохондриального генома человека.
Следующая процедура иллюстрирует, как найти нуклеотидную последовательность в общедоступной базе данных и считать информацию о последовательности в окружение MATLAB. Многие общедоступные базы данных для нуклеотидных последовательностей доступны из Сети. Командное окно MATLAB обеспечивает интегрированное окружение для переноса информации о последовательности в окружение MATLAB.
Консенсусная последовательность для генома митохондрий человека имеет GenBank® номер присоединения NC_012920. Поскольку вся запись GenBank довольно велика, и вас может интересовать только последовательность, можно получить только информацию о последовательности.
Получите информацию о последовательности из веб-базы данных. Для примера, чтобы получить информацию о последовательности для митохондриального генома человека, в Командном окне MATLAB, введите
mitochondria = getgenbank('NC_012920','SequenceOnly',true)
The getgenbank функция извлекает нуклеотидную последовательность из базы данных GenBank и создает символьный массив.
mitochondria = GATCACAGGTCTATCACCCTATTAACCACTCACGGGAGCTCTCCATGCAT TTGGTATTTTCGTCTGGGGGGTGTGCACGCGATAGCATTGCGAGACGCTG GAGCCGGAGCACCCTATGTCGCAGTATCTGTCTTTGATTCCTGCCTCATT CTATTATTTATCGCACCTACGTTCAATATTACAGGCGAACATACCTACTA AAGT . . .
Если у вас нет веб-соединения, можно загрузить данные из файла MAT, включенного в программное обеспечение Bioinformatics Toolbox™, с помощью команды
load mitochondria
The load функция загружает последовательность mitochondria в Рабочее пространство MATLAB.
Получите информацию о последовательности. Напечатать
whos mitochondria
Информация о размере последовательности отображается в Командном Окне MATLAB.
Name Size Bytes Class Attributes mitochondria 1x16569 33138 char
Следующая процедура иллюстрирует, как определить мономеры и димеры, а затем визуализировать данные на графиках и столбиковых диаграммах. Срезы последовательности ДНК с высоким процентом A + T нуклеотидов обычно указывают на межгенные части последовательности, в то время как низкие A + T и более высокие G + C нуклеотидные проценты указывают на возможные гены. Во много раз высокое содержимое CG-динуклеотидов находится перед геном.
После чтения последовательности в окружение MATLAB можно использовать функции статистики последовательности, чтобы определить, имеет ли ваша последовательность характеристики белковой кодирующей области. Эта процедура использует человеческий митохондриальный геном в качестве примера. См. раздел «Чтение информации о последовательности из Интернета».
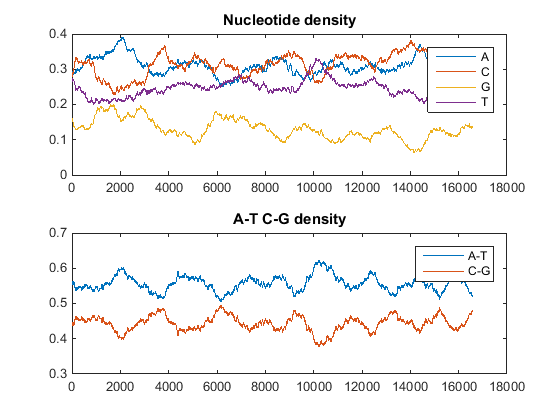
Постройте графики плотностей мономеров и комбинированных плотностей мономеров. В Командном Окне MATLAB введите
ntdensity(mitochondria)
Этот график показывает, что геном богат A + T.
Подсчитайте нуклеотиды, используя basecount функция.
basecount(mitochondria)
Список отсчётов нуклеотидов показан для цепи 5 '-3'.
ans =
A: 5124
C: 5181
G: 2169
T: 4094
Подсчитайте нуклеотиды в обратном дополнении последовательности, используя seqrcomplement функция.
basecount(seqrcomplement(mitochondria))
Как ожидалось, нуклеотидные отсчеты на обратной цепи комплемента комплементарны цепи 5 '-3'.
ans =
A: 4094
C: 2169
G: 5181
T: 5124
Используйте функцию basecount с chart опция визуализации нуклеотидного распределения.
figure basecount(mitochondria,'chart','pie');
Круговая диаграмма отображается в графическом окне MATLAB.
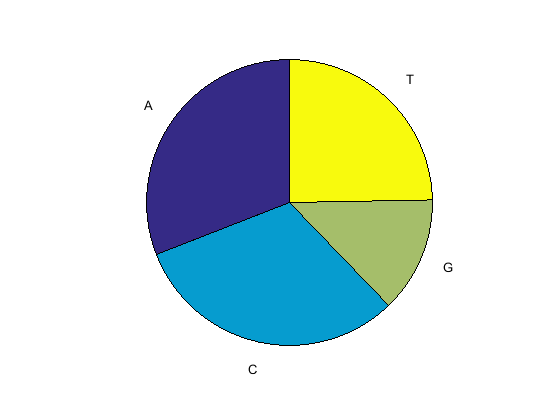
Подсчитайте димеры в последовательности и отобразите информацию в столбчатую диаграмму.
figure dimercount(mitochondria,'chart','bar')
ans =
AA: 1604
AC: 1495
AG: 795
AT: 1230
CA: 1534
CC: 1771
CG: 435
CT: 1440
GA: 613
GC: 711
GG: 425
GT: 419
TA: 1373
TC: 1204
TG: 513
TT: 1004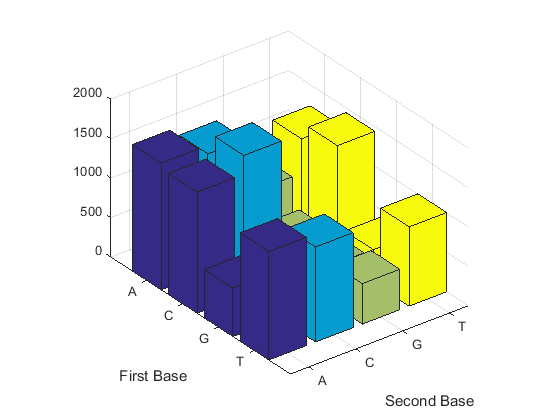
Следующая процедура иллюстрирует, как смотреть на кодоны для шести систем координат. Код тринуклеотидов (кодонов) для аминокислоты, и существует 64 возможных кодона в нуклеотидной последовательности. Знание процента кодонов в вашей последовательности может быть полезным, когда вы сравниваете с таблицами для ожидаемого использования кодонов.
После чтения последовательности в окружение MATLAB можно проанализировать последовательность на предмет состава кодона. Эта процедура использует геном митохондрий человека в качестве примера. См. раздел «Чтение информации о последовательности из Интернета».
Отсчитывайте кодоны в нуклеотидной последовательности. В Командном Окне MATLAB введите
codoncount(mitochondria)
Количество кодонов для отображения первой системы координат считывания.
AAA - 167 AAC - 171 AAG - 71 AAT - 130 ACA - 137 ACC - 191 ACG - 42 ACT - 153 AGA - 59 AGC - 87 AGG - 51 AGT - 54 ATA - 126 ATC - 131 ATG - 55 ATT - 113 CAA - 146 CAC - 145 CAG - 68 CAT - 148 CCA - 141 CCC - 205 CCG - 49 CCT - 173 CGA - 40 CGC - 54 CGG - 29 CGT - 27 CTA - 175 CTC - 142 CTG - 74 CTT - 101 GAA - 67 GAC - 53 GAG - 49 GAT - 35 GCA - 81 GCC - 101 GCG - 16 GCT - 59 GGA - 36 GGC - 47 GGG - 23 GGT - 28 GTA - 43 GTC - 26 GTG - 18 GTT - 41 TAA - 157 TAC - 118 TAG - 94 TAT - 107 TCA - 125 TCC - 116 TCG - 37 TCT - 103 TGA - 64 TGC - 40 TGG - 29 TGT - 26 TTA - 96 TTC - 107 TTG - 47 TTT - 78
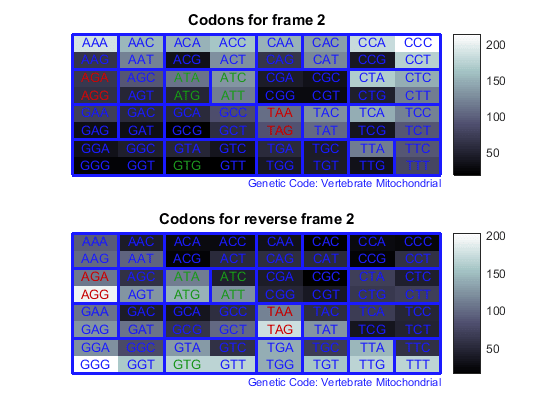
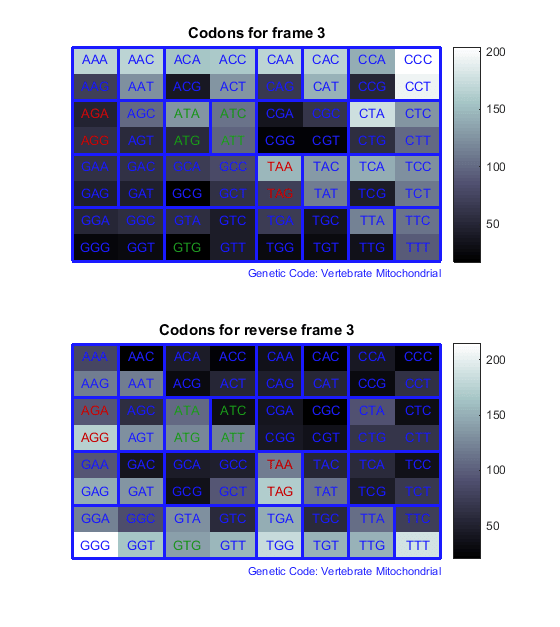
Подсчитайте кодоны во всех шести системах координат считывания и постройте график результатов в тепловых картах.
for frame = 1:3
figure
subplot(2,1,1);
codoncount(mitochondria,'frame',frame,'figure',true,...
'geneticcode','Vertebrate Mitochondrial');
title(sprintf('Codons for frame %d',frame));
subplot(2,1,2);
codoncount(mitochondria,'reverse',true,'frame',frame,...
'figure',true,'geneticcode','Vertebrate Mitochondrial');
title(sprintf('Codons for reverse frame %d',frame));
end
Тепловые карты отображают все 64 кодона в 6 системах координат считывания.

Следующая процедура иллюстрирует, как найти открытые системы координат чтения с помощью определенного генетического кода. Определение кодирующей белок последовательности для эукариотического гена может быть трудной задачей, поскольку интроны (некодирующие секции) смешиваются с экзонами. Однако прокариотические гены обычно не имеют интронов, и последовательности мРНК удаляют интроны. Идентификация стартового и стопового кодонов для трансляции определяет раздел кодирования белка или открытую систему координат чтения (ORF) в последовательности. Если вы знаете ORF для гена или мРНК, можно перевести нуклеотидную последовательность в соответствующую ей аминокислотную последовательность.
После чтения последовательности в окружение MATLAB можно проанализировать последовательность для открытых систем координат. Эта процедура использует геном митохондрий человека в качестве примера. См. раздел «Чтение информации о последовательности из Интернета».
Отображение открытые системы координат считывания (ORFs) в нуклеотидной последовательности. В Командном Окне MATLAB введите:
seqshoworfs(mitochondria);
Если вы сравниваете этот выход с генами, показанными на странице NCBI для NC_012920, генов меньше, чем ожидалось. Это происходит потому, что митохондрии позвоночных используют генетический код, немного отличный от стандартного генетического кода. Список генетических кодов см. в таблице Генетический код в aa2nt страница с описанием.
Отображение ORF-файлов с использованием Vertebrate Mitochondrial код.
orfs= seqshoworfs(mitochondria,...
'GeneticCode','Vertebrate Mitochondrial',...
'alternativestart',true);
Заметьте, что теперь на третьей системе координат чтения есть два больших ORF. Один начинается с положения 4470, а другой начинается с 5904. Они соответствуют генам ND2 (NADH-дегидрогеназа подмодуля 2 [Homo sapiens]) и COX1 (цитохром с оксидазой подмодуля I) генов.
Найдите соответствующий стоп-кодон. Начальное и стоповое положения для ORF имеют те же индексы, что и начальные положения в полях Start и Stop.
ND2Start = 4470; StartIndex = find(orfs(3).Start == ND2Start) ND2Stop = orfs(3).Stop(StartIndex)
Положение упора отображений.
ND2Stop =
5511Используя индексы последовательности для начала и остановки гена, извлеките подпоследовательность из последовательности.
ND2Seq = mitochondria(ND2Start:ND2Stop)
Подпоследовательность (область кодирования белка) сохранена в ND2Seq и отобразится на экране.
attaatcccctggcccaacccgtcatctactctaccatctttgcaggcac actcatcacagcgctaagctcgcactgattttttacctgagtaggcctag aaataaacatgctagcttttattccagttctaaccaaaaaaataaaccct cgttccacagaagctgccatcaagtatttcctcacgcaagcaaccgcatc cataatccttc . . .
Определите распределение кодонов.
codoncount (ND2Seq)
Количество кодонов показывает высокое количество ACC, ATA, CTA, и ATC.
AAA - 10 AAC - 14 AAG - 2 AAT - 6 ACA - 11 ACC - 24 ACG - 3 ACT - 5 AGA - 0 AGC - 4 AGG - 0 AGT - 1 ATA - 23 ATC - 24 ATG - 1 ATT - 8 CAA - 8 CAC - 3 CAG - 2 CAT - 1 CCA - 4 CCC - 12 CCG - 2 CCT - 5 CGA - 0 CGC - 3 CGG - 0 CGT - 1 CTA - 26 CTC - 18 CTG - 4 CTT - 7 GAA - 5 GAC - 0 GAG - 1 GAT - 0 GCA - 8 GCC - 7 GCG - 1 GCT - 4 GGA - 5 GGC - 7 GGG - 0 GGT - 1 GTA - 3 GTC - 2 GTG - 0 GTT - 3 TAA - 0 TAC - 8 TAG - 0 TAT - 2 TCA - 7 TCC - 11 TCG - 1 TCT - 4 TGA - 10 TGC - 0 TGG - 1 TGT - 0 TTA - 8 TTC - 7 TTG - 1 TTT - 8
Посмотрите аминокислоты на кодоны ATA, CTA, ACC, и ATC.
aminolookup('code',nt2aa('ATA'))
aminolookup('code',nt2aa('CTA'))
aminolookup('code',nt2aa('ACC'))
aminolookup('code',nt2aa('ATC'))
Следующие отображения:
Ile isoleucine Leu leucine Thr threonine Ile isoleucine
Следующая процедура иллюстрирует, как экстрагировать кодирующую белок последовательность из генной последовательности и преобразовать ее в аминокислотную последовательность для белка. Определение относительного аминокислотного состава белка даст вам характерный профиль для белка. Часто этот профиль является достаточной информацией для идентификации белка. Используя аминокислотную композицию, атомарную композицию и молекулярную массу, можно также искать в общедоступных базах данных подобные белки.
После определения местоположения открытой системы координат считывания (ORF) в гене можно преобразовать ее в аминокислотную последовательность и определить ее аминокислотный состав. Эта процедура использует геном митохондрий человека в качестве примера. См. «Открытые системы координат чтения».
Преобразуйте нуклеотидную последовательность в аминокислотную. В этом примере преобразуется только последовательность, кодирующая белок между начальным и стоповым кодонами.
ND2AASeq = nt2aa(ND2Seq,'geneticcode',...
'Vertebrate Mitochondrial')
Последовательность преобразуется с помощью Vertebrate Mitochondrial генетический код. Потому что свойство AlternativeStartCodons установлено в 'true' по умолчанию первый кодон att преобразуется в M вместо I.
MNPLAQPVIYSTIFAGTLITALSSHWFFTWVGLEMNMLAFIPVLTKKMNP RSTEAAIKYFLTQATASMILLMAILFNNMLSGQWTMTNTTNQYSSLMIMM AMAMKLGMAPFHFWVPEVTQGTPLTSGLLLLTWQKLAPISIMYQISPSLN VSLLLTLSILSIMAGSWGGLNQTQLRKILAYSSITHMGWMMAVLPYNPNM TILNLTIYIILTTTAFLLLNLNSSTTTLLLSRTWNKLTWLTPLIPSTLLS LGGLPPLTGFLPKWAIIEEFTKNNSLIIPTIMATITLLNLYFYLRLIYST SITLLPMSNNVKMKWQFEHTKPTPFLPTLIALTTLLLPISPFMLMIL
Сравните преобразование с опубликованным преобразованием в базе данных GenPept.
ND2protein = getgenpept('YP_003024027','sequenceonly',true)
The getgenpept функция извлекает опубликованное преобразование из базы данных NCBI и считывает его в рабочее пространство MATLAB.
Подсчитайте аминокислоты в белковой последовательности.
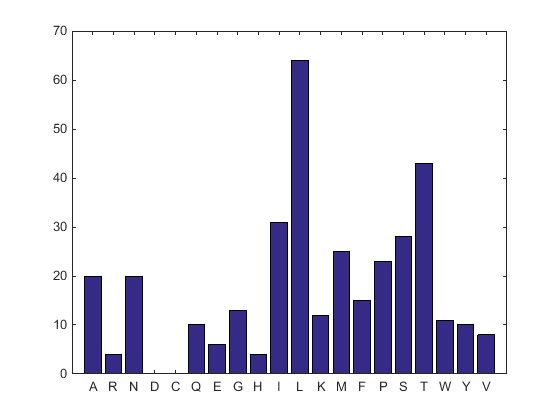
aacount(ND2AASeq, 'chart','bar')
Отображается штриховой график. Заметьте высокое содержимое лейцина, треонина и изолейцина, а также заметьте отсутствие цистеина и аспарагиновой кислоты.
Определите атомарный состав и молекулярную массу белка.
atomiccomp(ND2AASeq) molweight (ND2AASeq)
В рабочем пространстве MATLAB отображается следующее:
ans =
C: 1818
H: 2882
N: 420
O: 471
S: 25ans = 3.8960e+004
Если бы эта последовательность была неизвестна, можно было бы использовать эту информацию для идентификации белка путем сравнения его с атомарным составом других белков в базе данных.